Занятие 1
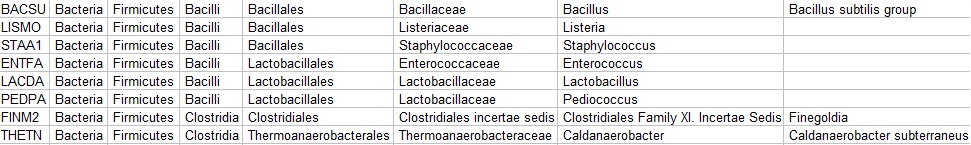
Отобранные бактерии
| Название | Мнемоника |
| Bacillus subtilis | BACSU |
| Enterococcus faecalis | ENTFA |
| Finegoldia magna | FINM2 |
| Lactobacillus delbrueckii | LACDA |
| Listeria monocytogenes | LISMO |
| Pediococcus pentosaceus | PEDPA |
| Staphylococcus aureus | STAA1 |
| Thermoanaerobacter tengcongensis | THETN |
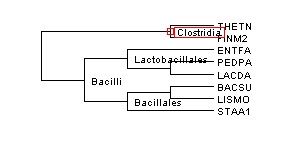
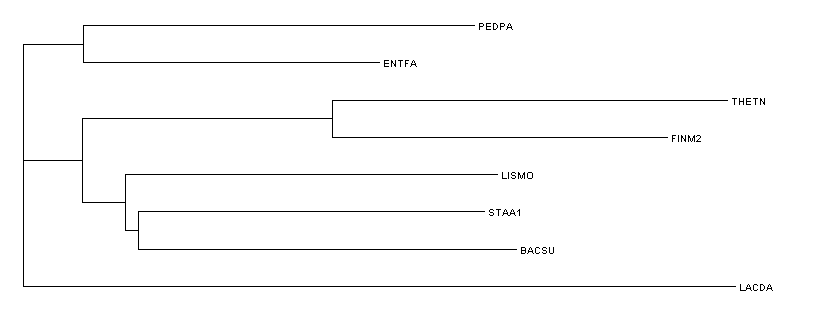
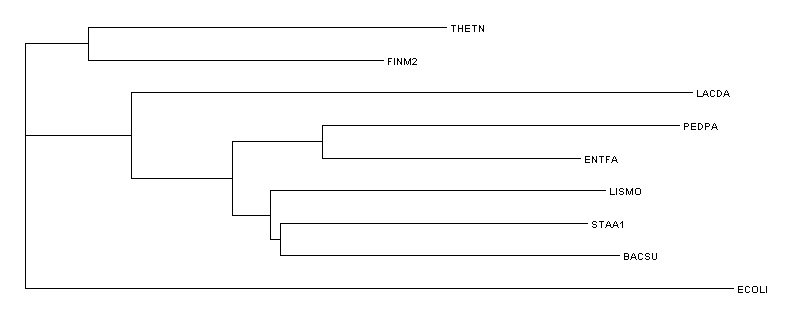
Скобочная формула дерева
((THETN,FINM2),((ENTFA,(PEDPA,LACDA)),(STAA1,(BACSU,LISMO))));
Изображение дерева
Ветви дерева
Дерево содержит 5 нетривиальных ветвей:
1) {THETN,FINM2} {PEDPA,LACDA,ENTFA,BACSU,LISMO,STAA1} 2) {PEDPA,LACDA} {ENTFA,BACSU,LISMO,STAA1,THETN,FINM2} 3) {PEDPA,LACDA,ENTFA} {BACSU,LISMO,STAA1,THETN,FINM2} 4) {BACSU,LISMO} {STAA1,THETN,FINM2,PEDPA,LACDA,ENTFA} 5) {BACSU,LISMO,STAA1} {THETN,FINM2,PEDPA,LACDA,ENTFA} В этом филогенетическом дереве есть ветви, соответствующие отдельным таксонам:
Реконструкция дерева по белкам семейства EFTS.
1.fprotpars
Программа выдала одно дерево, отличающееся от филогенетического: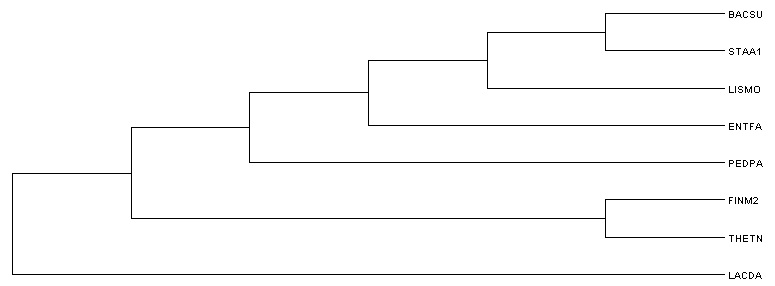
((((((BACSU,STAA1),LISMO),ENTFA),PEDPA),(FINM2,THETN)),LACDA);
В полученном дереве отсутствуют нетривиальные ветви:
2) {PEDPA,LACDA} {ENTFA,BACSU,LISMO,STAA1,THETN,FINM2}
3) {PEDPA,LACDA,ENTFA} {BACSU,LISMO,STAA1,THETN,FINM2}
4) {BACSU,LISMO} {STAA1,THETN,FINM2,PEDPA,LACDA,ENTFA}
А ветвей
{THETN,FINM2,LACDA} {PEDPA,ENTFA,BACSU,LISMO,STAA1}
{THETN,FINM2,LACDA,PEDPA} {ENTFA,BACSU,LISMO,STAA1}
{BACSU,STAA1} {LISMO,THETN,FINM2,PEDPA,LACDA,ENTFA}
нет в исходном дереве.
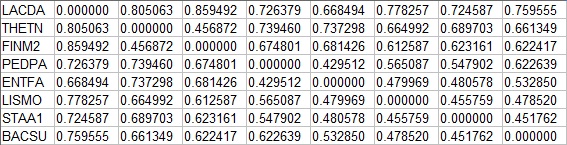
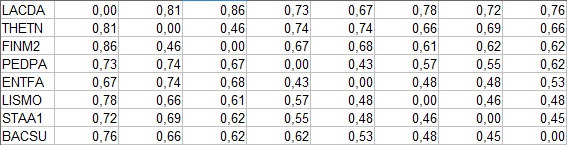
2.fprotdist
Программой была получена матрица:
Для удобства преобразуем:
Проверим таблицу на ультраметричность и аддитивность: Для выполнения принципа ультраметричности необходимо, чтобы для любых трех листьев расстояния между двумя были равны и не меньше третьего. Для примера возьмем LACDA, FINM2 и THETN : 0,81; 0,86 и 0,46 - принцип выполняется с трудом, отклонение от равенства 0,05. Для LISMO, STAA1 и BACSU: 0,46; 0,48 и 0,45 - принцип не выполняется (двое равны и меньше третьего), отклонение от равенства 0,01. Для выполнения принципа аддитивности необходимо, чтобы для любых четырех листьев две из сумм расстояний между парами листьев были равны и больше третьей. На примере LACDA, THETN, FINM2 и PEDPA: 1,48; 1,60 и 1,13 - принцип не выполняется. При помощи команды fneighbo были получены следующие деревья: NJ
((PEDPA:0.24454,ENTFA:0.18497):0.03747,((THETN:0.24710,FINM2:0.20977):0.15647, (LISMO:0.23257,(STAA1:0.21585,BACSU:0.23591):0.00869):0.02689):0.03664,LACDA:0.44521); UPGMA
(LACDA:0.38013,((THETN:0.22844,FINM2:0.22844):0.10692,((PEDPA:0.21476, ENTFA:0.21476):0.05433,(LISMO:0.23357,(STAA1:0.22588,BACSU:0.22588):0.00769):0.03552):0.06627):0.04477); Почему-то все программы иначе восстановили ветви {BACSU,LISMO,STAA1}, а LACDA по этому белку, похоже, достаточно сильно отличается от остальных. Ни одно дерево не является в точности филогенетическим, ближе всего к нему дерево, построенное алгоритмом Neighbor-Joining. (Укорененние в {THETN,FINM2}.) Возможно, эволюция этих белков шла несколько рассогласованно с эволюцией самих организмов.
Укоренение в среднюю точку
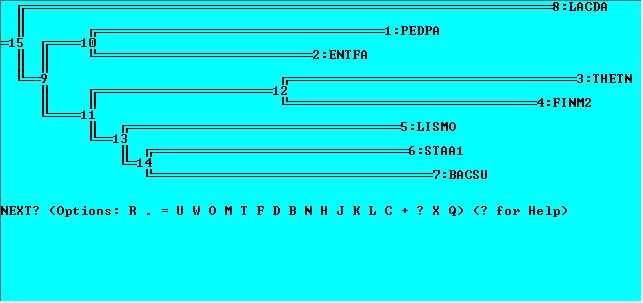
Укоренение произошло в тривиальную ветвь {Lacda}, что совершенно не соответствует истинному корню. Метод укоренения в среднюю точку нельзя использовать для деревьев, построенных MP UPGMA, т.к. первый не выдаёт длины ветвей, а второй укореняет дерево.
Использование внешней группы
Корень найден верно. Укоренение в {THETN,FINM2} {PEDPA,LACDA,ENTFA,BACSU,LISMO,STAA1}.
Бутстрэп
+---------------LACDA
+--56.0-|
| | +-------FINM2
+--43.5-| +--100.0-|
| | +-------THETN
| |
+-------| +-----------------------PEDPA
| |
| | +---------------LISMO
| +----------79.7-|
| | +-------BACSU
| +--61.5-|
| +-------STAA1
|
+---------------------------------------ENTFA
Как и следовало ожидать, дерево оказалось недостоверным. Из пяти нетривиальных ветвей достоверна на >80%
только одна: {THETN,FINM2} {PEDPA,LACDA,ENTFA,BACSU,LISMO,STAA1}, она соответствует истинному дереву.
Вторая по достоверности ветвь - {BACSU,LISMO,STAA1} {THETN,FINM2,PEDPA,LACDA,ENTFA},- тоже соответствует
филогенетическому дереву видов, но уже третья - {BACSU,STAA1} {LISMO,THETN,FINM2,PEDPA,LACDA,ENTFA},
достоверность которой оценена в 61,5% восстановлена неверно.
Построение дерева по нуклеотидным последовательностям.
Последовательности 16S рибосомальной РНК каждой из бактерий были выделены из файлов EMBL-банка
(полученных при помощи FTP-клиента), визуализированных программой Genom Explorer. Там находилась
нужная РНК и экспортировалась. Далее выровненные последовательности подавались на вход программе
fdnadist, и затем полученная матрица отправлялась fneighbor. Результат:
Neighbor-joining method
Negative branch lengths allowed
+------FINM2
+-1
! +-----THETN
!
! +---LACDA
! +-2
! ! +---PEDPA
3-4
! ! +-STAA1
! +-5
! ! +--LISMO
! +-6
! +-BACSU
!
+------------------------------------------------ENTFA
remember: this is an unrooted tree!
Полученное дерево является "соседним" к правильному. Если оторвать тривиальную ветвь ENTFA и "привить"
на ветвь {LACDA,PEDPA}, получится правильное дерево. Таким образом дерево, построенное по рибосомальной
РНК оказалось ближе всего к филогенетическому.
Построение и анализ дерева, содержащего паралоги.
Были выбраны следующие ортологичные последовательности:
score p-value
CLPX_BACSU 770 0.0
CLPX_LISMO 632 0.0
CLPX_THETN 572 e-164
CLPX_ENTFA 545 e-156
HSLU_ENTFA 100 6,00E-22
CLPX_PEDPA 533 e-152
B0S2N5_FINM2 520 e-148
B0S0E3_FINM2 50 7,00E-07
CLPX_STAA1 564 e-161
HSLU_STAA1 93 1,00E-19
HSLU_LACDA 97 6,00E-21
Дерево построено программой fprotpars.
+--CLPX_LISMO
+--5
+--------------4 +--CLPX_BACSU
! !
! +-----CLPX_STAA1
!
+--3 +--HSLU_ENTFA
! ! +--8
! ! +-----7 +--HSLU_LACDA
! ! ! !
! +--------6 +-----HSLU_STAA1
1 !
! ! +--CLPX_PEDPA
! +--------2
! +--CLPX_ENTFA
!
+-----------------------CLPX_THETN
Паралоги: CLPX_STAA1 и HSLU_STAA1; HSLU_ENTFA и CLPX_ENTFA.
Ортологи: CLPX_ENTFA и CLPX_THETN; CLPX_LISMO и CLPX_BACSU; CLPX_BACSU и HSLU_LACDA.