В этом практикуме я использовал проект по секвенированию бактерии Buchnera aphidicola str. Tuc7 SRR4240380.
Чтения были обработаны программой trimmomatic, сначала для того, чтобы избавиться от адаптеров:
TrimmomaticSE -phred33 SRR4240380.fastq.gz output.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7
Остатками адаптеров оказались 97710 последовательностей (1.87% от исходного числа - 5217318).
Затем с правых концов были удалены нуклеотиды, качество прочтения концов которых ниже 20, и оставлены тголько последовательности длиной от 32 нуклеотидов:
TrimmomaticSE -phred33 SRR4240380.fastq.gz output.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7 TRAILING:20 MINLEN:32
Было удалено еще 250073 последовательности (4.88% от остававшихся). Файл output.fastq.gz до обработки имел размер 110861610 байт, а после - 103215899 байт.
Далее с помошью программы velveth были подготовлены k-меры длины 31, на их основе была составлена сборка программой velvetg:
velveth . 31 -fastq.gz output.fastq.gz -short
velvetg .
Таблица с длинами и покрытиями получившихся контигов
N50 = 12550 (длина контига 36).
Самые длинные контиги - 3 (длина 25945, покрытие 27.45), 20 (длина 23880, покрытие 24.81) и 22(длина 23837, покрытие 25.78). Медианное покрытие - 23.69649, есть 2 контига, покрытие которых более чем в 5 раз превышает медианное: 55 (длина 964, покрытие 134.67) и 11 (длина 2136, покрытие 127.8). Также есть 5 контигов, покрытие которых более чем в 5 раз уступает медианному: 237 (длина 124, покрытие 4.45), 143 (длина 96, покрытие 4.43), 298 (длина 93, покрытие 4.26), 107 (длина 110, покрытие 3.71) и 231 (длина 92, покрытие 3.59).
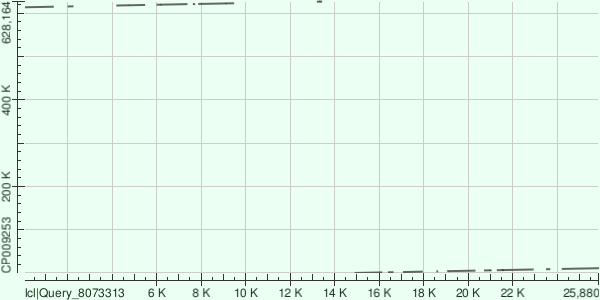
Результаты выравниваний megablast (длина слова 24) с хромосомой Buchnera aphidicola (CP009253) для контигов 3, 20 и 22:

Из графика и таблицы видно, что контигу 3 соответствуют участки хромосомы 133-11060 и 613671-626880. Разрыв в данном случае объясняется тем, что хромосома кольцевая. Megablast выдал 16 выравниваний, во всех 16 последовательности совпадают на 78-98%, e-value от 0 до 5.72e-08; всего в них 2437 SNP и 16 инделей.
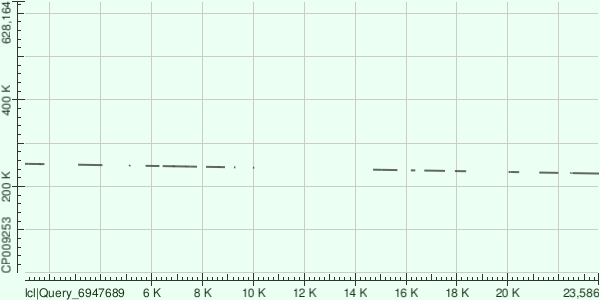
Контигу 20 соответствует участок 229650-252161, причем контиг принадлежит обратной цепи. Выравниваний здесь также 16, последовательности совпадают на 77-100%, e-value от 0 до 5.08e-05; 1670 SNP, 15 инделей.
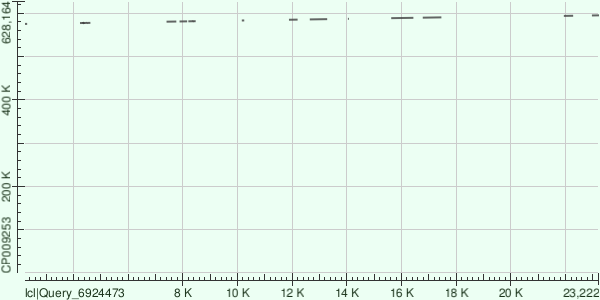
Контигу 22 соответствует участок 575219-595025. Выравниваний 13, последовательности совпадают на 80-97%, e-value от 3.16e-114 до 5.46e-11; 902 SNP, 8 инделей.