Обзор генома бактерии Phenylobacterium zucineum
Аннотация
В работе проводится анализ генома бактерии Phenylobacterium zucineum. Это факультативная внутриклеточная бактерия, которая не убивает клетку-хозяина
Введение
Phenylobacterium zucineum - внутриклеточная бактерия, выделенная из клеточной линии лейкемии человека K562. Это палочковидная грамотрицательная бактерия [1]. Род Phenylobacterium, помимо данной бактерии, включает в себя ещё четыре вида: P. lituiforme, P. falsum, P. immobile, P. koreense. P. Zucineum является факультативной внутриклеточной бактерией, тогда как остальные из рода были выделены из подземных водоносных горизонтов, щелочных грунтовых вод, почвы и активного ила очистных сооружений соответственно. P. zucineum – единственный вид из рода Phenylobacterium, который может проникать и жить в человеческих клетках. Предварительные данные исследований говорят, что P. zucineum может заражать людей. Интересно, что она сохраняет стабильную связь с клеткой-хозяином, не влияя на ее рост и морфологию, т.е. P. zucineum инфицирует клетки человека и паразитирует в них, но не убивает [1].
Таксономическое положение изучаемой бактерии P. zucineum [2]:
В работе мы провели частичный анализ генома бактерии: выяснили распределение длин белков, длины пересечений нуклеотидных последовательностей на обеих цепях хромосомы и изучили нуклеотидный состав генома.
Материлы и методы
- Геном бактерии взят с сайта NCBI.
- Гистограмма длин белков составлена методом электронных таблиц, сервис Google sheets. Были отобраны данные про белки, а после составлена гистограмма по данным столбца с длиной белков. Карман (промежуток, длина которого измерена в нуклеотидах) выбран 50. Белки, длина которых превышала 1300 нуклеотидов, объединены в один столбец (см. сопроводительные материалы S1).
- Длина пересечений на цепях хромосомы определена методом электронных таблиц, сервис Google sheets. Также были отобраны данные про белки. Далее, по столбцу, который обозначает + и - цепи, отобрана одна из цепей. Для неё были найдены участки, на которых последовательность одного белка заканчивалась позже, чем начиналась последовательность другого белка. По этим данным построена гистограмма. Аналогично для другой цепи (см. сопроводительные материалы S2).
- Частотный анализ триплетов составлен при помощи Python. Поиск проводился по файлу с кодирующими последовательностями. Был составлен словарь со всеми триплетами. Далее я посчитала каждое вхождение триплета в белковую последовательность. Гистограмма была составлена с помощью библиотеки Python Matplotlib (см. сопроводительные материалы S3).
- Анализ частоты встречаемости пар нуклеотидов составлен при помощи Python. Поиск проводился по файлу с кодирующими последовательностями. Я составила словарь с парами нуклеотидов и посчитала каждое вхождение пары в белковую последовательность и по результатам составила гистограмму (см. сопроводительные материалы S4).
Результаты
Длины белков, закодированных в геноме бактерии
По гистограмме длин белков, закодированных в геноме бактерии Phenylobacterium zucineum (рис. 1) видно, что наиболее распространены белки длиной 50-350 аминокислот. Самая распространённая длина белков – 100-300 аминокислот. И есть всего 11 белков, которые превышают длину 1300 аминокислот.
Длина пересечений участков, кодирующих белки, на разных цепях хромосомы
На гистограммах пересекающихся участков, в которых закодированы белки бактерии Phenylobacterium zucineum (рис. 2, 3) мы видим, что в подавляющем большинстве случаев пересечения отсутствуют – 1630 и 1617 (рис. 2 и рис. 3 соответственно). Однако длина пересечения 4 тоже достаточно распространена – 253 и 197 (рис. 2 и рис. 3 соответственно). Следующие по распространенности участки: 1 и 8 (длина в нуклеотидах, рис. 2 и рис. 3 соответственно). При этом, если длина пересечения 1, то между белками отсутствуют нуклеотиды, не участвующие в кодировании, т.е. между старт-кодоном нуклеотидной последовательности одного белка и стоп-кодоном предыдущей нет нуклеотидов, не участвующих в кодировании. Все остальные участки распространены минимально.
Количественный анализ триплетов
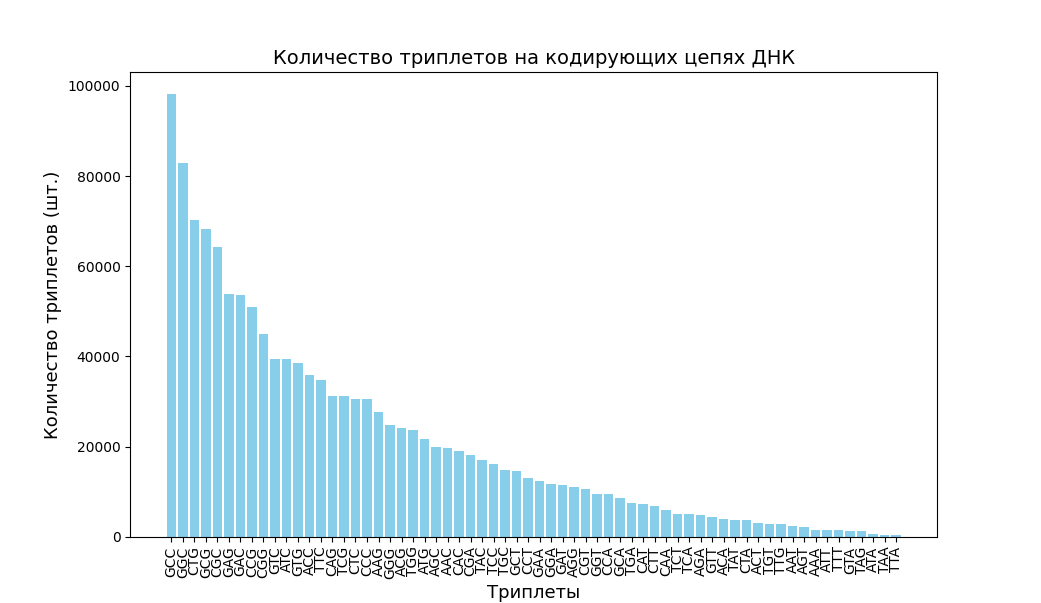
Рис. 4 - Частота встречаемости триплетов на кодирующей последовательности ДНК (CDS)
Наиболее распространённый триплет: GCC (115557 раз)
Наименее распространённый триплет: TTA (101 раз)
Триплет GCC кодирует аминокислоту аланин (Ala). Возможно, такой высокий уровень встречаемости этого кодона связан с особенностями работы таких внутренних структур бактерии, как рибосом. Также вероятно, что tRNA, которая переносит данную аминокислоту, является наиболее доступной.
Триплет TTA кодирует аминокислоту лейцин (Leu). Кроме него, её кодируют есть ещё пять кодонов: TTG, CTT, CTC, CTA, CTG. Сам триплет TTA встречается наиболее редко, однако его компенсируют триплеты CTG и CTC, которые распространены значительно больше и позволяют поддерживать высокий процент лейцина в белках.
Интересно, что чаще всего встречаются триплеты, в составе которых есть гуанин (G) и цитозин (C).
Анализ частоты встречаемости пар нуклеотидов
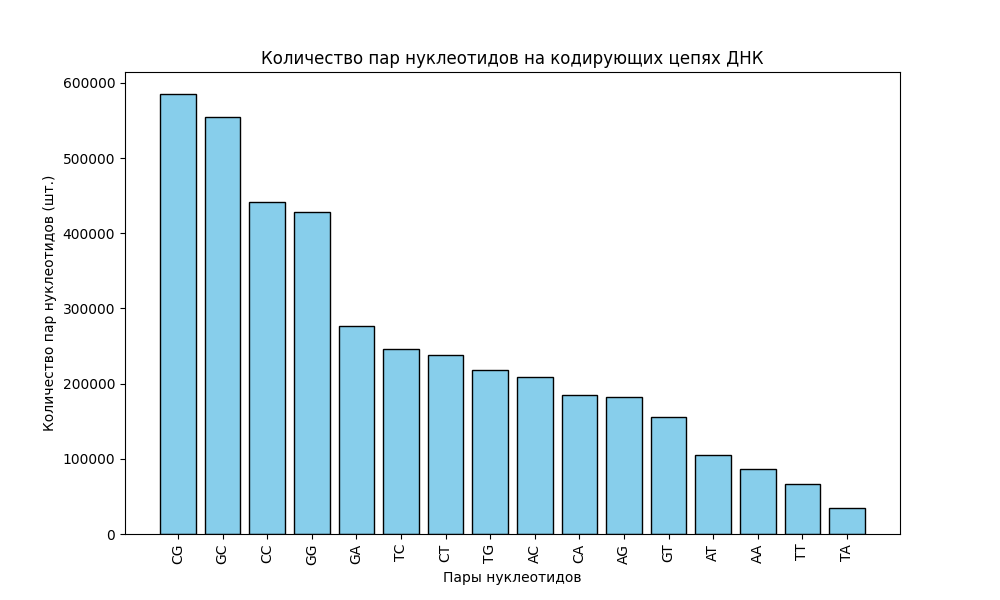
Рис. 5 - Частота встречаемости пар нуклеотидов в кодирующей последовательности ДНК (CDS)
Как видно по гистограмме, наиболее распространены пары нуклеотидов CG и GC, а также CC и GG. Я предположила по результатам анализа триплетов (см. п. 4.3), что пары, состоящие из гуанина и цитозина, или только из гуанина, или только из цитозина, будут чаще встречаться в геноме, чем другие. Проведя краткий анализ, я выяснила, что это действительно так. Также, области с высоким содержанием CG-динуклеотидов могут быть важными регуляторными элементами транскрипции [3].
Выводы
По результатам анализа триплетов я предположила, что пары, состоящие из гуанина и цитозина, или только из гуанина, или только из цитозина, будут чаще встречаться в геноме, чем другие. Проведя краткий анализ, выяснилось, что это действительно так.
Кроме того, были получены данные о частоте перекрываний последовательностей на цепях ДНК, длине белков, распределении триплетов и частоте встречаемости пар нуклеотидов.
Сопроводительные материалы
- S1
- S2: Пересечения плюс-цепи хромосомы. Пересечения минус-цепи хромосомы
- S3, S4
Литература
- Yingfeng Luo, Xiaoli Xu, Zonghui Ding, Zhen Liu, Bing Zhang, Zhiyu Yan, Jie Sun, Songnian Hu & Xun Hu, Complete genome of Phenylobacterium zucineum – a novel facultative intracellular bacterium isolated from human erythroleukemia cell line K562 / - Yingfeng Luo, Xiaoli Xu, Zonghui Ding, Zhen Liu, Bing Zhang, Zhiyu Yan, Jie Sun, Songnian Hu & Xun Hu // BMC Genomics : электронный журнал. – URL: https://bmcgenomics.biomedcentral.com/articles/10.1186/1471-2164-9-386. – Дата публикации: 13.08.2008.
- GTDB – Genome Taxonomy Database [Электронный ресурс]. – Режим доступа: https://gtdb.ecogenomic.org/species?id=Phenylobacterium%20zucineum, свободный – (03.12.2024)
- Wikipedia – [Электронный ресурс]. – Режим доступа: https://ru.wikipedia.org/wiki/GC-состав, свободный - 13.12.2024

