Изначально я выбрала домашнюю лошадь (Equus caballus), но у меня всё легло на компе из-за её генома, поэтому пришлось взять другой организм, Naegleria fowleri. У неё нужный белок не нашла, я клянусь, может я слепая, но я не нашла. Поэтому в итоге выбрала Arabidopsis thaliana.
Сборка RefSeq: GCF_000001735.4
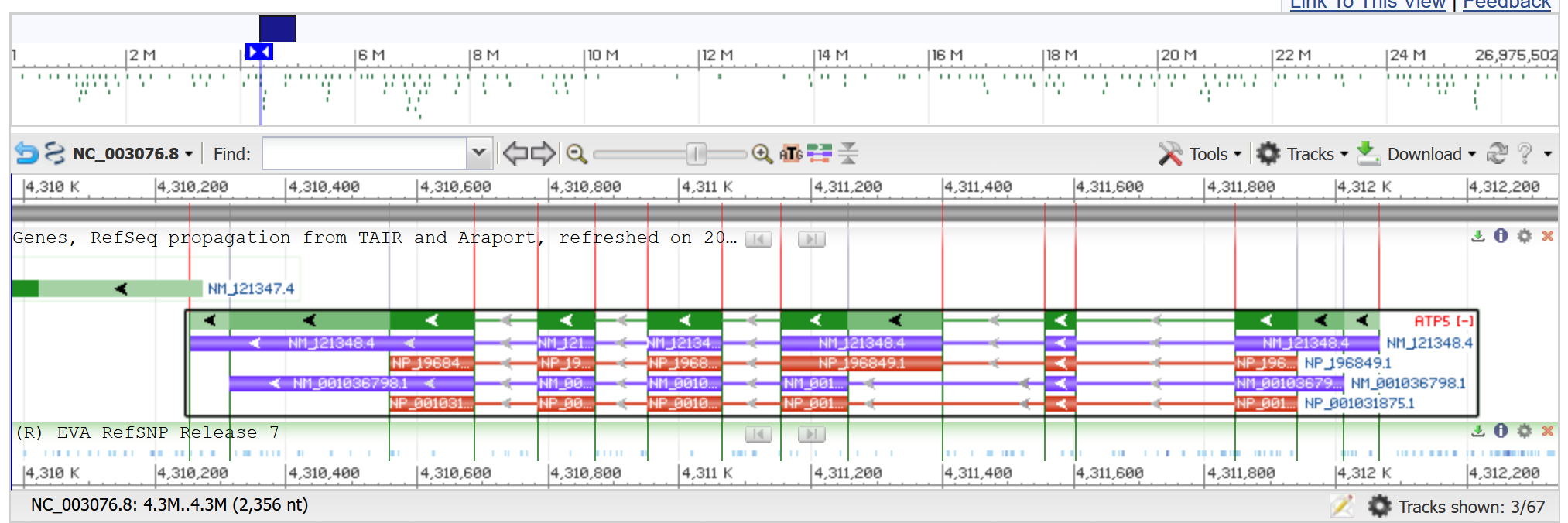
В задании требуется найти в геноме выбранного эукариота ген, кодирующий δ-субъединицу АТФ-синтазы
Последовательность белка, аннотированная как нужный ген была найдена с помощью текстового поиска. Её идентификатор: AT5G13450
Идентификатор нуклеотидной записи, к которой относится ген: NP_196849.1
FASTA-файл нуклеотидной последовательностью

Так как мой организм является простейшим, можно было выбрать любой из представленных в задании таксонов. Выбранный таксон: Пчёлы (Apoidea)
Использованная база данных: RefSeq Genomes
Число сборок, входящих в таксон: 38
Длина слова: 3
Остальные параметры по умолчанию

Выдача странная, так как Query Cover - 76-77% для лучших находок, а наши организмы неродственные. Возможно, совпавшие участки - интроны или консервативные домены
Использованная база данных: RefSeq Genomes
Число сборок, входящих в таксон: 38
Длина слова: 11
Остальные параметры по умолчанию
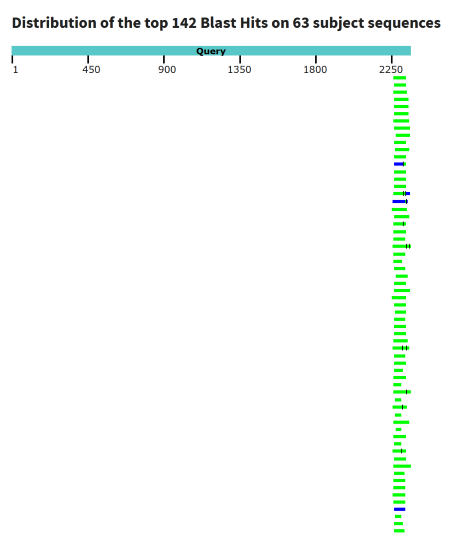
Процент покрытия маленький (3-4%). Мне кажется странным, что анализ через blastn показал очень маленькие участки совпавших результатов, хотя именно blastn предназначен для выявления нуклеотидных гомологов (генов с консервативными участками ДНК
На локальном компьютере с помощью BLAST+ была проиндексирована последовательность моего организма для последующей работы локального BLAST. Используемая команда:
makeblastdb -in GCF_000001735.4_TAIR10.1_genomic.fna -dbtype nucl -out anya.fna
-dbtype - указываем тип последовательности
После этого мы скачали последовательности рРНК (16S и 23S) Escherichia coli и провели локальный поиск BLAST по полученной базе данных по методу blastn
Команда для выполнения (16S): blastn -task blastn -query ./16s.fa -db ./anya.fna -outfmt 7 -evalue 0.005 -out 16sblast_anya
Алгоритм выдал 12 хитов
Все выравнивания имеют:
1) длина выравнивания либо 1545 нт, либо короткие высококонсервативные выравнивания длиной 47-97 нт
2) идентичность ≈ 73-96%
3) все хиты найдены в хлоропласте (NC_000932.1)
4) E-value ≈ 7.99e-10
Команда для выполнения (23S): blastn -task blastn -query ./23s.fa -db ./anya.fna -outfmt 7 -evalue 0.005 -out 23sblast_anya
Алгоритм выдал 25 хитов
Все выравнивания имеют:
1) длина выравнивания либо 1545 нт, либо короткие высококонсервативные выравнивания длиной ≈ 150-300 нт, либо очень короткие участки 38-67 нт
2) идентичность ≈ 70-92%
3) все хиты найдены в хлоропласте (NC_000932.1)
4) E-value от 0.0 до 0.004, но в целом ≈ 9.59e-11
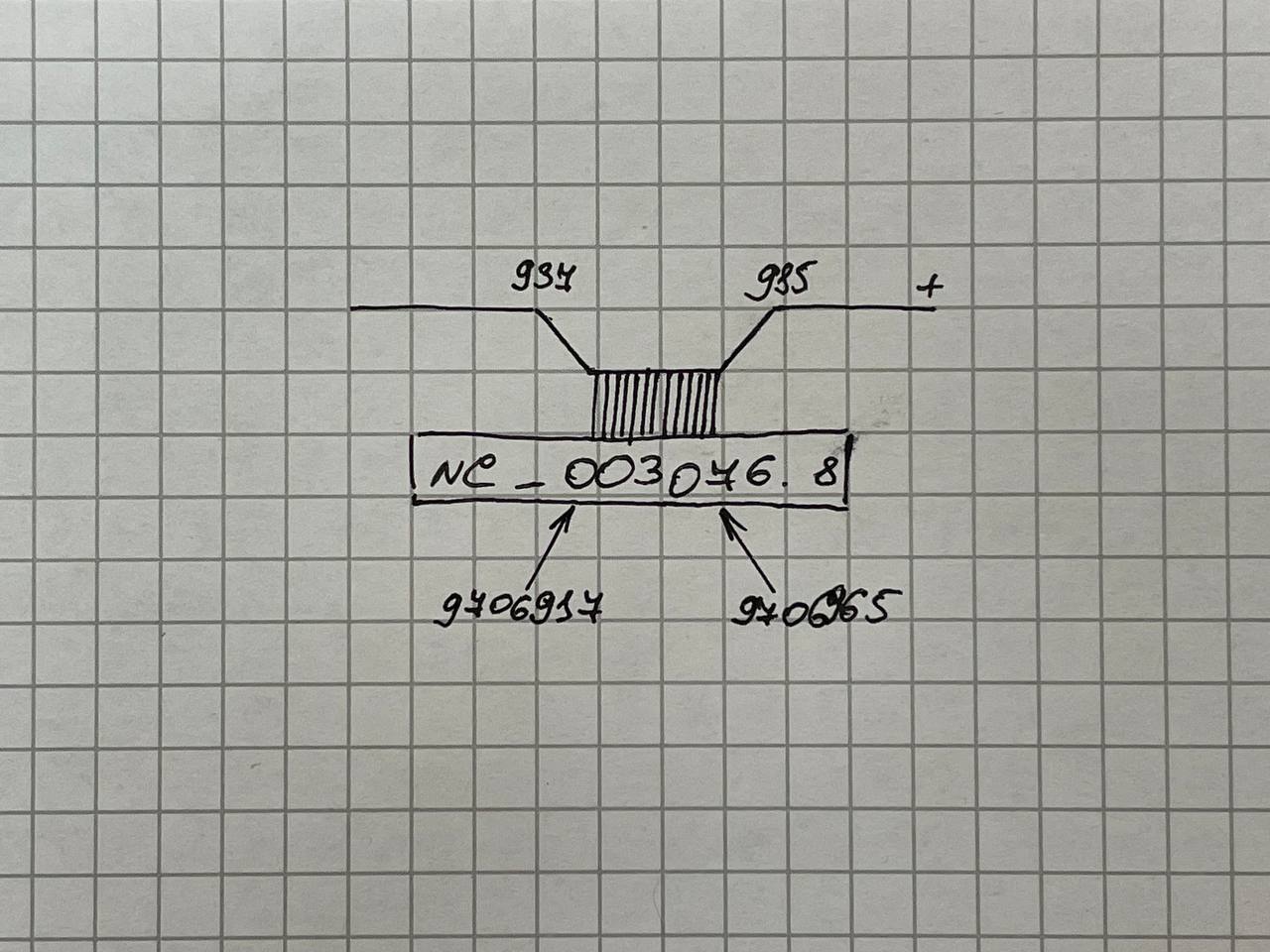
На рисунке ниже изображена схема 16S рРНК алгоритма BLAST
Для выполнения задания были выбраны два разных штамма кишечной палочки: Escherichia coli K-12 MG1655, Escherichia coli O157:H7 Sakai.
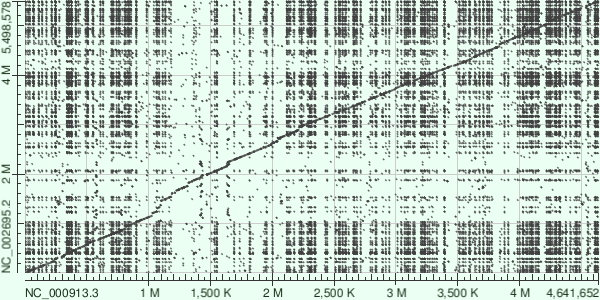
Диагональ посередине это большие консервативные блоки, сохранившиеся между штаммами. Много мелких делеций. Возможно есть транслокация в начале генома
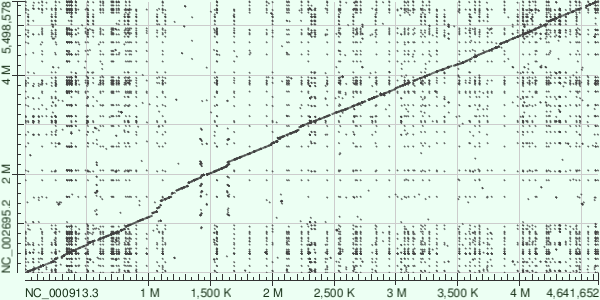
На карте BLASTN по всей площади графика есть большое количество мелких локальных совпадений. Это ожидаемо, так как blastn находит короткие участки гомологии. На главной диагонали видна линия сходства между двумя штаммами