Получение файлов со структурами, их изучение.
Заданный файл PDB - 1p8k был получен с сайта PDB (единственная выдача - файл 1p8k.pdb); а биологическая единица изучаемого комплекса - с сайта Macromolecular Structure Database EBI (файл 1p8k_mmol.pdb).Следующий этап - изучение полученных структур средствами RasMol.
Результаты: различий между структурами не обнаружено.
Имеются различия в размерах и, соответственно, содержании PDB-файлов: 1p8k.pdb содержит больше информации об "отдельных"
компонентах - ДНК и белке (в т.ч. поля REMARK; о последовательностях ДНК и белка - поле SEQRES; о модифицикации аминокислот - поле MODRES (также в HETATM; есть и брутто-формула - поле FORMUL);
о вторичной структуре белка (шпильки, стренды)- поля HELIX, SHEET и т.д. - не в том смысл, чтобы перечислить все поля файла PDB).
1p8k_mmol.pdb - тут информация, касающаяся комплекса ДНК-белок вцелом (без "идентификации" отдельных молекул).
О функциональных особенностях белка (а это интрон-кодируемая эндонуклеаза I-ANII)- см. п. 4.
В соответствии с заданием, дальнейшая работа ведется с файлом с биологической единицей.
Исследование контактов между молекулами белка и нуклеиновой кислоты.
Как в тексте полученных документов названы нужные атомы:- атомы остатка сахара - O5*, C5*, C4*, O4*, C3*, O3*, C2*, C1*;
- атомы остатка фосфорной кислоты - O1P, OP2, P;
- атомы азотистых оснований (на примере С) - N1, C2, O2, N3, C4, N4, C5, C6.
Тогда:
– pol_dr - полярные атомы 2'-дезоксирибозы;
– pol_ph - полярные атомы остатков фосфорной кислоты: " *.O?P " (это два атома кислорода: O1P и O2P);
– pol_bs_mjg (pol_bs_mig) - полярные атомы азотистых оснований, обращенные в большую (малую) бороздку;
– npolar_drнеполярные атомы 2'-дезоксирибозы: " *.C?* " (эквивалентно: "dna and backbone and carbon", поскольку все атомы углерода остова ДНК находятся в остатке сахара);
– npolar_bs_mjg (npolar_bs_mig)неполярные атомы ... бороздки: "carbon and ...".
Был выдан скрипт dna.def. В нем определены еще 3 множества:
base — все атомы оснований ДНК
(эквивалентно dna and not backbone);
mjg — атомы оснований ДНК, обращённые в большую бороздку
("major groove");
mig — атомы оснований ДНК, обращённые в малую бороздку
("minor groove").
При поиске полярных контактов использован порог расстояния в 3,5 Å, а при поиске неполярных — 4,5 Å (cоответственно, полярный контакт, или водородная связь, имеет место, если оба контактирующих атома полярны; неполярный контакт, или гидрофобное взаимодействие, предполагает, что оба атома неполярны).
Для выполнения поставленной задачи - исследования контактов между молекулами белка и ДНК - составлен данный скрипт ("2 в 1": определение
необходимых множеств + выделение исследуемых окрестностей; в поле echo дается "расшифровка" определений).
Результаты оформлены в таблицу "Число атомов белка, участвующих во взаимодействии с ...":
| Полярные | Гидрофобные | Всего | |
| Контакты белка с ... | |||
| ... остатками 2'-дезоксирибозы | 8 | 58 | 66 |
| ... остатками фосфорной кислоты | 29 | 34 | 63 |
| ... остатками азотистых оснований со стороны большой бороздки | 11 | 46 | 57 |
| ... остатками азотистых оснований со стороны малой бороздки | 0 | 0 | 0 |
Итак:
- число полярных контактов с остатками фосфорной кислоты больше числа полярных контактов с остатками 2'-дезоксирибозы более чем в 3 раза; это согласуется с функцией белка (режет остов ДНК по фосфатным группам);
- сравнительно велико число гидрофобных взаимодействий "белок - ДНК" (в сумме 92 для сахарофосфатного остова и 46 для азотистых оснований); возможно, это необходимо
для надежной "фиксации" ДНК и белка (необходимость при резке ДНК);
общее число контактов (и полярных, и гидрофобных) весьма велико - 186 (опять же, это связано с функцией - расщепить определенные уч-ки ДНК); - исследуемый белок взаимодействует с большой бороздкой ДНК (целых 11 полярных контактов в сравнении с 0 для малой бороздки; 46 гидрофобных контактов); вообще не контактирует с остатками азотистых оснований со стороны малой бороздки (ни гидрофобных, ни полярных взаимодействий нет). Понятно, что эндонуклеаза для узнавания ДНК располагается со стороны большой бороздки (в нее и азотистых оснований, в принципе, обращено больше).
Поиск специфических контактов, обеспечивающих узнавание сайта в молекуле ДНК.
Задание: "Предположить, какие остатки белка отвечают за специфичность узнавания белком ДНК. ".Для выполнения поставленной задачи необходимо посмотреть, какие аминокислотные остатки контактируют с атомами азотистых оснований большой бороздки ДНК ( образуя, соответственно, полярное и неполярное взаимодействия). Это нетрудно сделать, взяв за основу предложенный ранее скрипт. Cписок аминокислотных остатков, образующих полярные контакты (в скобках указаны соответствующие атомы) - следовательно, отвечающих за специфичность узнавания белком ДНК:
- Glu 35 (OE1)
- Arg 59 (NH1)
- Arg 61 (NH1, NH2)
- Arg 70 (NH1, NH2)
- Arg 72 (NH1, NH2)
- Tyr 154 (OH)
- Lys 200 (NZ)
- Lys 202 (NZ)
- Glu** 35
- Arg* 59 (NH1)
- Arg* 61 (NH1, NH2)
- Tyr****** 154 (OH)
- Lys* 202 (NZ)
Средствами RasMol построено изображение выбранного остатка и контактирующего с ним фрагмента ДНК в шарнирной модели:
Скрипт, генерирующий данное изображение, можно увидеть здесь.
В статье Structural and biochemical analyses of DNA and RNA binding by a bifunctional homing endonuclease and group I intron splicing factor (Jill M. Bolduc et al., 2003) я нашла подтверждение
своему предположению: все 4 указанных а.о. arg - Arg 59, Arg 61, Arg 70, Arg 72 - отвечают за специфичность узнавания белком такой пуриновой последовательности ДНК: 5'-GGAGG-3'.
Статья доступна на этом ресурсе.
Описание функций исследованного белка.
Найдено описание заданного белка в UniProt и описание соответствующего ДНК-связывающего домена в Pfam (была выбрана ссылка PF00961 в поле PFAM - это и есть ДНК-связывающий домен).О белке: он совмещает 2 функции: это интрон-кодируемая ДНК эндонуклеаза I-AniI и мРНК матураза bI1.
- С одной стороны, белок разрезает ДНК в месте соединения 2х экзонов cobA гена (в безинтронных генах), способствуя тем самым вставке интрона, содержащего последовательность данного белка (группа I интронов; РНК-копии таких участков ДНК являются рибозимами – каталитически активными молекулами РНК; способны к аутосплайсингу ). Этот процесс еще называют "homing'ом" интрона (а эндонуклеазы - 'homing' эндонуклеазами (см. пояснения далее)). Узнавание специфично (последовательности, по которым происходит узнавание ,большие), "резка" происходит по последовательности 5'-GAGGAGGTTTCTCTGTA-3'. Действие - на митохондриальную ДНК. В этом роль белка как эндонуклеазы.
- С другой стороны, белок катализирует процесс вырезания интронов из пре-мРНК и осуществляющий "созревание" мРНК (стабилизация вторичной/четвертичной структур мРНК). В этом его функция как матуразы.
- Cайты узнавания РНК и ДНК и каталитические сайты разобщены (независимы функции эндонуклеазы и матуразы).
- Кофактором является ион Mg2+ - его наличие необходимо для I-AniI эндонуклеазной активности.
- Белок относится к LAGLIDADG семейству эндонуклеаз.
- Гомодимер.
- Дислокация в клетке - в митохондриях.
- Организм - "источник" - Emericella nidulans (Aspergillus nidulans).

Еще одна "визуализация" (теперь изображены только домены LAGLIDADG):
B Pfam было получено множественно выравнивание (оба типа: "сокращенное" - из 91 последовательности и "расширенное" (полное) - из 508 последовательности). Выравнены последовательности доменов, а не всего белка.
Значит номер 61 (по последовательности, см. файл 1p8k_mmol.pdb) аминокислотного остатка Arg 61 не будет соответствовать 61ой позиции выравнивания.
Рассматриваем "сокращенный" вариант выравнивания. И вот что получается: среди консервативных а.о. не оказывается рассмотренного ранее Arg 61. Причем даже не требовалось полной консервативности!!!! Далее открылась очень интересная вещь: когда я попыталась в выравнивании
в строке, соответствующей выравниванию рассматриваемого белка, найти целиком участок VSFRKR (это по нумерации pdb остатки с 56 по 61), потерпела неудачу. Так был найден только участок, отмеченный желтым цветом (в нумерации pdb остатки с 56 по 59). А этого участка - совершенно иная последовательность, нежели все в том же pdb. Тогда я вспомнила об обстоятельстве, смутившем меня
при получении информации из UniProt: в единственном(!) файле-выдаче UniProt содержится информация о белке в 488 аминокислотных остатка - предшественнике интрон-кодируемой ДНК эндонуклеазы I-AniI; в файле содержится цитохром b.
Но вот, опять же, с полной доменной структурой я разобралась выше: cytochrome b соответствует зеленому блоку на схеме, т.е. перед LAGLIDADG доменами (т.о.
его включение не должно повлиять на выравнивание и, тем более, "разобщение" pdb-шных участков 56-59 и 60-61).
Asp61 не оказался консервативным. Это может быть связано только с названной выше проблемой. Хотя... еще одно объяснение (разумное ли?:-)): как я уже говорила, белки данного семейства кодируются ORF'ами, имеются ограничения на их длину, позицию. М.б. поэтому не консервативен Asp61. Но это как-то маловероятно.
Построение схемы контактов белка с ДНК.
Программа nucplot предназначена для визуализации контактов между ДНК и белком. Запускается на сервере kodomo-count с использованием команды:nucplot 1p8k_mmol.pdbВ выдаче получено несколько файлов. Графическое изображение контактов содержится в файле nucplot.ps (всего 4 страницы). Привожу вторую страницу:
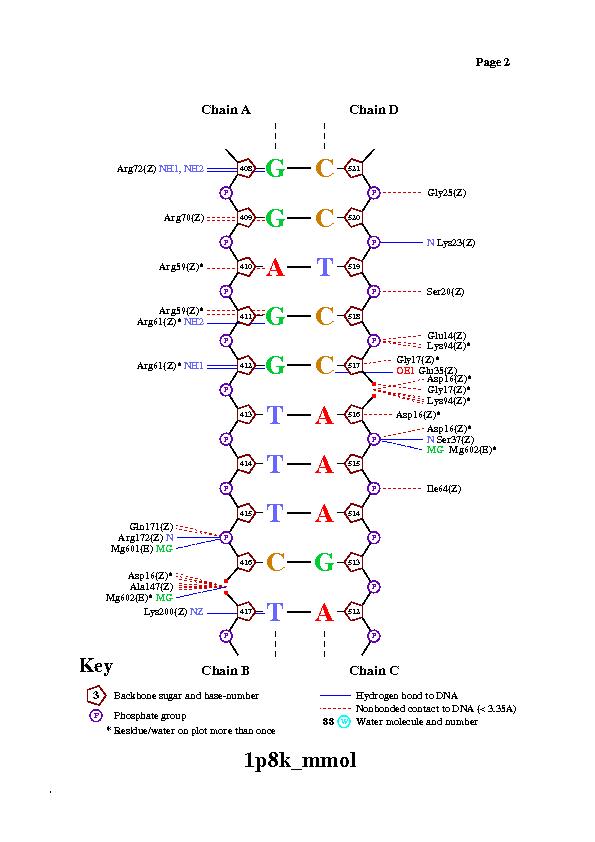
Изучив данную схему, видно следующее:
- общее число контактов значительно меньше общего числа контактов, указанных в таблице. Это можно объяснить тем, что алгоритм nucplot предъявляет более жесткие требования к определению взаимодействий (полярное/неполярное), нежели расстояние в 3.5/4.5 ангстрем между одновременно полярными/неполярными атомами. Особенно меньше число гидрофобных взаимодействий;
- для особенно интересующей меня аминокислоты - Asp61 не оказалось только гидрофобного взаимодействия, в то время как оба полярных контакта есть (для Lys 202, например, полярных взаимодействий программа не выявила); а значит действительно можно предполагать, что она отвечает за специфичность узнавания белком ДНК.