- PDB-код: 1sz1
- В файле описана тРНК нуклеотидилтрансфераза [тРНК аденилилтрансфераза, тРНК ССА-пирофосфорилаза, ССА-добавляющий (ССА-восстанавливающий) энзим]
- Организм-"источник": ARCHAEOGLOBUS FULGIDUS (систематика: Archaea; Euryarchaeota; Archaeoglobi; Archaeoglobales; Archaeoglobaceae; Archaeoglobus); система экспрессии - ESCHERICHIA COLI.
- тРНК (76-MER); идентификаторы цепей тРНК: цепи E и F
- Помимо 2х цепей тРНК - цепей E и F, в PDB-файле также содержатся 2 белковые цепи - цепи А и В.
В дальнейшем работа будет вестись с цепью Е.
G C G G A U U U A 2MG C U C A G H2U H2U G G G A G A G C M2G C C A G A OMC U OMG A A YYG A PSU 5MC U G G A G 7MG U C 5MC U
G U G 5MU PSU C G 1MA U C C A C A G A A U U C G C A C C A
В состав тРНК входят модифицированные основания (выше - выделены жирным шрифтом):
| Обозначение | Название | Брутто-формула |
| 2MG | 2N-метилгуанозин-5'-монофосфат | 2(C11 H16 N5 O8 P1) |
| H2U | 5,6-дигидроуридин-5'-монофосфат | 4(C9 H15 N2 O9 P1) |
| M2G | N2-диметилгуанозин-5'-монофосфат | 2(C12 H18 N5 O8 P1) |
| OMC | О2'-метилцитидин-5'-монофосфат | 2(C10 H16 N3 O8 P1) |
| OMG | О2'-метилгуанозин-5'-монофосфат | 2(C11 H16 N5 O8 P1) |
| YYG | модифицированный гуанозин-5'-фосфат (синоним к 4-(3-[5-O-PHOSPHONORIBOFURANOSYL]-4,6-DIMETHYL-8-OXO-4,8-DIHYDRO-3H-1,3,4,5,7A-PENTAAZA-S-INDACEN-YLAMINO-BUTYRIC ACID METHYL ESTER) | 2(C21 H29 N6 O12 P1) |
| PSU | псевдоуридин-5'-монофосфат | 4(C9 H13 N2 O9 P1) |
| 5MC | 5-метилцитидин-5'-монофосфат | 4(C10 H16 N3 O8 P1) |
| 7MG | 7N-метил-8-гидрогуанозин-5'-монофосфат | 2(C11 H18 N5 O8 P1) |
| 5MU | 5-метилуридин-5'-монофосфат | 2(C10 H15 N2 O9 P1) |
| 1MA | 6-гидро-1-метиладенозин-5'-монофосфат | 2(C11 H18 N5 O7 P1) |
Kак пронумерована цепь РНК в PDB-записи: 1ый рибонуклеотид - гуанозин-5'-фосфат - номер 1; последний рибонуклеотид - аденозин-5'-фосфат - номер 76; вставок/пропусков в нумерации нет.
restrict *eCруктура тРНК проанализирована программой find_pair - результаты тут; команда:
save H:/Term3/Practice8/1sz1_e.pdb
find_pair -t 1sz1_e.pdb 1sz1_e.fpВ выдаче получается 3 спирали: спираль 1 и спираль 2 (обе - длиной в 14 пар нуклеотидов; в первой - 3 не Уотсон-Криковских взаимодействия; во второй спирали таких взаимодействий 6 штук) и спираль 3 (длина = 1 пара нуклеотидов). Не совсем понятно, что представляет собой 3ья спираль в 1 пару оснований (но к этому вопросу я вернусь при рассмотрении структуры в RasMol)... Был смысл повторить программу find_pair добавив еще одну опцию - опцию -d (для получения отдельных файлов для каждой из спиралей). См. результаты и подробности про каждую из спиралей тут - 1sz1_e_h.fp_0001, 1sz1_e_h.fp_0002, 1sz1_e_h.fp_0003.
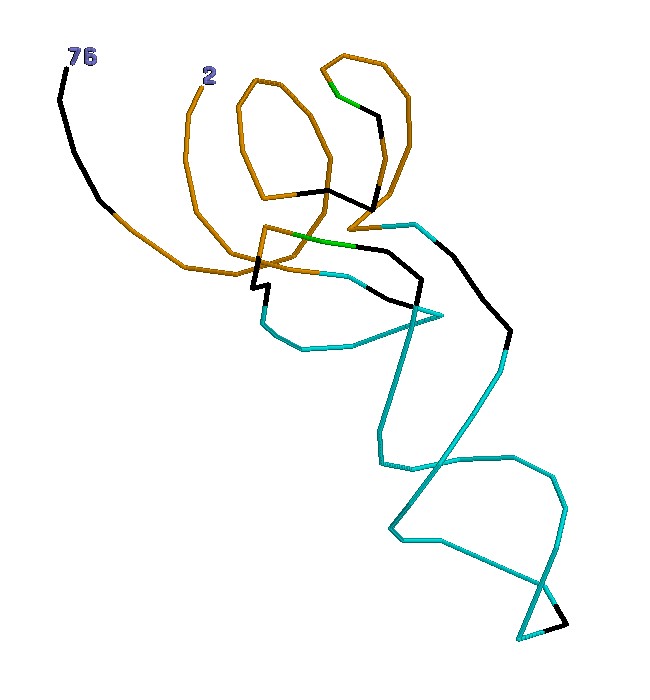
На последовательности тРНК разными цветами отмечены участки, входящие в разные спирали (спираль1, спираль2, спираль3):
1 * * * * * * * * 10 * * * * * * * * *20 * * * * * * * * *30 * * * * * * * * * 40 * * * * * * * * * 50
G C G G A U U U A 2MG C U C A G H2U H2U G G G A G A G C M2G C C A G A OMC U OMG A A YYG A PSU 5MC U G G A G 7MG U C 5MC U
51* * * * * * * *60 * * * * * * * * *70 * * * * *76
G U G 5MU PSU C G 1MA U C C A C A G A A U U C G C A C C A
background white wireframe off backbone 60 restrict *e color black define hel1 1-7:e, 18:e, 49-55:e, 58:e, 61-72:e define hel2 8:e, 10-15:e, 22-33:e, 36:e, 38-44:e, 48:e define hel3 19:e, 56:e select hel1 color orange select hel2 color cyan select hel3 color green select *.p and 2:e label 2 select *.p and 76:e label 76 set fontsize 14 set fontstroke 2 color labels blackЗатем - поиск наиболее удачного ракурса и экспорт в GIF. Результат (предварительно импортировано в JPEG):
Результаты/выводы:
- не Уотсон-Криковскиe взаимодействия (в скобках - нуклеотиды, без скобок - их нумерация):
- 4 [G]-----[U] 69
- 14 [A]-----[U] 8
- 15 [G]-----[C] 48
- 36 [A]-----[U] 33
- 38 [A]-----[OMC] 32
- 39 [PSU]-----[A] 31
- 44 [A]-----[M2G] 26
- 54 [5MU]-----[1MA] 58
- 55 [PSU]-----[G] 18
- примеры внеспирального стекинг-взаимодействия между основаниями (сначала "красим"
основания спиралей (с использованием скрипта rna.txt), затем - глазами ищем стекинг-взаимодействия у непокрашенных оснований - "стопочки" оснований):
- A73 - C74 - C75
- A21 - 7MG 46 - A9 - G45
- на какую из форм ДНК похожи спирали РНК? Для ответа на этот вопрос необходимо оценить 2 главных параметра: число нуклеотидов на виток и шаг спирали. Сравним
с ранее полученными результатами:
A-форма B-форма Файл тРНК (цепь 1) Тип спирали (правая или левая) правая правая правая Шаг спирали (Å) 28.02 33.75 28.96 Число оснований на виток 11 10 11 Ширина большой бороздки 7.98 17.21 11.05 Ширина малой бороздки 16.80 11.69 16.04 Шаг спирали измерялся от С61 до С72; большая бороздка: А64 - G3; малая бороздка: А64 - G53.
Становится ясно, что спирали тРНК (аналогичны результаты и для спирали 2) похожи на А-форму ДНК: одинаковое число нуклеотидов на виток; сходное значение шага спирали; одинаковый тип спирали (правая); ширина большой бороздки меньше ширины малой бороздки.
О третьей "спирали": find_pair определил 3 спирали в цепи Е тРНК; последняя - 3ья спираль - состоит всего лишь из одной пары комплементарных нуклеотидов (правда, find_pair определяет ее как 1 изолированную пару нуклеотидов). Спиралью такую структуру назвать все же сложно; вообще, смотрится в RasMol как продолжение спирали 1. Очевидно, ее отнесение к отдельной спирали связано с особенностями алгоритма find_pair.В файле 1sz1_e.out (результат работы analyze) подтверждено, что цепь тРНК относится к А-форме (критерии те же плюс, например, конформации рибозы - преимущественно это C3'-endo (как у дезоксирибозы А-формы)).
einverted 1sz1_e.fasta
Параметры приняты по умолчанию, кроме параметра "Minimum score threshold", равного 15. Соответственно, возможные двуспиральные участки:
SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps
12 tcagt 16
|||||
35 agtca 31
SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps
49 ctgtg 53
|||||
65 gacac 61
Цветовое решение соответствует цветам, использованным в пункте 3 (1ое выравнивание/последовательности - для 2ой цепи; 2ое выравнивание/последовательности - для 1ой цепи).
Результаты поиска, на мой взгляд, нельзя назвать удачными. Вроде бы и определены участки потенциальных двуспиральных участков, соответствующие 2м спиралям, но, во-первых, их меньше, чем могло бы быть (сравним, сколько участков последовательностей тРНК составляют спирали 1 и 2 - см. п. 3 этого задания; объяснение: при использовании einverted не учитываются модифицированные основания [определено вводимой последовательностью]). Bо-вторых, 1ое выравнивание (т.е. вроде как соответствующее 2ой спирали), оказывается "неверным" - очевидно пространственное несоответствие: программа find_pair для спирали 2 отражает такие двуспиральные участки:
12 [U] - [A] 23 13 [C] - [G] 22 14 [A] - [U] 8 15 [G] - [C] 48
два последних взаимодействия - не Уотсон-Криковские36 [A] - [U] 33 38 [A] - [OMC] 32 39 [PSU] - [A] 31
все три взаимодействия - не Уотсон-Криковские;
с этим и могут быть связаны "расхождения" при использовании программы einverted. Эту мысль подтверждает и то, что в случае "правильного" выравнивания для 1 спирали не Уотсон-Криковских взаимодействий не было.
mfold SEQ=1sz1_e_mf.fasta P=10Чем больше значение параметра P, тем больше вариантов предсказания выдается.
Сначала ,в соответствии с подсказками, в fasta-последовательности модифицированные основания были заменены буквой N (последовательность - см. тут). Как я ни меняла с такой последовательностью значение параметра Р, вторичная структура была явно далека от реальности (ведь комплементарных взаимодействий с N не образуется; далее - пример при Р=10):
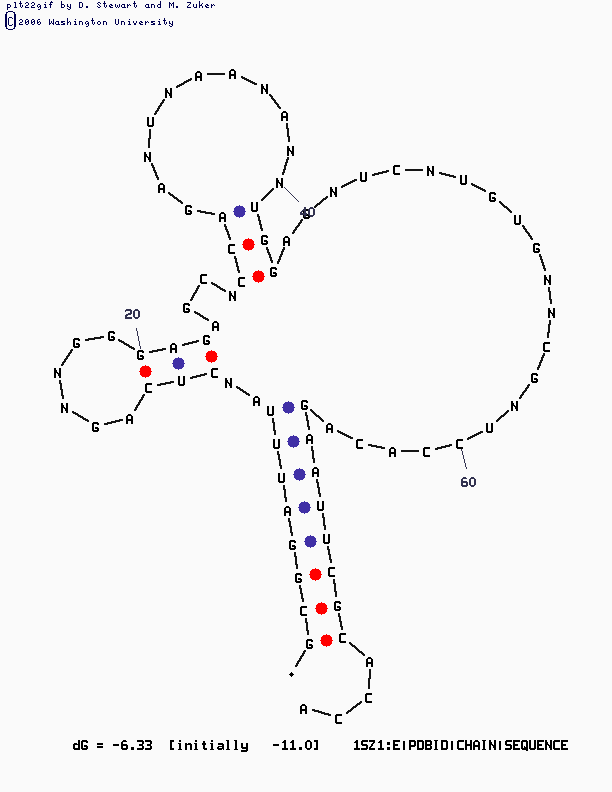
Поэтому эффективнее на вход подавать нуклеотидную последовательность с заменой модифицированных оснований на наиболее сходные канонические. Команда:
mfold SEQ=1sz1_e_mf_1.fastaВ первой же выдаче есть картинка ("вторая из двух") вторичной структуры, максимально близкая к реальности (дальше я изменяла параметр Р, но "эталоном" осталась именно эта вторичная структура: во-первых, минимальна энергия Гиббса; во-вторых, очень даже "приличными" оказались число азотистых оснований в антикодоновой петле (должно быть 7), "удачно" расположение антикодона - GAA - это OMG A A; в-третьих, согласуется с записью в pdb и предсказанием спиральных участков):
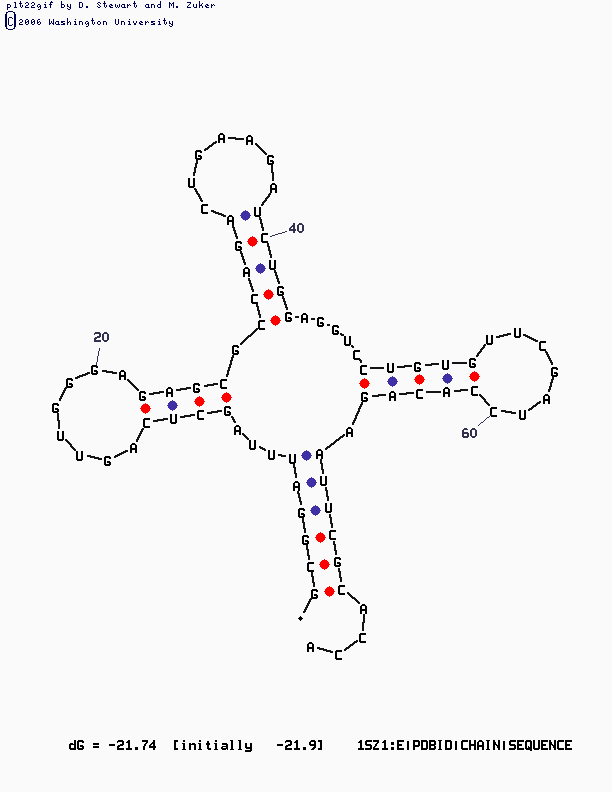
Программа mfold весьма и весьма полезна. Но для получения приемлимо-разумного результата требует максимум внимания со стороны пользователя.