Использование средств специализированного ресурса Brenda для характеристики фермента EC 6.1.1.11 - АТФ-зависимая серил-тРНК лигаза.
Все упражнение выполняются с использованием опции "Quick search" , т.е. начиная с главной странички. Выбирается для поиска вкладка "EC-Number", а затем в окошко поиска вводится 6.1.1.11. Получена строчка, в которой можно найти ссылки на схему ферментативной реакции, перечень всех последовательностей и полное описание.
Вот схема ферментативной реакции с участием рассматриваемого фермента (по данным BRENDA):
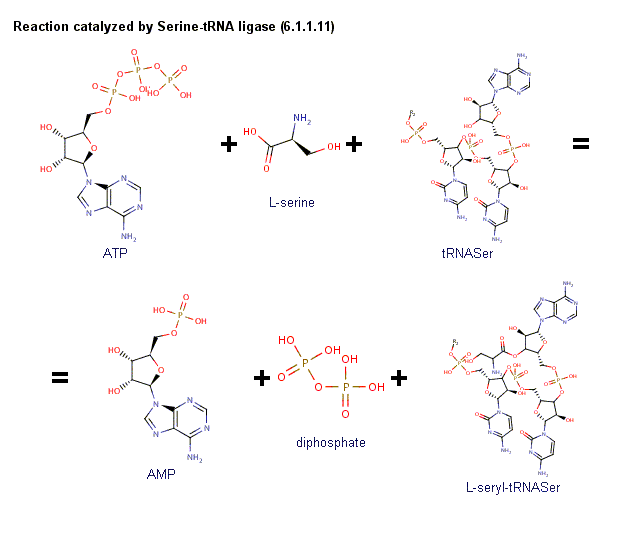
- Сколько всего ферментов с таким кодом описано в БД?
В БД BRENDA описаны 323 последовательности ферментов с EC 6.1.1.11; - Какая субъединичная структура характерна для ферментов данного типа?
Димер (поле SUBUNITS); - Какие бывают пострансляционные модификации у таких ферментов?
Посттрансляционные модификации данного типа фермента не описаны (поле Posttranslational Modification - "No entries in this field"). - Какие болезни человека так или иначе связаны с данным ферментом?
Болезней, связанных с данным ферментом, неописано (поле DISEASE - "No entries in this field"). Но логично предположить,
что в случае отсутствия такого фермента (отвечающего за образование комплекса серин-РНК) нарушается нормальный синтез белка;
это влечет за собой гибель организма на самых ранних этапах развития.
Сравнение аннотаций функций в БД GOA у ферментов из далеких организмов. Сравнение этих аннотаций с аннотациями в UniProt ( SRS ).
Работа ведется с белками, которые были найдены и исследованы в обязательном упражнении №2 (поиск ведется по AC UniProt). Это 4 белка: 2 из организма человека (SYSC_HUMAN, SYSM_HUMAN), 1 белок из кишечной палочки Escherichia coli К 12 (SYS_ECOLI) и 1 из Methanococcus jannaschii (SYS_METJA).* Запрос в SRS на эти белки выглядят так:
- ((([uniprot-ID:SYSC_HUMAN*] | [uniprot-ID:SYSM_HUMAN*]) | [uniprot-ID:SYS_ECOLI*]) | [uniprot-ID:SYS_METJA*])
Аннотирование в GOA:
| Белок | Клеточный компонент (component) | Биологический процесс (process) | Функция (function) |
|---|---|---|---|
| SYSC_HUMAN (АС P49591) | 2 ассоциированных термина GO;
|
4 ассоциированных терминов GO;
|
6 ассоциированных терминов GO;
|
| SYSM_HUMAN (АС Q9NP81) | 1 ассоциированный термин;
|
3 ассоциированных термина GO;
|
5 ассоциированных терминов GO;
|
| SYS_ECOLI (АС P0A8L1) | 0 ассоциированных терминов | 3 ассоциированных термина;
|
5 ассоциированных терминов GO;
|
| SYS_METJA (АС Q58477) | 0 ассоциированных терминов | 2 ассоциированных термина;
|
5 ассоциированных терминов GO;
|
Специфичность для всех ферментов - присоединение серина к тРНК.
Для Escherichia coli К 12 и Methanococcus jannaschii не ассоциирован ни один из терминов онтологии Component; а для рассматриваемых белков
из организма человека ассоциированные термины онтологии Component имеют коды подтверждения NR - Not recorded, IEA, ISS - это указывает на низкое качество аннотирования, значит "размещение" исследуемого фермента в клетке недостаточно точно описано.
Что касается онтологии Рrocess, то здесь количество аннотаций для 4х ферментов от 2ух (в случае Methanococcus jannaschii) до 4х (человек) [причем для белка SYSC_HUMAN, помимо трансляции, аминоацилирования тРНК для трансляции белка и серил - тРНК аминоацилирования, указано еще и участие в процессинге тРНК (код аннотации TAS)].
Однако "хорошие подтверждения" аннотаций этой онтологии есть только у белка человека SYSC_HUMAN (код TAS - для трансляции и участии в созревании тРНК); в других случаях процессы, в которых задействован фермент серин-тРНК лигаза, "определены" из электронных аннотаций (код IEA) либо на основании сходства последовательностей (код ISS).
По онтологии Function: число аннотаций практически одинаковое, но опять же, в случае белков человека - SYSC_HUMAN - имеются лучшие доказательства некоторых функций (код TAS для связывания РНК и связывания АТФ).
Теперь сравниваются аннотации БД GOA с аннотациями в UniProt. B UniProt в каждой из записей приводится только 3-4 ссылки на идентификаторы GO:
| Белок | Клеточный компонент (C) | Биологический процесс (P) | Функция (F) |
|---|---|---|---|
| SYSC_HUMAN (АС P49591) | |||
| SYSM_HUMAN (АС Q9NP81) | |||
| SYS_ECOLI (АС P0A8L1) | |||
| SYS_METJA (АС Q58477) |
Но это не следует понимать как "UniProt менее информативен, чем GOA", просто каждая из этих БД хороша по-своему: главное в БД UniProt - хранящиеся в ней последовательности (есть и другая ценная информация: например, ссылки на статьи; данные о структуре фермента и пр.); главное в GOA - хранящиеся в ней аннотации.
Использование средств специализированного ресурса Brenda для сравнения cвойств 2-х ферментов из далеких организмов.
Для сравнения выбраны серин-тРНК лигазы (EC 6.1.1.11) человека (SYSC_HUMAN) и кишечной палочки (SYS_ECOLI). Была проведена фильтрация серин-тРНК лигаз по указанным организмам. Оказалось, что оптимум рН (поле "pH OPTIMUM") приведен только для фермента из организма человека и составляет 7.6 - фермент работает в слабощелочной среде. Такие данные подтверждены результатами статьи Martha Heckl, Kristina Busch and Hans J. Gross (ссылка на PubMed приведена также в поле "pH OPTIMUM - LITERATURE" (кроме этого, по этой же ссылке можно получить список характеристик фермента, подтвержденных в этой статье)).
Данные об оптимуме для фермента кишечной палочки отсутствуют.
- Ингибиторы исследуемых ферментов не указаны (поле "INHIBITORS" - "No entries in this field").
- Активаторы исследуемых ферментов не указаны (поле "ACTIVATING COMPOUND" - "No entries in this field").
- Металлы/ионы, необходимые (возможно-?) для протекания ферментативной реакции, не указаны (поле "METALS and IONS" - "No entries in this field").
- Кофактором является АТФ (указано для фермента Homo sapiens, поле "COFACTOR").