При построении выравниваний использовались следующие программы пакета EMBOSS:
- needle - глобальное выравнивание
- water - локальное выравнивание
- matcher - локальное выравнивание
ЧАСТЬ 1 - Матрица переходов
Были даны последовательности:
VVIGA и VIG для глобального выравнивания
KLPVREFDA и LPFDA для локального выравнивания.
При построении матриц переходов использовались следующие параметры: вес совпадения = 2, вес замены = -1, штраф за делецию = -2.
| Глобальное выравнивание | Локальное выравнивание |
 |
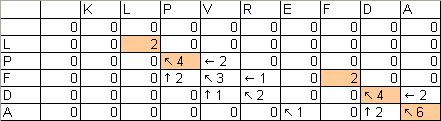 |
| Вес оптимального пути равен 3. Путь выбирался таким образом, чтобы в последней строчке число было максимальное, от него и выделялись цветом соответствующие ячейки. Само выравнивание: VVIGA VI-G- Кстати, можно заметить, что выравнивание построилось не совсем оптимальным способом, потому что последовательности придумывались на ходу. |
Вес оптимального пути равен 6, субоптимального равен 4. Полученное выравнивание: Субоптимальный путь: 2 LP 3 1 LP 2 Оптимальный путь: 7 FDA 9 3 FDA 5 |
ЧАСТЬ 2 - Поиск участков локальной гомологии
Анализ результатов, полученных с помощью программы построения локального выравнивания matcher.
Последовательности, для которых строилось выравнивание включали в себя последовательность белка SdhA(см. первый семестр):
REFDAVVIGAVNMEPKLRPA
При помощи данной программы можно определить координаты двух участков, из которых была построена вторая последовательность:
REFDAVVIGA - с 6 по 15
VNMEPKLRPA - с 571 по 580
Выравнивания:
SdhA ESESMTRRSVNMEPKLRPA
: .... .::::::::::
seq3 EFDAVVIGAVNMEPKLRPA
и
SdhA REFDAVVIGA
::::::::::
seq3 REFDAVVIGA
Matcher выдал еще третье выравнивание (так было указано при запуске программы), но из него выяснить координаты последовательности было нельзя, так как полностью они не совпадали.
ЧАСТЬ 3 - Влияние параметров на глобальное выравнивание
С помощью программы needle были построены глобальные выравнивания для тех же последовательностей, что и в части 2, используя матрицу аминокислотных замен EBLOSUM62. Но в одном случае за открытие гэпа давался штраф 10, а в другом 1, за продолжение гэпа в обоих случаях штраф был 1. Выравнивание для 1-го случая:
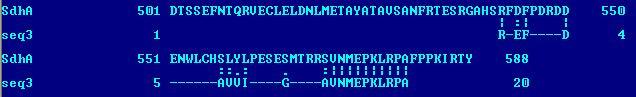
Для второго:
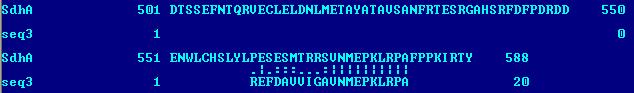
Различия видны очень хорошо - во втором случае гэпы отсутствуют, так как только за их открытие вычитается по 10 баллов, а цена выравнивания должна быть максимальной. Матрицы BLOSSUM для первого и второго выравниваний были одинаковы. Говорить о том, на сколько удачно проведены эти выравнивания трудно, так как seq3 получена слиянием двух кусков последовательности SdhA и поэтому смысла тут особенного не наблюдается.