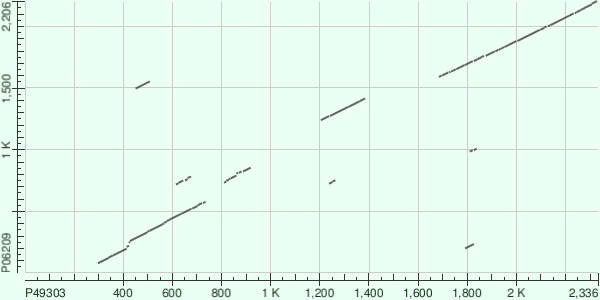
| Best | Second best | |
|---|---|---|
| Protein name | RNA-directed RNA polymerase* | Protein 2C |
| % identity | 29 | 48 |
| % positives | 49 | 61 |
| Length | 644|615 | 180|177 |
| Gaps | 59 | 5 |
| Score | 258 bits (660) | 158 bits (400) |
| *3D-POL у вируса ящура | ||
Вес в битах
Значения веса в битах рассчитывалось по приближенной формуле:
\begin{equation} S_b = 1 + \frac{S-M}{Q_1-M} \end{equation}Предположительно гомологичные белки (MOAE_BACSU/MOAE_ECOLI)
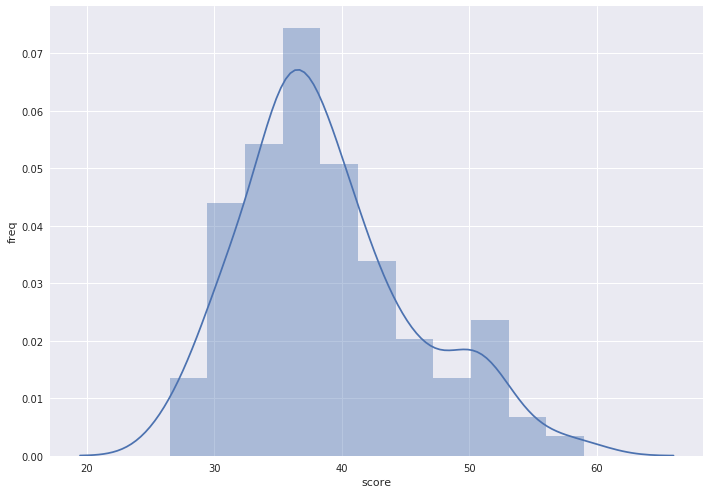
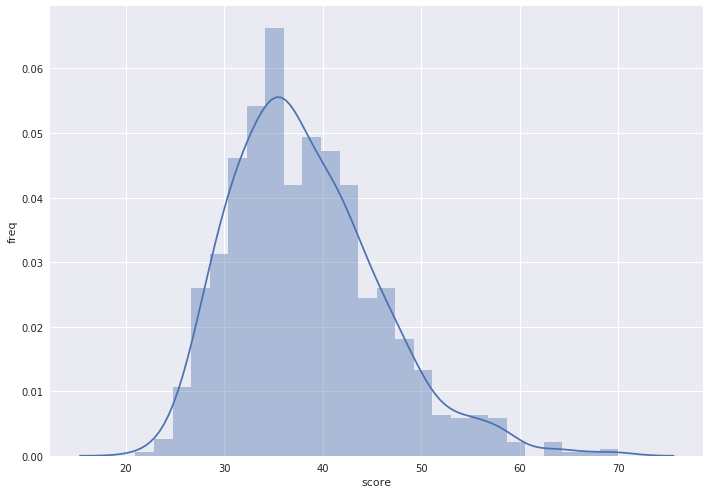
| shuffle 100 | shuffle 1000 | |
|---|---|---|
| Median | 37.50 | 37.00 |
| Quartile (75%) | 42.00 | 42.50 |
| Score (MOAE_BACSU/MOAE_ECOLI) | 201.00 | |
| Bit score approx. | 37.33 | 30.82 |
| P-value | $5.78 \times 10^{-12}$ | $5.27 \times 10^{-10}$ |
Примечательно, что от увеличения количества выравниваний в выборке вес изменился довольно значительно.
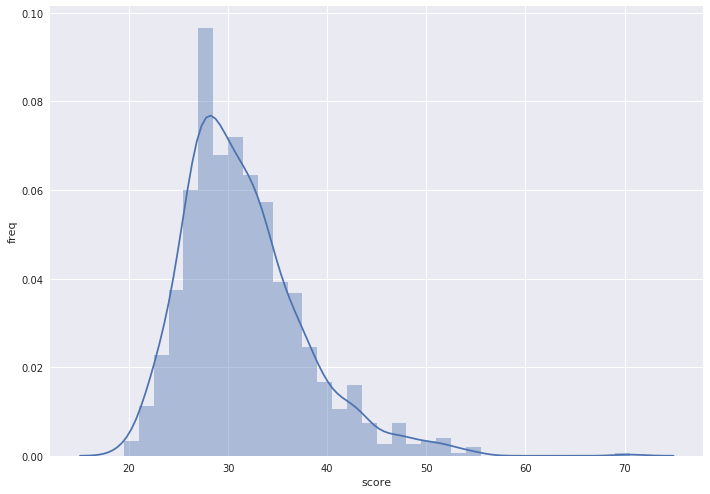
Предположительно негомологичные белки
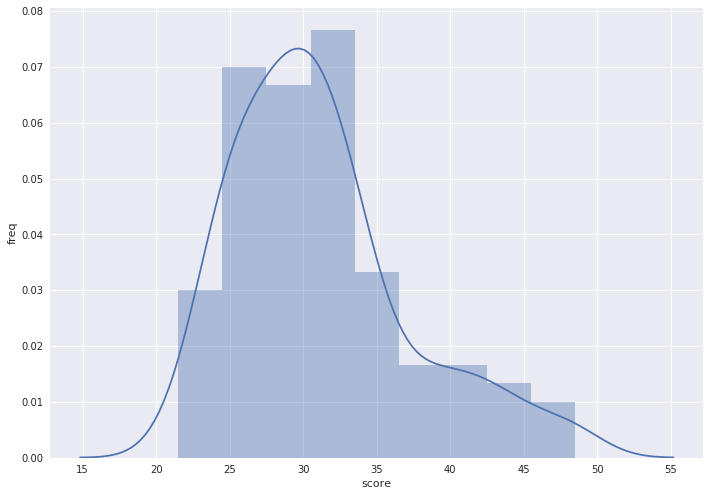
| shuffle 100 | shuffle 1000 | |
|---|---|---|
| Median | 30.25 | 30.50 |
| Quartile (75%) | 33.63 | 34.50 |
| Score (HIUH_BACSU/PTH_ECOLI) | 27.50 | |
| Bit score approx. | 0.19 | 0.25 |
| P-value | 0.88 | 0.84 |
Влияние матрицы и штрафа за продолжение гэпа
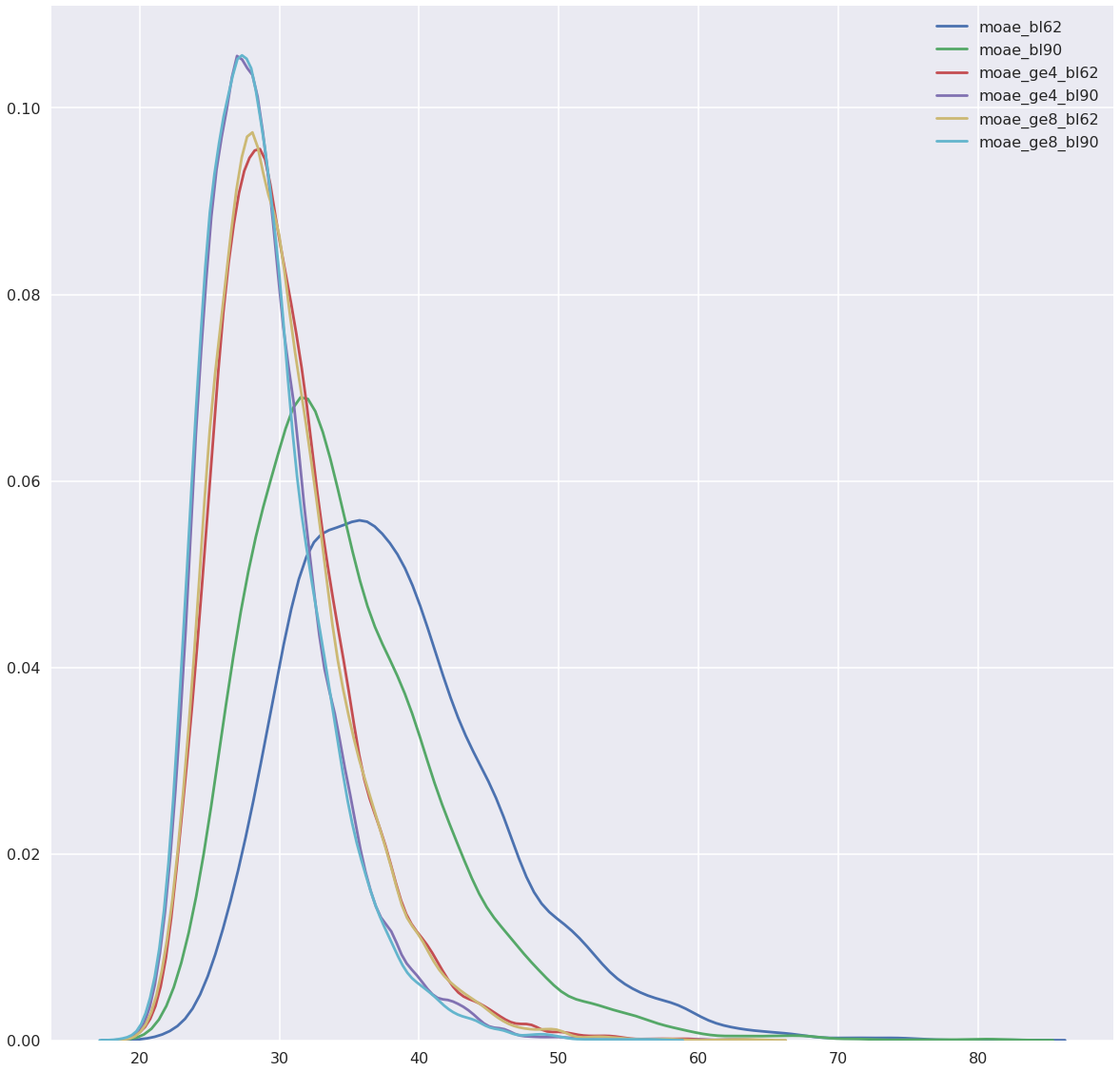
Для этого эксперемента были взяты белки с идентификаторами MOAE_BACSU и MOAE_ECOLI, которые рассматривались в прошлом практикуме.
С повышением штрафа за продолжение с 0,5 до 4 значительно сузилось распределение весов случайных выравниваний ($\sigma = 7.6 \rightarrow \sigma = 4.8$), медиана уменьшилась. Форма распределения при этом принципиально не изменяется ("тяжелый" правый хвост).
При замене матрицы медиана уменьшается тоже.
Из-за сужения распределения повышается вес оптимального выравнивания, так как уменьшается доля выравниваний, удаленных от медианы.
| Parameters | Bit score of the optimal alignment | |
|---|---|---|
| gapextend | matrix | |
| 0.5 | BLOSSUM62 | 30.8 |
| 4 | BLOSSUM62 | 58.0 |
| 8 | BLOSSUM62 | 44.0 |
| 0.5 | BLOSSUM90 | 31.8 |
| 4 | BLOSSUM90 | 53.7 |
| 8 | BLOSSUM90 | 53.7 |
BLAST
Идентификатор белка: ANF53595.1; Brevundimonas naejangsanensis
| Best | Second best | |
|---|---|---|
| Protein name | UDP-3-O-acyl-GlcNAc deacetylase | |
| Entry name (ID) | LPXC_PHEZH | LPXC_MAGSA |
| UniProt AC | B4RFF2 | Q2W0G6 |
| Organism | Phenylobacterium zucineum HLK1 | Magnetospirillum magneticum AMB-1 |
| % identity | 65 | 49 |
| % positives | 77 | 67 |
| Length | 291|291 | 290|292 |
| Gaps | 0 | 2 |
| Score | 371 bits (952) | 285 bits (729) |
| Expect | 7e-129 | 7e-95 |
| % coverage | 97 | 96 |
© Бушмакин Илья, 2018