Таксономия прочтенной последовательности
Для задания генировался консенсус с помощью consambig из пакета EMBOSS.
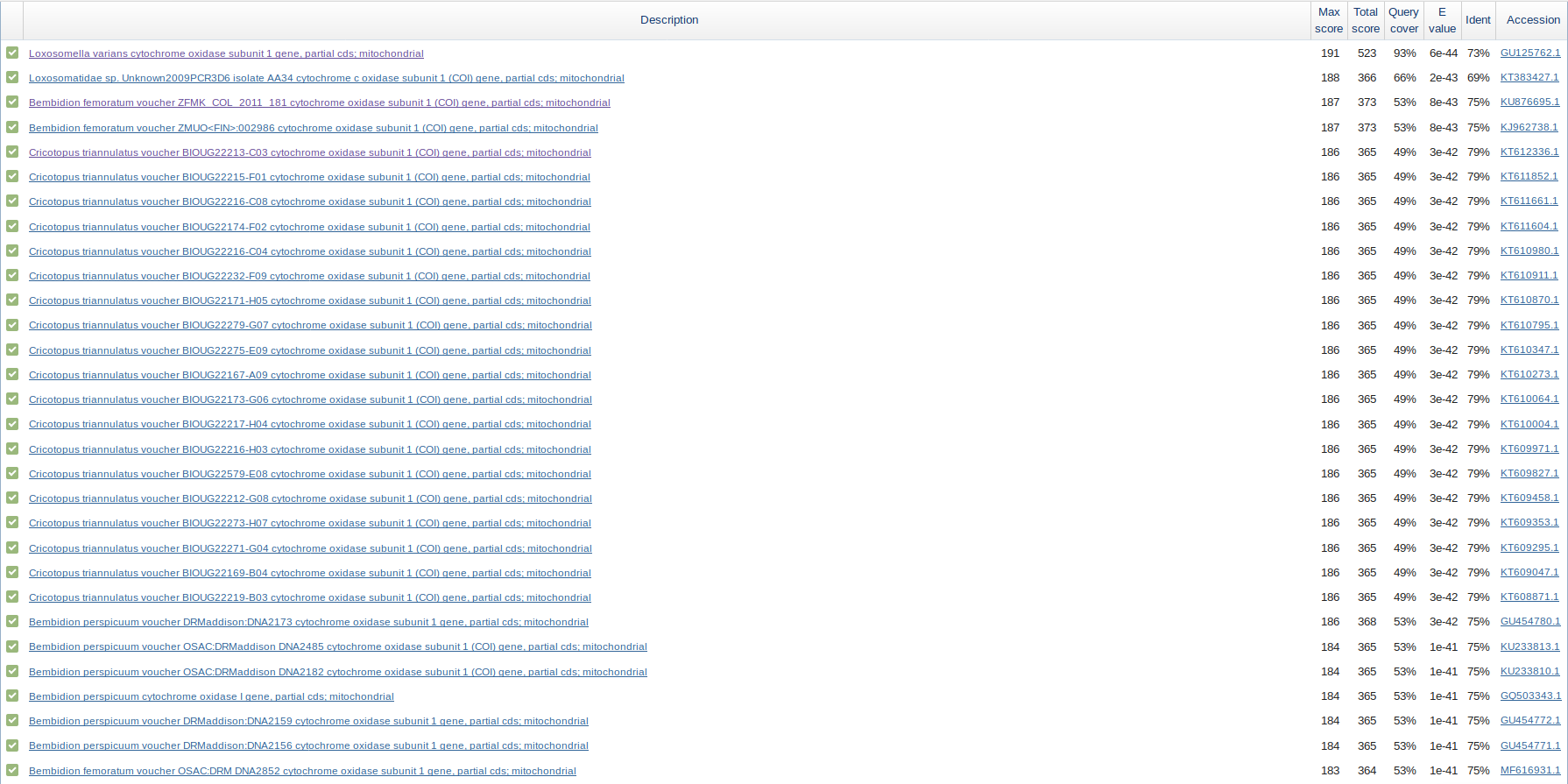
Поиск blastn по nr показал, что данная последовательность принадлежит первой субъединице цитохром оксидазы из ЭТЦ митохондрий. Среди первых 100 находок других генов не попадалось.
Среди первых четырех находок - два представителя семейства Loxosomatidae (E=6e-44, 2e-43) и два жука Bembidon fermoratum (E=8e-43) (3. Все остальные принадлежат Ecdysozoa, но находки имеют гораздо больший E-value (>. Вероятно, данная последовательность принадлежит виду Loxosomella varians, со скидкой на ошибки в прочтении нуклеотидной последовательности.
Сходство с последовательностями совершенно других таксонов может быть обЬяснено консервативностью последовательности цитохром оксидазы.
BLAST. Сравнение вариантов
Последовательность из первого задания
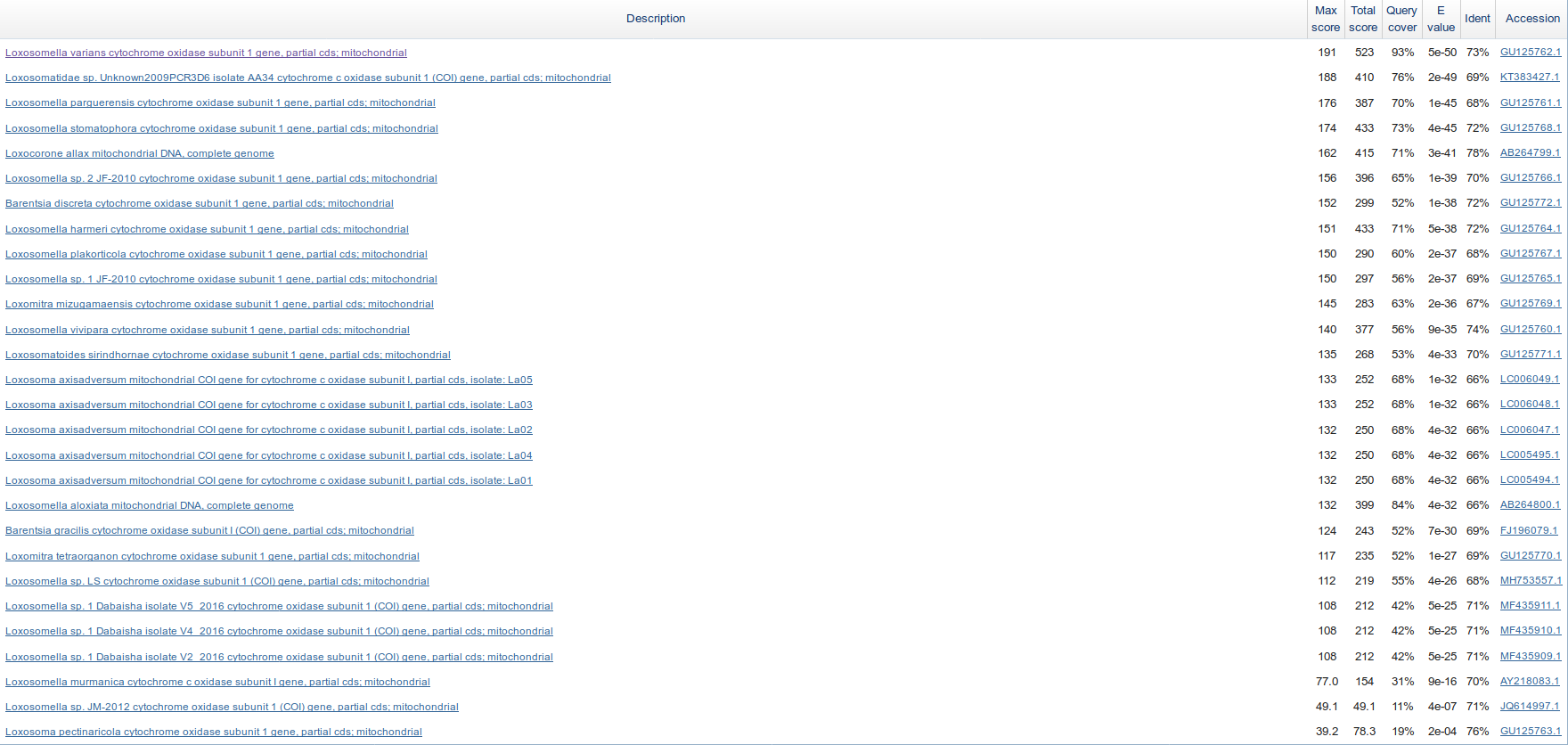
Использовался blast с сайта ncbi, E-value threshold = 0.001, против nr. Поиск ограничивался goblet worms.
| Критерий | megablast | blastn | blastn, чувствительный |
|---|---|---|---|
| Word length | 20 | 11 | 7 |
| Match/Mismatch | 1/-2 | 2/-3 | 1/-1 |
| Находок | 3 | 28 | 29 |

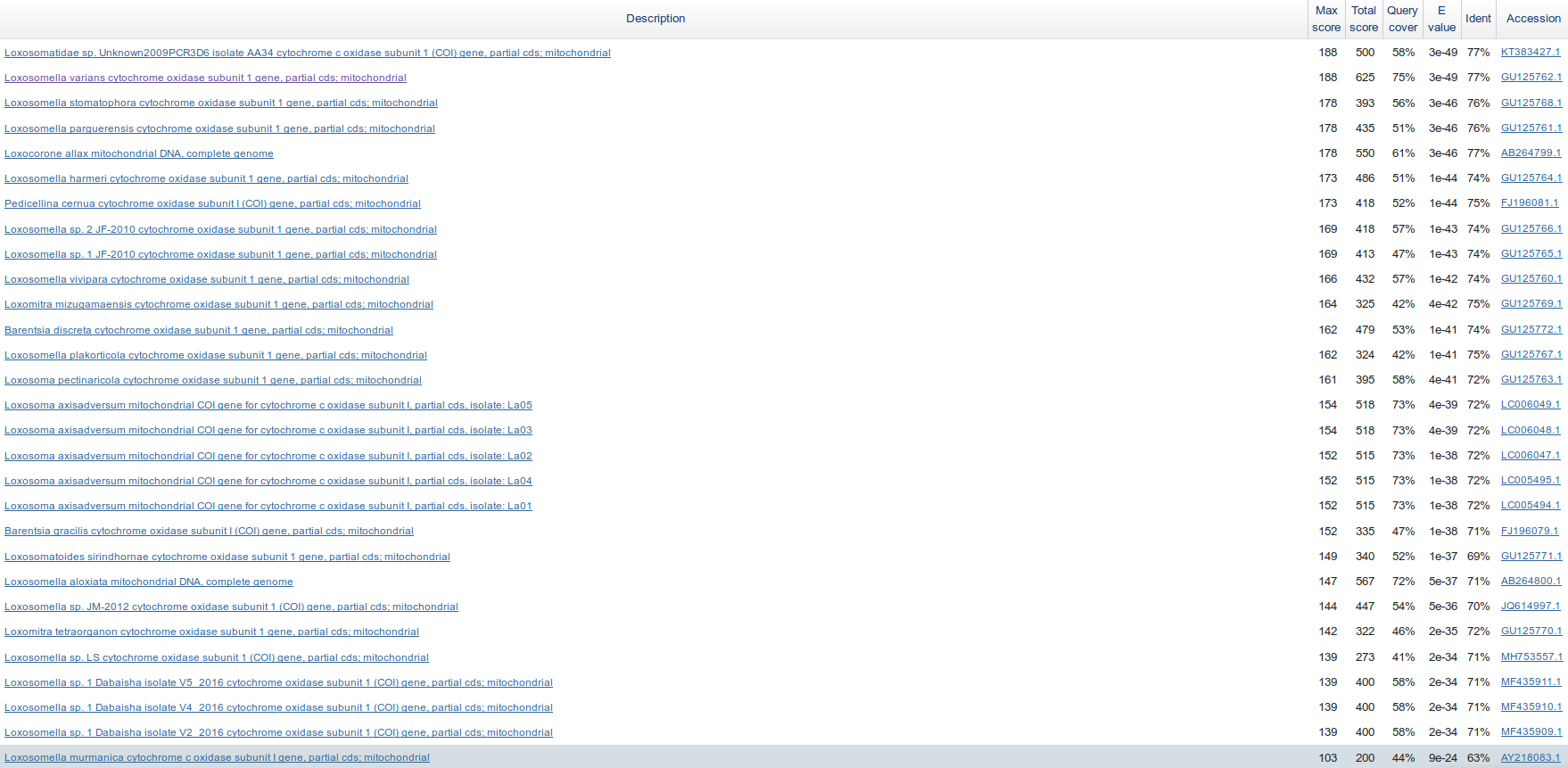
Сравнение результатов с rns из Mnemiopsis leidyi
Для этого сравнения blast-ы запускались с теми же входными параметрами, входная последовательность - ген рРНК малой субъединицы рибосомы митохондрий Mnemiopsis leidyi. Ниже приведено количество находок для разных алгоритмов.
megablast 1 blastn, default 93 blastn, sensitive 2325

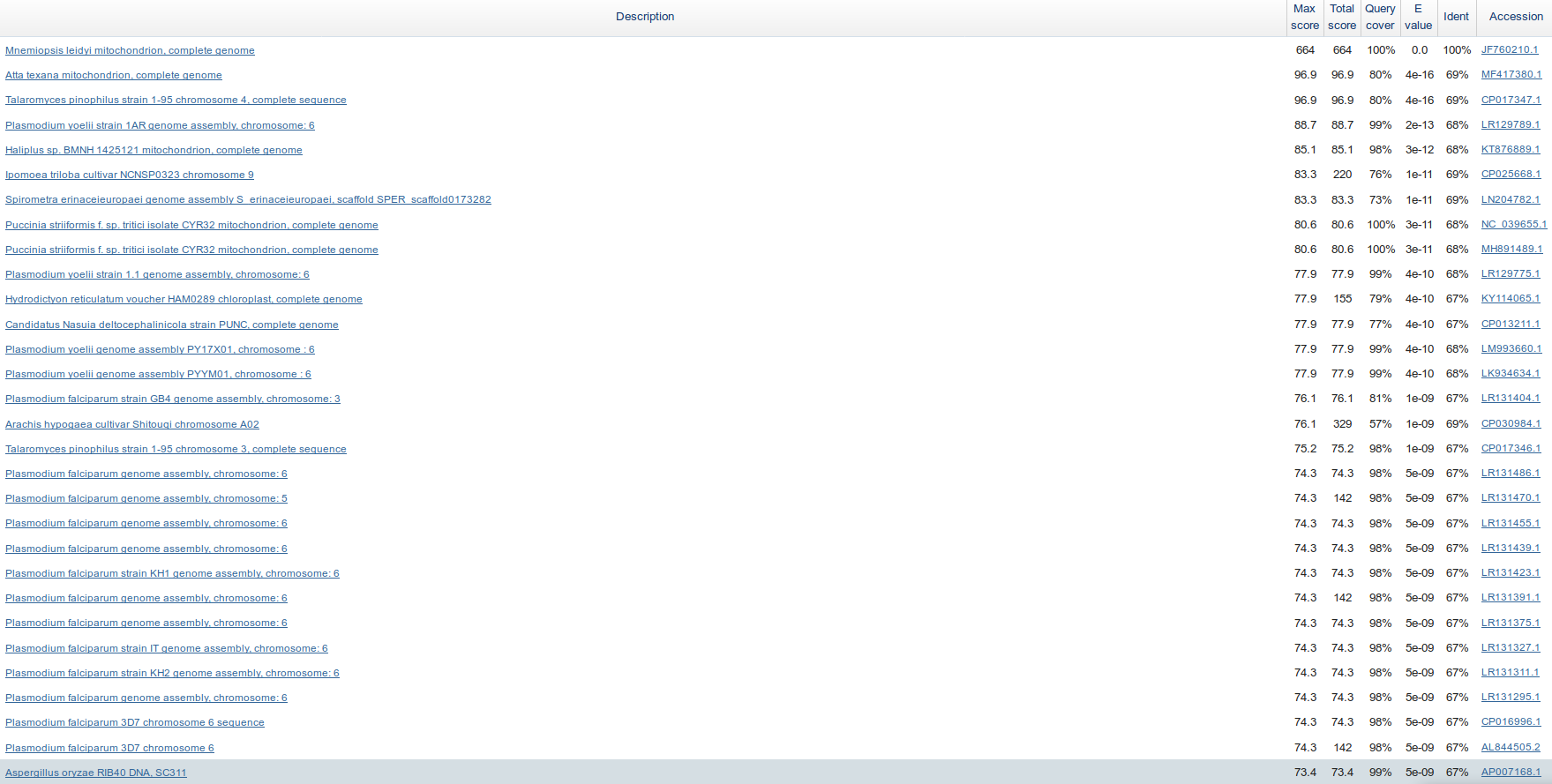
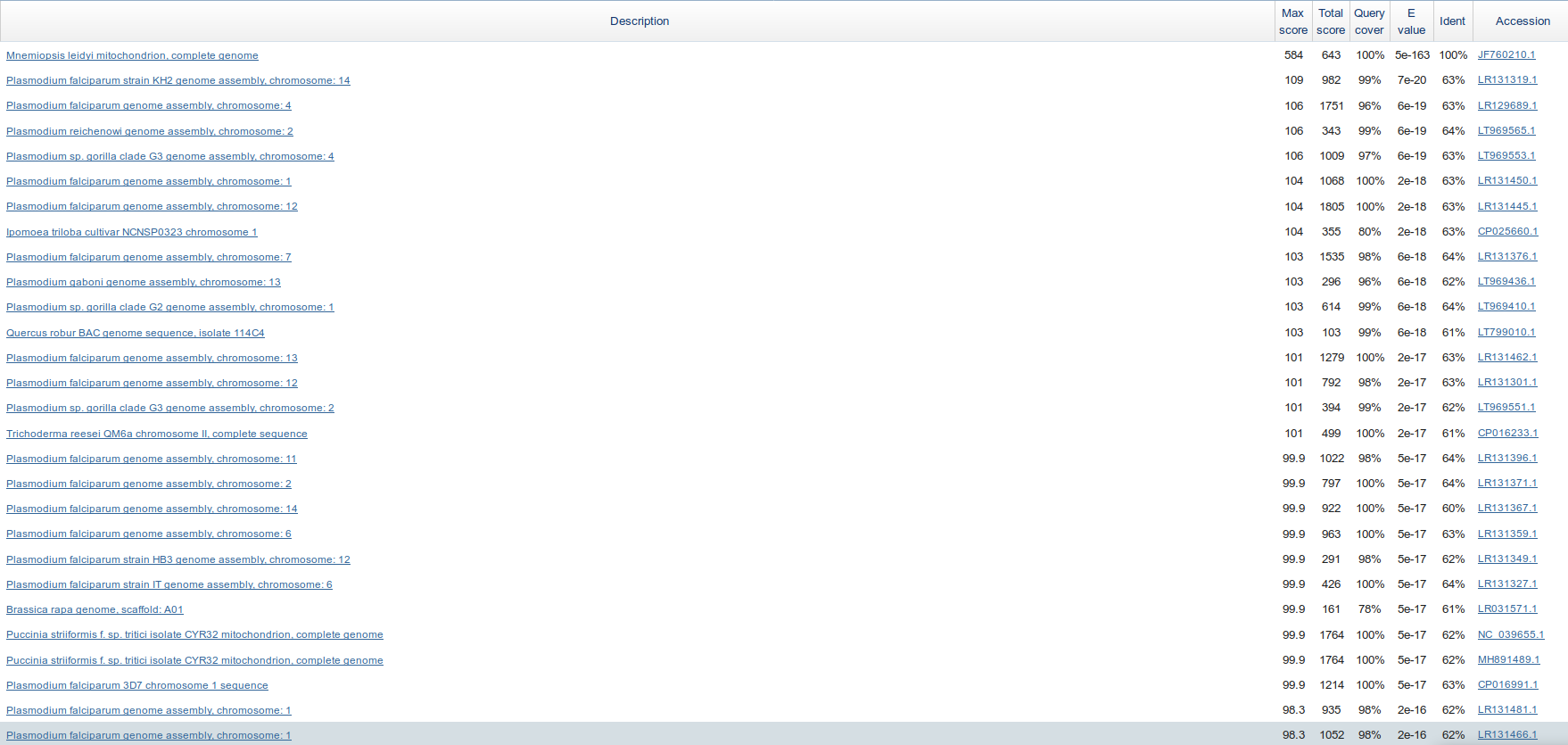
Megablast с длинной 28 не справлялся с поиском, видимо из-за сравнительно короткой последовательности в запросе. Чувствительный blastn не сильно отличается от blastn по-умолчанию, но на бОльшей выборке он выдает слишком много результатов.
Гомологи HSP71_YEAST, TERT_SCHPO, TBB_NEUCR в геноме Amoeboaphelidium protococcarum
Для этой части работы использовался tblastn, с запросами в виде последовательности из UniProt.
tblastn -query query.fasta -db X5.fasta -outfmt 7 > outputfile.txt
HSP71_YEAST
blast output fileЛучшее выравнивание выявлено в скаффолде 199, 79% идентичности, 920 bit score, что может говорить о наличии гомолога в этом фрагменте. Следующие фрагменты имеют bit score меньше, поэтому, вероятно это близкие родственники белка, но не его функциональные аналоги.
TERT_SCHPO
blast output fileЛучшее выравнивание выявлено в скаффолде 17, 25% идентичности, 108 bit score, что может говорить о наличии гомолога в этом фрагменте. Вероятно, это не гомолог белка. Также об этом говорит около 60% mismatch.
TBB_NEUCR
blast output fileЛучшее выравнивание выявлено в unplaced-665, 81.5% идентичности, 742 bit score, что может говорить о наличии гомолога в этом фрагменте. Следующие фрагменты в выравнивании имеют bit score меньше, поэтому, вероятно это близкие родственники белка, но не его функциональные аналоги.
Ниже приведено лучшее выравнивание. Отчетливо видно, что большая часть выравнивания не содержит гэпов и мало мисматчей.
unplaced-665
Length=20473
Score = 742 bits (1915), Expect = 0.0, Method: Compositional matrix adjust.
Identities = 367/450 (82%), Positives = 398/450 (88%), Gaps = 22/450 (5%)
Frame = -2
Query 1 MREIVHLQTGQCGNQIGAAFWQTISGEHGLDASGVYNGTSELQLERMN------------ 48
MREIVHL TG CGN IGA FW+ IS EHG+D +G Y G S+LQLER+N
Sbjct 7236 MREIVHL*TG*CGN*IGAKFWEVISDEHGIDPNGRYEGDSDLQLERINGEFLNVLFCA** 7057
Query 49 ----------VYFNEASGNKYVPRAVLVDLEPGTMDAVRAGPFGQLFRPDNFVFGQSGAG 98
VYFNEASG KYVPRAVLVDLEPGTMD+VRAGP+G LFRPDNF+FGQSGAG
Sbjct 7056 AFCITLLLIVVYFNEASGGKYVPRAVLVDLEPGTMDSVRAGPYGNLFRPDNFIFGQSGAG 6877
Query 99 NNWAKGHYTEGAELVDQVLDVVRREAEGCDCLQGFQITHSlgggtgagmgtllISKIREE 158
NNWAKGHYTEGAELVD VLDVVR+EAEGCDCLQGFQITHSLGGGTGAGMGTLLISKIREE
Sbjct 6876 NNWAKGHYTEGAELVDSVLDVVRKEAEGCDCLQGFQITHSLGGGTGAGMGTLLISKIREE 6697
Query 159 FPDRMMATFSVVPSPKVSDTVVEPYNATLSVHQLVENSDETFCIDNEALYDICMRTLKLS 218
+PDRMM TFSVVPSPKVSDTVVEPYNATLSVHQLVENSDETFCIDNEALYDIC RTLKL+
Sbjct 6696 YPDRMMCTFSVVPSPKVSDTVVEPYNATLSVHQLVENSDETFCIDNEALYDICFRTLKLT 6517
Query 219 NPSYGDLNHLVSAVMSGVTVSLRFPGQLNSDLRKLAVNMVPFPRLHFFMVGFAPLTSRGA 278
P+YGDLNHLVSAVMSGVT S+RFPGQLN+DLRKLAVNMVPFPRLHFFMVGFAPLTSRG+
Sbjct 6516 TPTYGDLNHLVSAVMSGVTTSIRFPGQLNADLRKLAVNMVPFPRLHFFMVGFAPLTSRGS 6337
Query 279 HHFRAVSVPELTQQMFDPKNMMAASDFRNGRYLTCSAIFRGKVSMKEVEDQMRNVQNKNS 338
+RA+SV ELT QMFD KNMMAASD R+GRYL +AIFRGK+SMKEV++QM +VQ KNS
Sbjct 6336 QQYRALSVAELTTQMFDAKNMMAASDPRHGRYLAVAAIFRGKMSMKEVDEQMLSVQTKNS 6157
Query 339 SYFVEWIPNNVQTALCSIPPRGLKMSSTFVGNSTAIQELFKRIGEQFTAMFRRKAFLHWY 398
SYFVEWIPNNV+TA+C IPP+GLKMS+TF+GNSTAIQELFKRI +QF+ MF+RKAFLHWY
Sbjct 6156 SYFVEWIPNNVKTAVCDIPPKGLKMSATFIGNSTAIQELFKRISDQFSVMFKRKAFLHWY 5977
Query 399 TGEGMDEMEFTEAESNMNDLVSEYQQYQDA 428
TGEGMDEMEFTEAESNMNDLVSEYQQYQDA
Sbjct 5976 TGEGMDEMEFTEAESNMNDLVSEYQQYQDA 5887
Поиск генов в скаффолде 497 генома Amoeboaphelidium protococcarum
С помощью infoseq был выбран scaffold-497 длинны 70081. С помощью seqret получена последовательность. Поиск генов проводился в RefSeq proteins (Fungi), алгоритмом blastx на сайте ncbi.
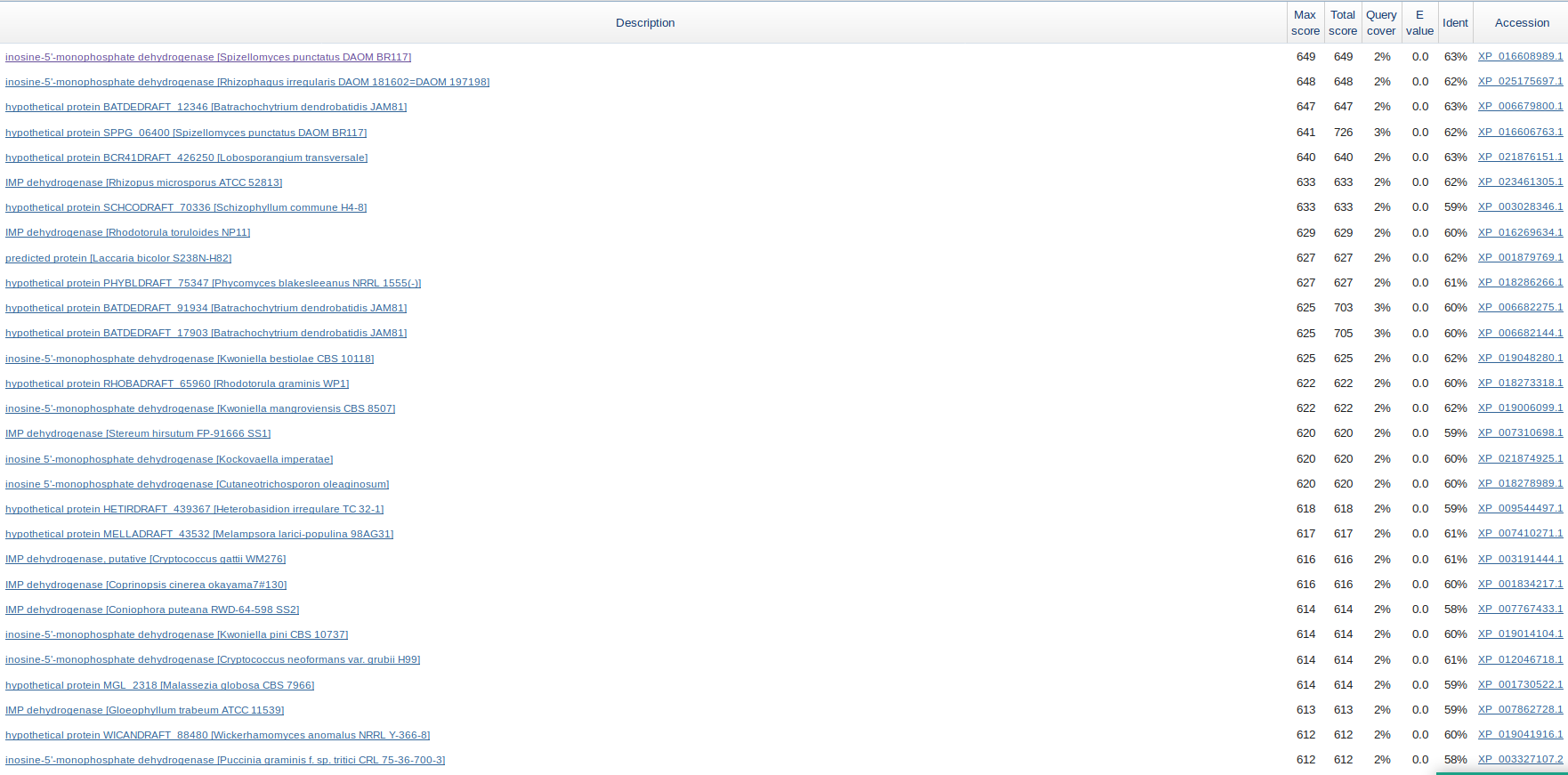
Большинство белков в выдаче являются дегидрогеназами ИМФ. Рассмотрим лучшее выравнивание:
Score Expect Method Identities Positives Gaps Frame
649 bits(1675) 0.0 Compositional matrix adjust. 325/513(63%) 402/513(78%) 11/513(2%) +3
Query 36081 DGLSAKELFDSTKFGGLTYNDFLMLPGYIDFPAQAVSLESHLTRKIKLKTPFVSSPMDTV 36260
DGL+A+EL + GGLTYNDFL+LPGY+DFPA SLES +T++ LKTPF+SSPMDTV
Sbjct 54 DGLTAEELMNPQLSGGLTYNDFLILPGYVDFPASICSLESRITKRFTLKTPFLSSPMDTV 113
Query 36261 TEAEMAIAMALMGGVGIVHYNCSVEDQCAMIRRVKKYENGFISDPVVLGPTNTVADVLQI 36440
TE +MAI MAL GG+G++H+NC VE+Q M+R+VKK+ENGFI+DP LGP +TV DV I
Sbjct 114 TETQMAIHMALNGGLGVIHHNCPVEEQADMVRKVKKFENGFITDPKCLGPEHTVQDVFNI 173
Query 36441 KNQYGFCGIPITESGKIGSKLIGIVTRRDIDFMQDTATQQAT*LVNVMTPLDQLVTAPQH 36620
K QYGFCGIPITE+GK+ SKL+GIVT RDIDF+Q +Q L +VMT LVTAPQ
Sbjct 174 KQQYGFCGIPITENGKLHSKLLGIVTSRDIDFLQTLEDRQRL-LKDVMT--TDLVTAPQG 230
Query 36621 VTLSKANEILKGSKKGKLPIVNDRGELISLVSRKDLLKKRDYPQSSKSYRQTLRNEENNQ 36800
V+L +AN ILK SKKGKLPIV+ +G L +L++R DL+K RDYP +SK + Q
Sbjct 231 VSLFEANTILKASKKGKLPIVDGQGRLTALLARSDLVKARDYPWASKM-------PSSKQ 283
Query 36801 LLVGAAIGTRPDDKVRLESLYKAGVDVIVLDSSQGNSKW*IEMIQHIRQQYKDDI*VIAG 36980
LLV AAI T +D+VRL++L AG+D++VLDSSQGNS + IEMI++I+Q + I VIAG
Sbjct 284 LLVAAAISTHDEDRVRLQALVDAGLDIVVLDSSQGNSSFQIEMIKYIKQTHPH-IDVIAG 342
Query 36981 NVVTKA*AKNLIDAGADALRVGMGSGSICITQEVMACGRPQGTAVYQVA*YCHSRGIPVI 37160
NVVT+ A+ LI+AGADALRVGMGSGSICITQEVMACGRPQGTAV++V+ + G+PVI
Sbjct 343 NVVTQEQARRLIEAGADALRVGMGSGSICITQEVMACGRPQGTAVFRVSQFARQFGVPVI 402
Query 37161 ADGGISNCGHIIKALSLGASCVMMGSLLAATTESPSEYFYQEGKKLKRYRGMGSISAMEQ 37340
ADGGI N GHI+KAL+LGAS VMMGSLLA T+ESP EY+Y EG++LK+YRGMGSI AME+
Sbjct 403 ADGGIGNVGHIVKALALGASAVMMGSLLAGTSESPGEYYYHEGQRLKKYRGMGSIDAMEK 462
ble
Query 37341 GSAASKRYYSD*QVLKVAQGVSGAIEDKGSVM*FMPYLIAGV*QGLQDIGTDSVKKLQDE 37520
G AA KRY+S+ +KVAQGV+GA+ DKGSV F+ YL GV LQDIGT ++ L+D
Sbjct 463 GDAAGKRYFSEADKIKVAQGVAGAVVDKGSVKKFLGYLATGVQHSLQDIGTRTLVDLRDA 522
Query 37521 VRAGSVRFELRSPSSQVEGGVHGLVSYEKRLFA 37619
V G VRFE R+ S+Q+EGGVHGL SYEKRLF+
Sbjct 523 VVNGEVRFEKRTASAQLEGGVHGLHSYEKRLFS 555
Бласт показывает границы гена 36081 - 37619. В этом гене нет интронов.
Карта локального сходства геномов Pseudomonas putida и Pseudomonas monteilii
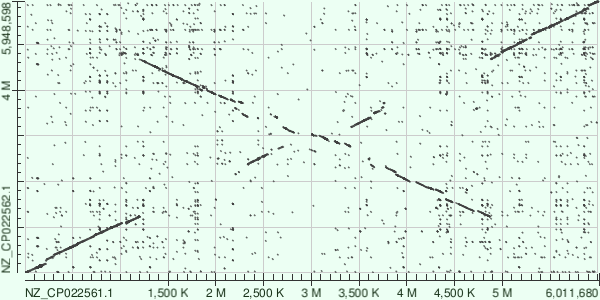
Два генома бактерий действительно похожи, хотя и имеются значительные отличия. Наиболее заметным является инверсия в центре генома. Также имеются небольшие инсерции/делеции (участки без соответствия в другом геноме) по всей длинне последовательностей.