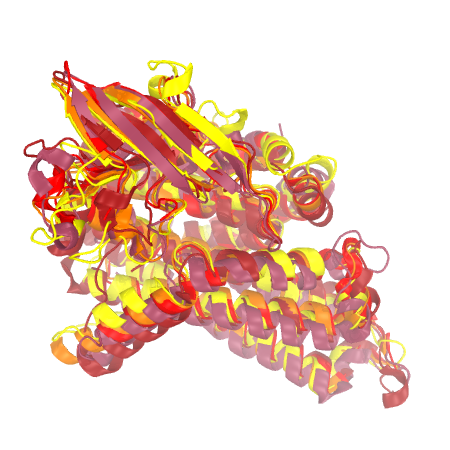
Для совмещения структур были выбраны следующие гомологи: 2VIG (1.04 Å; 86%), 2C12 (1.87 Å; 81%), 4NXL (2.03 Å; 81%), 4X28 (2.28 Å; 76%). PS: в скобках указаны значения RMSD и N-align (50-90%). Далее, в PDBeFold (отвратительно) и PyMol (красоты ради) данные структуры были выравнены. Выдача PDBeFold.
Рисунок 1. Совмещение структур гомологов

С помощью алгоритма множественного выравнивания Muscle в JalView было построено выравнивание гомологов:
Теперь давайте же рассмотрим, где наши выравнивания различаются. Для этого участки, хорошо выравненные в PDBeFold, были размечены на выравнивании, выполненном в JalView, чтобы легко можно было увидеть несоответствия (см. JalView_project). Так как PDBeFOLD выравнивает вторичные структуры, которые могут быть очень похожими, несмотря на различие в последовательностях, то именно структурное выравнивание будем считать более верным (учитывая, что структуры уже известны благодаря РСА). Недаром при огромном количестве последовательностей существует вполне определенное число фолдов. Рассмотрим пару примеров...
Рисунок 3. Пример несоответствия 1
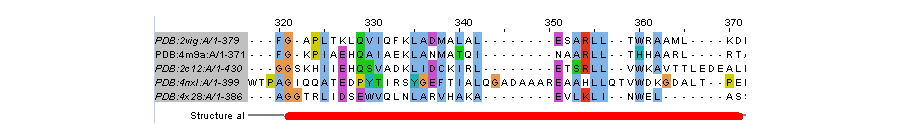
Здесь показано выравнивание, полученное в JalView, где красной полосой показан участок с хорошим структурным выравниванием. Согласно структуре 4m9a, в данном месте расположена α-спираль. В этом же районе расположены и спирали гомологов. Вывод - структура не врет, так как одни и те же элементы вторичной структуры могут быть собраны из разных остатков.
Рисунок 4. Пример несоответствия 2
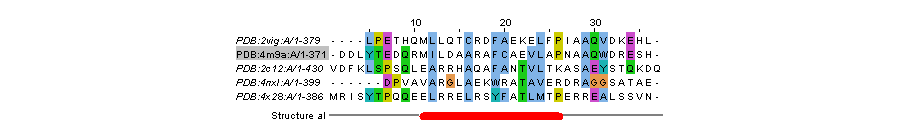
Здесь структуры не выравнены в самом начале (из-за "колебаний" концевых учасков при кристаллизации), далее расположена α-спираль. Ситуация немного иная: невыравненными в структурном выравнивании считаются концы α-спиралей (около 5 первых остатков спиралей, отмечены маленькими буквами); произошло это, скорее всего, из-за достаточного их пространственного расхождения относительно друг друга в составе третичной структуры.
Для поиска была выбрана структура 1cii:A (284-384). Был найден только 1 структурный гомолог. И это не сама структура. Однако при изменении параметра Lowest acceptable match до 30% структура 1cii обнаруживается в выдаче. Дело в том, что этот параметр характеризует долю элементов вторичной структуры целевой цепи (то, что мы ищем) в той структуре, что мы подаем на вход. При этом обратная характеристика (то,что ищем/ то,к чему ищем) должна быть равна 100%.
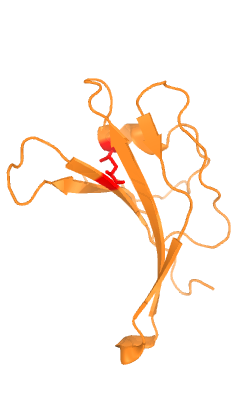
Были сохранены PDB структуры константного домена T-клеточного рецептора из цепочки альфа (2bnq region d:115-204) и из цепочки бета (1qrn region e:119-246). С помощью SheeP были получены карты β-листов для обеих структур. В PyMol отмечено положение консервативного Cys, образующего дисульфидную связь.
Рисунок 6. Домены из альфа-(A) и бета-цепочек(B) с картами β-листов


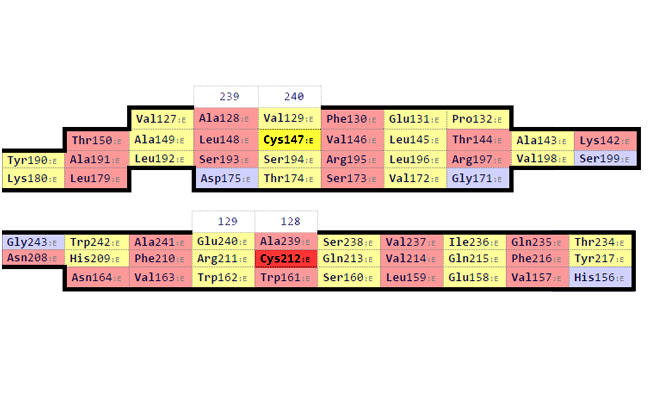
На изображении ниже показаны карты соответствующих друг другу листов и полученное в PyMol выравнивание участков ---Val-Cys-Leu--- (см. скрипт).
Рисунок 7. Карты "аналогичных" листов и их выравнивание

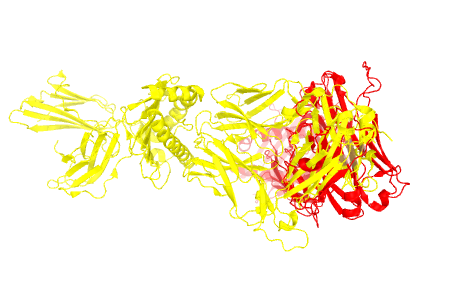

На рисунке выше выравнена левая часть показанного отдельно домена. Стрелками отмечены "конечные" участки домена: видно, что они выходят примерно в одинаковые стороны - сверху и снизу. Совпадают также общий ход цепи в пространстве и положения петель (как и их число), однако, как и ожидалось, β-тяжи выравнены только частично.
