Дана структура 2puc, для нее были получены несколько изображений (Рис1.1). Так как в файле содержался только мономер белка, то его пришлось достраивать до димера с помощью команды symexp в PyMol.
Рисунок 1. Области контактов белок-белок и ДНК-белок

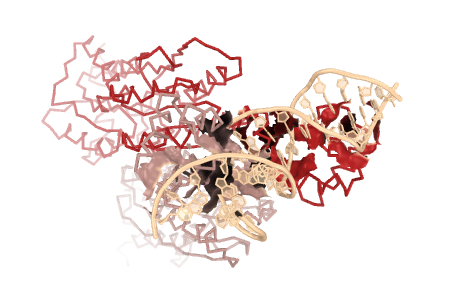
A. Поверхность контакта между мономерами. Белок показан как ribbon. Субъединицы выделены разными цветами.
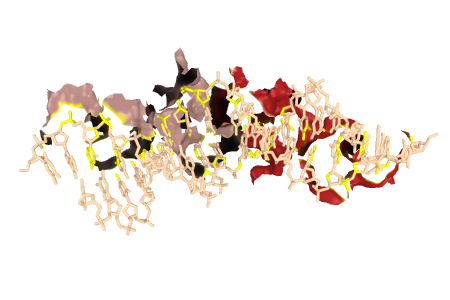
B. Поверхности контакта между ДНК и гомодимером. Белок показан как ribbon.
C. Поверхность контакта ДНК с гомодимером. Модель димера скрыта, ДНК отображена как sticks, взаимодействующие атомы выделены желтым.
B. Поверхности контакта между ДНК и гомодимером. Белок показан как ribbon.
C. Поверхность контакта ДНК с гомодимером. Модель димера скрыта, ДНК отображена как sticks, взаимодействующие атомы выделены желтым.
В CluD были найдены гидрофобные ядра размера не менее 10 атомов (Distance threshold = 5.0), которые расположены ближе всего к области контакта мономеров в составе димера. На рис.2 атомы ядер показаны сферами. Для демонстрации поверхностей в PyMol из Clud были сохранены списки входящих в состав ядер атомов. Затем выбирались нужные ядра, и атомы, входящие в их состав, подавались в PyMol для построения участка гидрофобной поверхности. См. пример скрипта, использованного для выделения нужных атомов.
Рисунок 2. Гидрофобные ядра
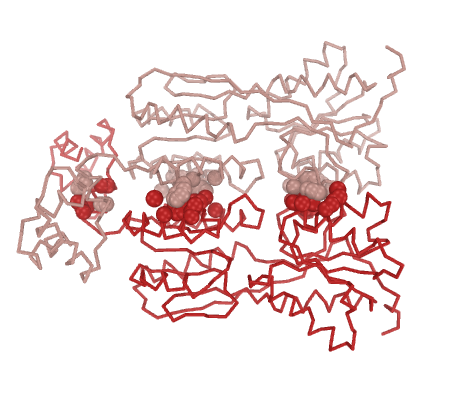
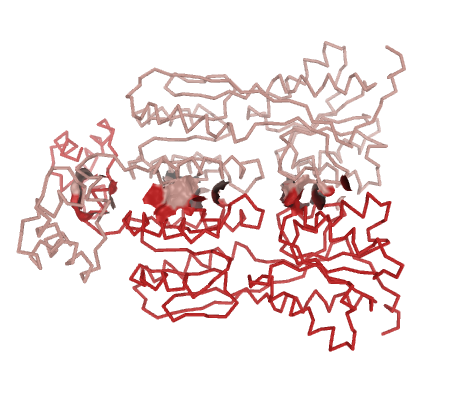
A. Положение гидрофобных ядер на участках взаимодействия белков.
B. Изображение гидрофобной поверхности ядер из Ри.2А.
B. Изображение гидрофобной поверхности ядер из Ри.2А.
PS: такую штуку можно поставить на поток с помощью R, чтобы не колдовать вручную в Notepad++; тогда не будет никакой необходимости в Jmol и в еще одном браузере.