Для работы была выбрана структура Acyl-CоА дегидрогеназы (PDB ID: 4M9A, разрешение - 2.20 Å). Было проверено, что структура соответствует следующим критериям:
1. Сервис EDS (Electrone Density Server) содержит описание структуры, что говорит о том, что экспериментальные данные также помещены в PDB (файл структурных факторов, Structure Factors).
2. Сервис PDBeFold посредством струткурных выравниваний находит 269 гомологов среди доступных структур. Приняв ограничения на RMSD (оценка сходства) от 0.7 Å до 3.0 Å, на N-align - 50-90%, получаем несколько гомологов из разных организмов, среди которых присутствуют следующие структуры: 2VIG (1.04 Å; 86%), 4KTO (1.19 Å; 90%), 3MPJ (1.23 Å; 81%), 1UKW (1.54 Å; 90%), 2C12 (1.87 Å; 81%), 4NXL (2.03 Å; 81%), 4X28 (2.28 Å; 76%) и т.д..
GCPII - Acyl-CoA дегидрогеназа из Burkholderia thailandensis (Грам-отрицательная бактерия), осуществляющая катализ начального шага реакции β-окисления жирных кислот в митохондриях.
Карта электронной плотности была загружена с сервиса EDS. Визуализация всех структур и манипуляции с ними производились с помощью PyMol и Photoshop.
Рисунок 1. Изображение электронной плотности вокруг полипептидной цепи белка 4M9A (анимация).
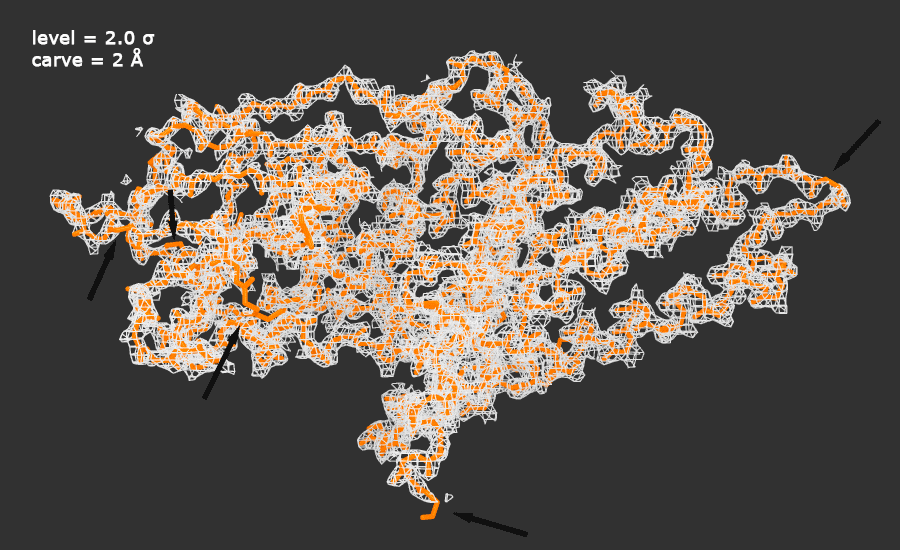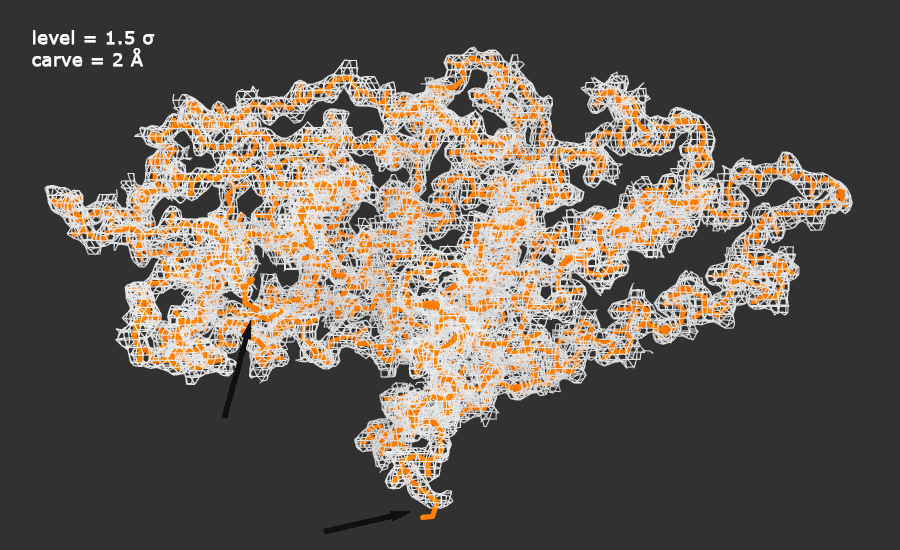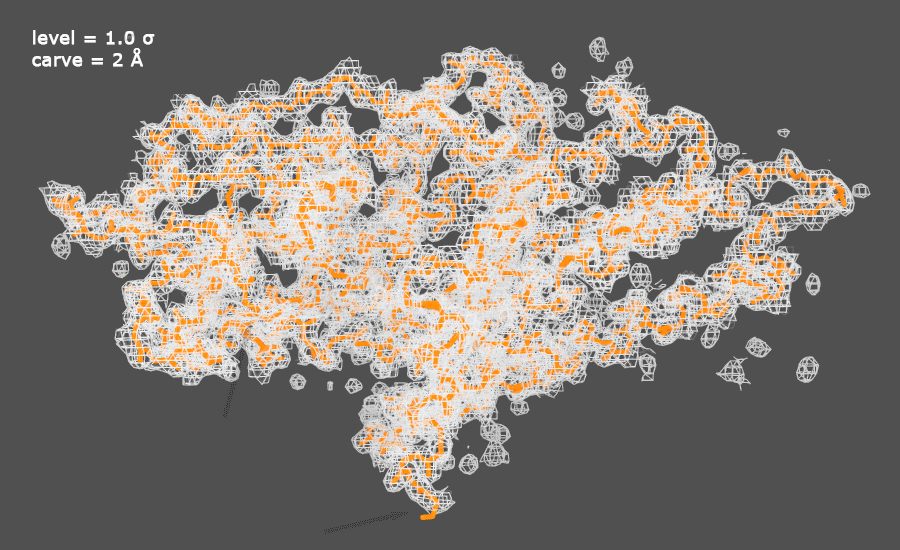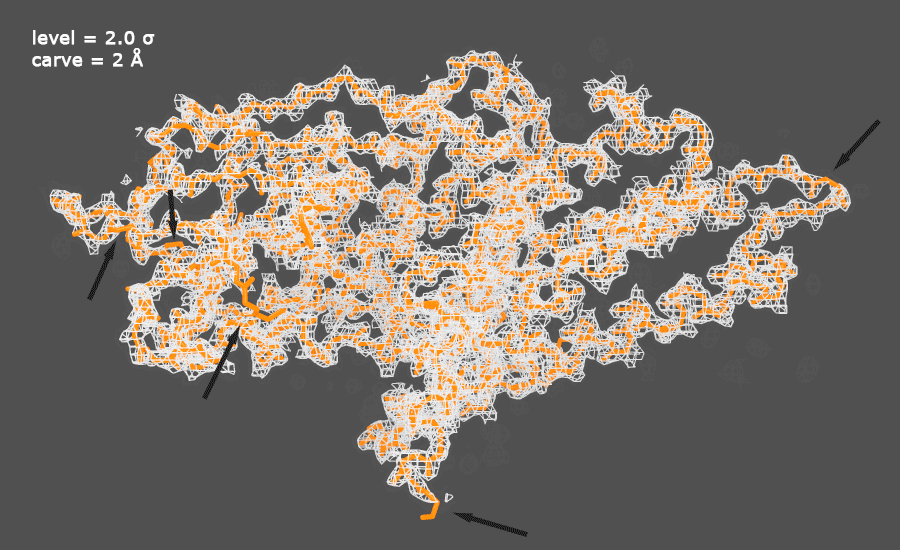
Белок выделен оранжевым цветом, карта электронной плотности - серым.
Карта электронной плотности во всех трех случаях была получена с помощью команд:
Исходный код
Карта электронной плотности во всех трех случаях была получена с помощью команд:
isomesh new_map, 4m9a_map, 1.0, chain A, carve = 2
isomesh new_map, 4m9a_map, 1.5, chain A, carve = 2
isomesh new_map, 4m9a_map, 2.0, chain A, carve = 2
color orange, chain A
color grey90, new_map
Исходный код
На изображении электронной плотности вокруг скелета стрелками выделены некоторые участки, где главная цепь не покрыта электронной плотностью.
Выбранные значения level определяются как количество электронов на ų. Параметр carve позволяет скрыть прочие секции карты плотности.
Для того, чтобы поближе познакомиться со сгущениями электронной плотности, были выбраны несколько наборов аминоксилот. На рисунках ниже представлена электронная плотность вокруг выбранных аминокислотных остатков для уровней электронной плотности 1.0 σ, 1.5 σ и 2.0 σ на указанном расстоянии от выбранных множеств (параметр carve = 2 Å).
Рисунок 2. Изображение ЭП вокруг участка Arg(375)-Gln(376)-Leu(377).
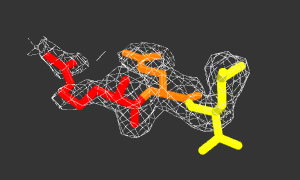
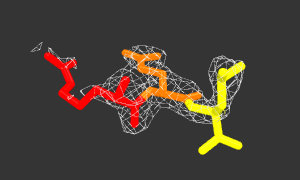
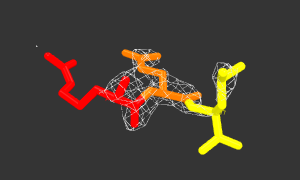
Arg(375) - красный, Gln(376) - оранжевый, Leu(377) - желтый.
Пример скрипта:
Исходный код
Пример скрипта:
isomesh new_map, 4m9a_map, 1.0, resi 375-377, carve = 2
isomesh new_map, 4m9a_map, 1.5, resi 375-377, carve = 2
isomesh new_map, 4m9a_map, 2.0, resi 375-377, carve = 2
color red, resi 375
color orange, resi 376
color yellow, resi 377
color gray90, new_map
Исходный код
↑На рисунке 2 видно, что карта электронной плотности вокруг бокового радикала Arg(375) прерывна.
Рисунок 3. Изображение ЭП вокруг участка Pro(311)-Cys(312)-Leu(313).
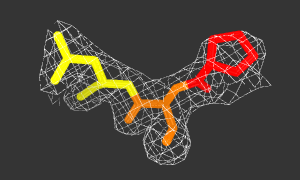
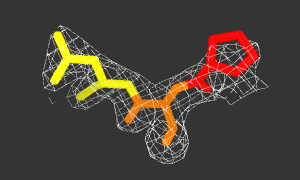
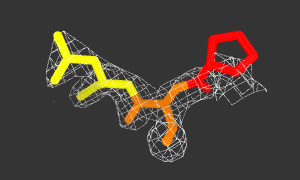
Pro(311) - красный, Cys(312) - оранжевый, Leu(313) - желтый. Исходный код
Рисунок 4. Изображение ЭП вокруг участка Gln(57)-Trp(58).
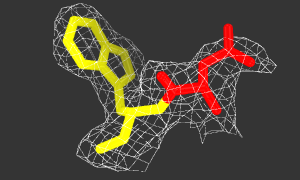
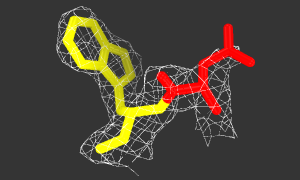
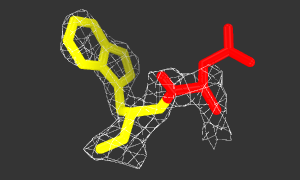
Gln(57) - красный, Trp(58) - желтый. Исходный код
≡
Исходя из изображения электронной плотности как целого белка, так и отдельных аминокислот, можно сделать вывод о том, что разрешения структуры 4M9A недостаточно для точного определения положения отдельных атомов, однако полипептидная цепь и положение большинства радикалов аминокислотных остатков уже достаточно хорошо видны для восстановления трехмерной структуры белка с учетом его последовательности.