Файл PDB имеет запись о кристаллографических характеристиках в поле CRYST1, которое для структуры 4M9A выглядит следующим образом:
CRYST1 87.260 111.000 170.250 90.00 90.00 90.00 P 21 21 21 16
Здесь первые три значения являются длинами направляющих векторов кристалла, следующие три - углами между ними, далее идут название кристаллографической группы (P 21 21 21) и число молекул в ячейке (16).
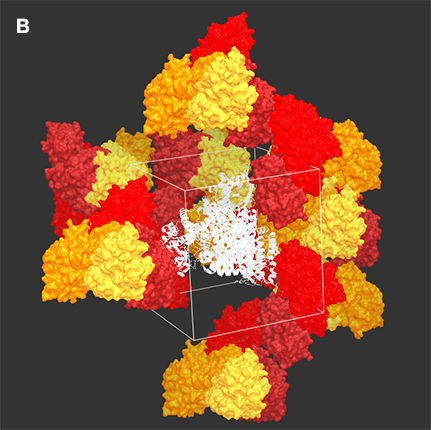
Белок является гомотетрамером, однако иначе называется "димером димеров", так как пространство, образуемое мономерами при взаимодействии, служит местом связывания FAD. С помощью команды symexp в PyMol были восстановлены соседние ячейки кристалла (Рис.1(B)).
Рисунок 1. Структура 4M9A и изображение соседних ячеек.
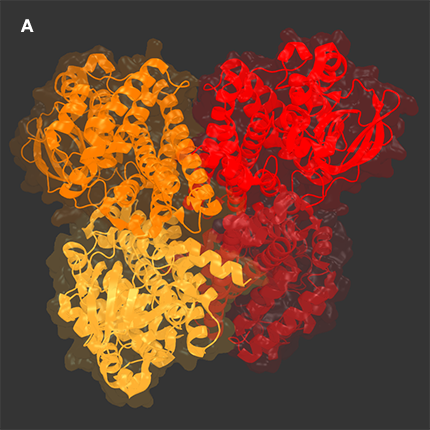
A. Гомотетрамер внутри одной ячейки. Различными цветами обозначены цепи.
B. Изображение молекул соседних ячеек. Структура белка в исходной ячейке и границы ячейки показаны белым. Для удобства часть молекул скрыта.
B. Изображение молекул соседних ячеек. Структура белка в исходной ячейке и границы ячейки показаны белым. Для удобства часть молекул скрыта.
На изображениях ниже представлены контакты исходной молекулы белка с молекулами соседних ячеек. Для расстановки водородных связей в PyMol использовались следующие команды:
show sticks, byres (set1n and (4m9aset around 3.5))
distance hbonds_1,set1n ,4m9aset , 3.5, 2
Исходный код,
На рис. 2(А) видно, что количество взаимодействующих остатков крайне мало (с учетом скрытых молекул).
Рисунок 2. Демонстрация межмолекулярных взаимодействий
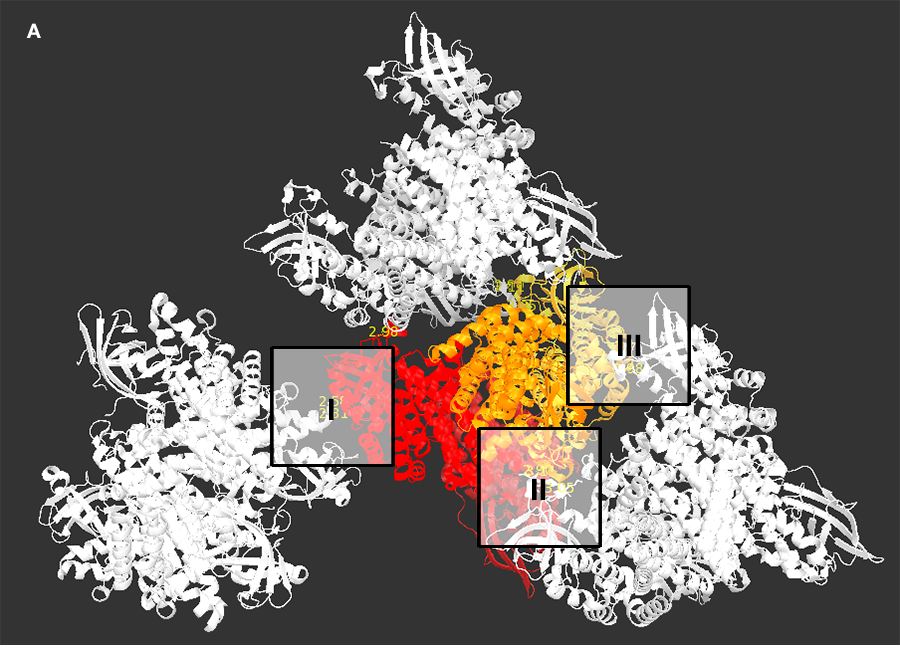
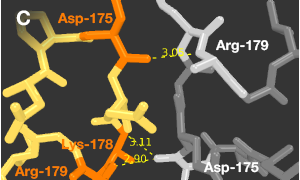
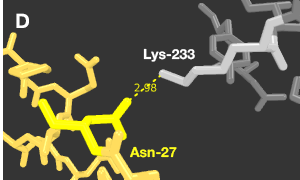
A. Выбранные для анализа области. Соседние молекулы белка выделены белым цветом.
B. Водородные связи в области I.
C. Водородные связи в области II.
D. Водородные связи в области III.
A-D: учавствующие во взаимодействиях аминокислотные остатки выделены более ярко.
B. Водородные связи в области I.
C. Водородные связи в области II.
D. Водородные связи в области III.
A-D: учавствующие во взаимодействиях аминокислотные остатки выделены более ярко.
Во взаимодействиях белков видна симметрия. Так, существуют, например, варианты:
Asn(27)·····Lys(233); Asn(27)·····Lys(233)
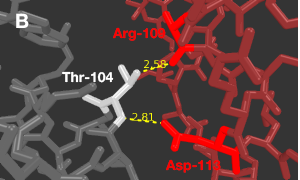
Arg(109)·····Thr(104)·····Asp(113); Arg(109)·····Thr(104)·····Asp(113)
Arg(109)·····Thr(104)·····Asp(113); Arg(109)·····Thr(104)·····Asp(113)
Поиск гидрофильных взаимодействий практически не дал результатов, следовательно, можно приступить к следующему этапу - поиску гидрофобных взаимодействий.
Рисунок 3. Расположение гидрофобных участков
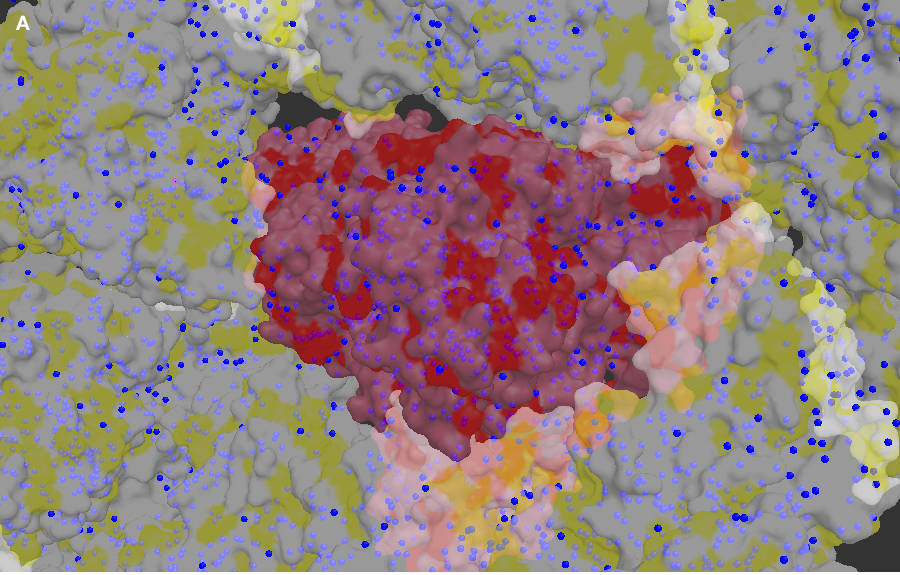

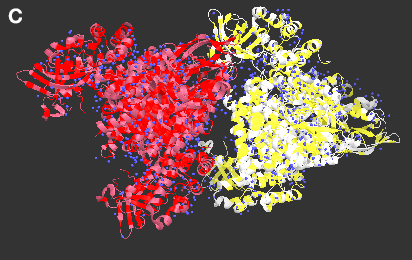
A-C: вид на распределение гидрофобных аминокислотных остатков с различных ракурсов. Гидрофобные остатки выделены ярким красным (исходный белок) и желтым (построенные симметрично белки), синим покрашены все молекулы воды.
Анализ гидрофобных и гидрофильных взаимодействий позволяет получить более полное представление о структуре и сделать вывод о том, что эти взаимодействия едва ли участвуют в олигомеризации молекул белка в организме ввиду их немногочисленности. Действующей формой фермента является гомотетрамер, расположенный в элементарной ячейке кристалла, и дальнейшей олигомеризации, скорее всего, не происходит.
3HDD является структурой комплекса гомеодомена engrailed у Drosophila и ДНК с разрешением 2.2 Å (гомеодомен - это ДНК-связывающий домен транскрипционных факторов, которым и является продукт гена engrailed), численные характеристики которой были рассчитаны на основе другой структуры с разрешением 2.8 Å. Изображени комплекса (рис.4) было получено в PyMol.
С помощью команды symexp были построены молекулы ДНК, находящиеся на расстоянии не более 3.5 Å от цепи B гомеодомена. Кроме того, были предсказаны водородные связи между цепью B и молекулами ДНК, находящимися как в исходной, так и в соседних ячейках кристалла.
Рисунок 4. Изображение комплекса ДНК-гомеодомен и анализ ДНК-белковых взаимодействий.
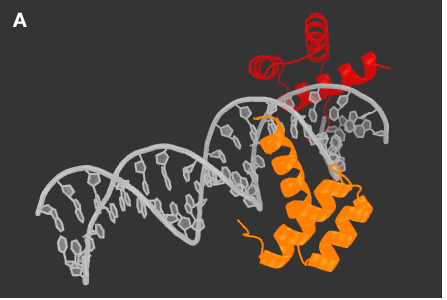
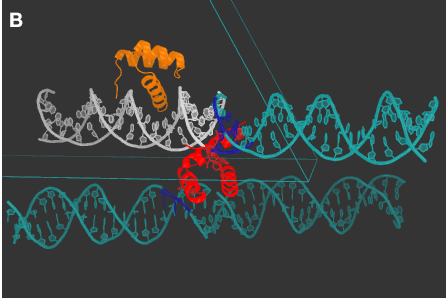
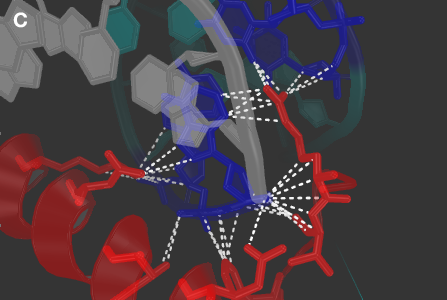
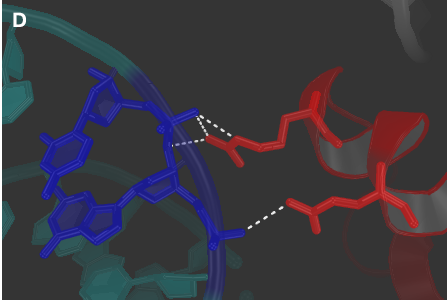
Исходные ДНК выделена белым цветом, цепь A белка - оранжевым, цепь B - красным. Построенные функцией symexp молекулы ДНК выделены бирюзовым.
A. Комплекс ДНК-гомеодомен.
B. Восстановление соседних с цепью B белка молекул ДНК из соседних ячеек кристалла.
C-D: предсказанные PyMol водородные связи между цепью B белка и молекулами ДНК. Участвующие в формировании связей нуклеотиды выделены синим (показаны как sticks), аминокислотные остатки - красным (показаны как sticks).
A. Комплекс ДНК-гомеодомен.
B. Восстановление соседних с цепью B белка молекул ДНК из соседних ячеек кристалла.
C-D: предсказанные PyMol водородные связи между цепью B белка и молекулами ДНК. Участвующие в формировании связей нуклеотиды выделены синим (показаны как sticks), аминокислотные остатки - красным (показаны как sticks).
На примере структуры 3HDD можно сделать вывод о том, что иногда содержимого одной ячейки кристалла недостаточно для анализа структуры. Так, объяснение странности расположения цепи B на краю двойной спирали ДНК стало возможным только после визуализации содержимого соседних ячеек.