Для выполнения практикума был выбран домен - PF00351: домен Биоптерин-зависимой гидроксилазы ароматических аминокислот (К этому семейству относится фенилаланин-4-гидроксилаза, белок, вызывающий фенилкетонурию.)
Выравнивание seed содержало 49 последовательностей. Окрасив выравнивание (Clustal, Above identity threshold) показался мотив [244-250] - GAGLLSS.
Полученный мотив загнали в Scan Prosite, на выходе получили 38 находок в 38 последовательностях. Провел выравнивание mafft и мотив наблюдался практически во всех последовательностях. Мотив неразрывался, однако в одной последовательности он не выровнился с основной массой, и в одной его разделило.
На основе полученного ранее выравнивания построил дерево и выбрал в нем кладу:

У данной клады нашел хороший мотив [204-210] - KLATCYF. И проведя поиск, этот мотив был найден только в этой кладе.

Для этого задания был выбран AC: O05886, - требуется для димеризации активных 70S-рибосом в 100S-рибосомы в стационарной фазе; 100S-рибосомы трансляционно неактивны и иногда присутствуют во время экспоненциального роста. Выделен из Mycobacterium tuberculosis.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 20 | P17161.1 | 0.003 | P17160.1 | 0.006 |
| 2 | 27 | P71346.3 | 1e-11 | - | - |
| 3 | 29 | P06727.4 | 0.003 | P33621.1 | 0.007 |
| 4 | 38 | P02651.2 | 2e-04 | O46409.1 | 0.007 |
| 5 | 46 | P32918.2 | 0.004 | O42296.1 | 0.008 |
| 6 | 134 | P0DUP8.1 | 0.005 | A0A6P6DKR7.1 | 0.005 |
У меня не получились сходящиеся результаты, с каждой следующей итерацией находок становилось все больше и разница между лучшей и худшей находкой постоянно менялась. Поэтому попробую уменьшить порог e-value до 0.001:
| Номер итерации | Число находок выше порога (0,001) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 17 | P28613.2 | 6e-04 | P26983.1 | 0.001 |
| 2 | 27 | P71346.3 | 5e-11 | - | - |
| 3 | 27 | P24694.1 | 2e-23 | P06727.4 | 0.002 |
На сей раз новых находок больше не становилось, а разница между лучшей и худшей находками была более явной. Выходит, что получилось найти гомологичную группу.
Выбрал 71 белок, с доменной архитектурой: PF21417 - PF00351 - PF00351. Для анализа запущен поиск МЕМЕ:
Было найдено 4 мотива, среди них нет расмотренных в других заданиях. Далее запущен FIMO:
Данные мотивы найдены 385 раз в 62 последовательностях.
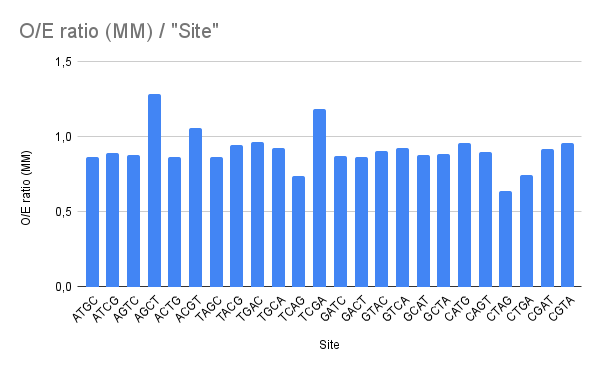
Провел оценку контрастов obs/exp в геноме, воспользовавшись командой и файлом с сайтами длины 4, образованными перестановками A, T, G, C:
По итогу получили файл с оценкой встречаемости всех сайтов, на его основе построили диаграмму: