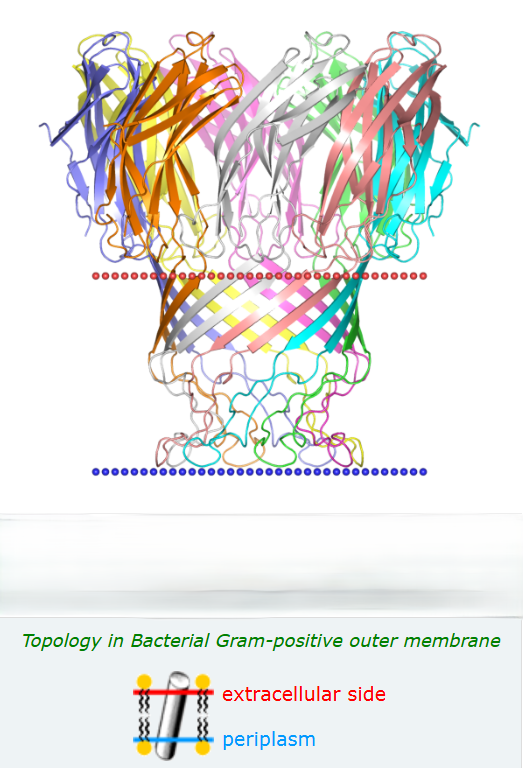
В базе данных OPM выбрал белок, содержащий β-листы: Porin MspA (Порин MspA | pdb: 1uun | uniprot: MSPA_MYCS2). В его функции входит проведение катионов, ионов Fe3+, аминокислот, фосфатов. Играет важную роль в транспортировке бета-лактамаз и гидрофильных фторхинолоновых антибиотиков, таких как норфлоксацин и хлорамфеникол.
Таблица 1 Информация по белку
| Type | Transmembrane |
| Class | Beta-barrel transmembrane |
| Superfamily | Mycobacterial Porin (MBP) |
| Family | Mycobacterial Porin (MBP) |
| Species | Mycolicibacterium smegmatis |
| Locallization | Bacterial Gram-positive outer membrane |


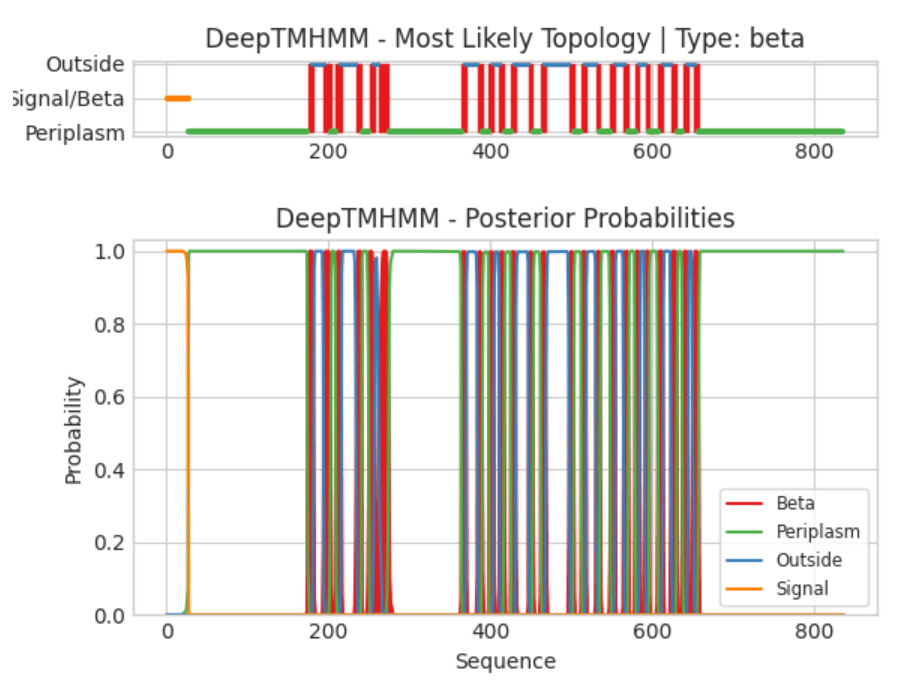
Порин представлен восемью идентичными субъединицами [Рис.1], следующий этап - предсказание трансмембранных участков. Для этого из uniprot получили последовательность белка и, используя сервис DeepTMHMM, получили результаты:[Рис.2|Доп.1]
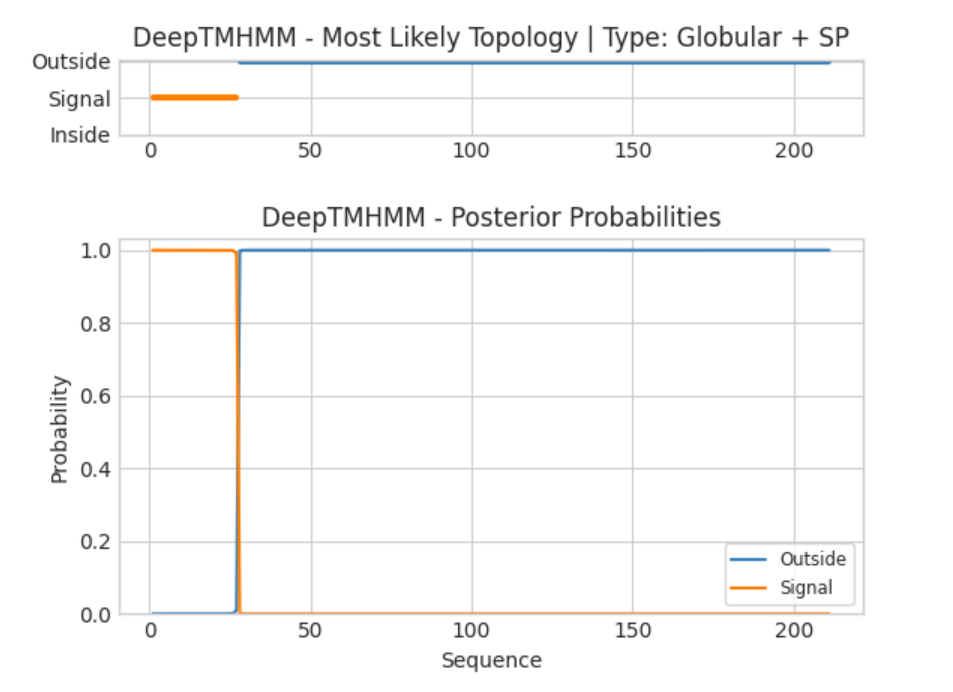
Как можем видеть результаты далеки от истины, так как данный пример иллюстрирует недостаток сервиса. Мы проанализировали последовательность одной субъединицы, а наш порин представлен комплексом из доменов и предсказание становится невозможным, раз так, придется взять другой белок и не забывать эту особенность сервиса.
Запасным вариантом был P pilus tip assembly PapC-PapD-PapK-PapG, structure 1 (pdb:7lhg, uniprot: PAPC_ECOLX)
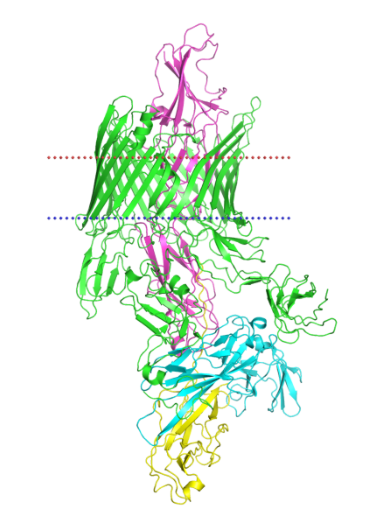
Таблица 2 Описание белка
| Type | Transmembrane |
| Class | Beta-barrel transmembrane |
| Superfamily | Fimbrial usher porin |
| Family | Fimbrial usher porin |
| Species | Escherichia coli |
| Locallization | Bacterial Gram-negative outer membrane |
Этот белок является более целостной системой [Рис.4], поэтому прошлая неудача не должна повториться. Скачал последовательность с сайта Uniprot. Сейчас оценим предсказание DeepTMHMM [Рис.5|Доп.2]:
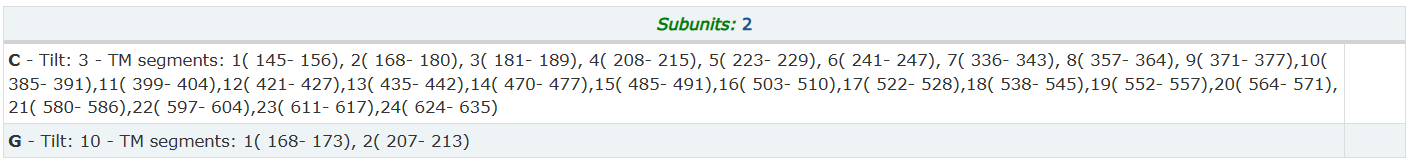
Мы - не мы, если не сравним, на сколько полученные результаты сопоставимы с другим предсказанием, так что сейчас и сравним [Таблица 3|Доп.3]. Но рассматривая предсказание DeepTMHMM, не забудем о наличии сигнального пептида (длиной 27 аминокислот). Его длину необходимо будет вычесть из предсказания трансмембранных участков
Таблица 3 Сопоставление предсказаний OPM и DeepTMHMM
| OPM | DeepTMHMM |
| 1( 145- 156), | 1(148-154) |
| 2( 168- 180), | 2(168-176) |
| 3( 181- 189), | 3(183-189) |
| 4( 208- 215), | 4(208-214) |
| 5( 223- 229), | 5(224-230) |
| 6( 241- 247), | 6(236-247) |
| 7( 336- 343), | 7(338-343) |
| 8( 357- 364), | 8(358-363) |
| 9( 371- 377), | 9(372-377) |
| 10( 385- 391), | 10(385-390) |
| 11( 399- 404), | 11(399-404) |
| 12( 421- 427), | 12(421-425) |
| 13( 435- 442), | 13(436-441) |
| 14( 470- 477), | 14(471-476) |
| 15( 485- 491), | 15(486-491) |
| 16( 503- 510), | 16(504-508) |
| 17( 522- 528), | 17(522-527) |
| 18( 538- 545), | 18(538-543) |
| 19( 552- 557), | 19(553-557) |
| 20( 564- 571), | 20(565-569) |
| 21( 580- 586), | 21(581-586) |
| 22( 597- 604), | 22(597-603) |
| 23( 611- 617), | 23(611-617) |
| 24( 624- 635) | 24(625-630) |
После удаления длины сигнального пептида из предсказания, участки стали очень похожи, некоторые даже совпадали полностью [OPM; DeepTMHMM] - {11 (399 - 404)}. Для визуализации результата БЕЗ удаленного пептида приведу таблицу с перекрытиями [Доп.3]:
Для этого задания расматриваться будет белок: Bacteriorhodopsin-I (Бактериородопсин I, pdb: 4pxk, uniprot: BACR1_HALMA, последовательность, функционал - управляемый светом протонный насос [Рис.6|Таблица 4])
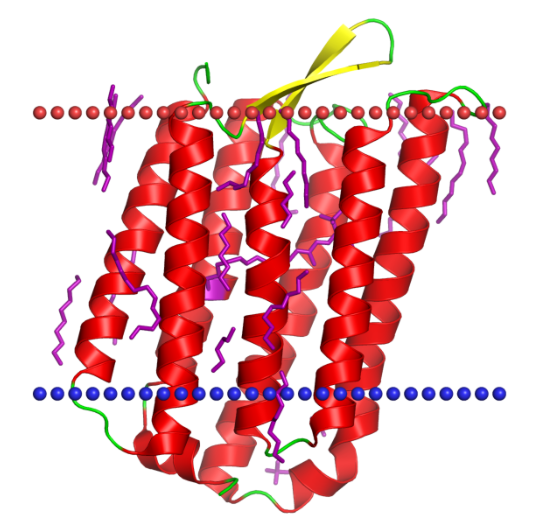
Таблица 4 Описание белка
| Type | Transmembrane |
| Class | Alpha-helical polytopic |
| Superfamily | Rhodopsin-like receptors and pumps |
| Family | Microbial and algal rhodopsins |
| Species | Haloarcula marismortui |
| Locallization | Archaebacterial membrane |
Данный белок представлен одной субъединицей, поэтому сейчас результаты, предположительно, должны быть максимально схожи у обоих предсказаний, в чем сейчас постараемся убедиться:
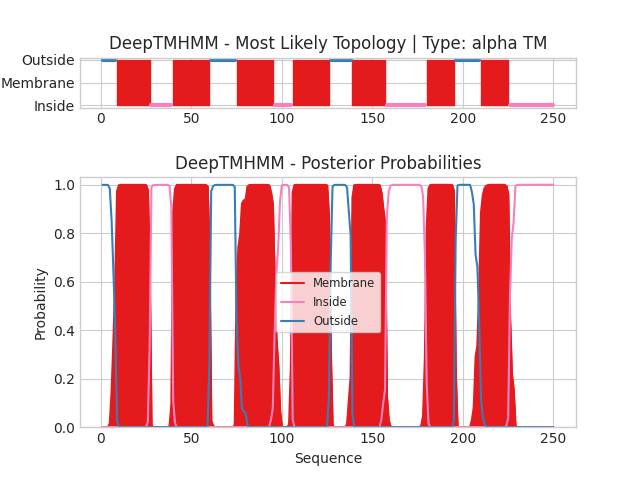
Предсказание выглядит обнадеживающе, все трансмембранные участки четко видны и принимают практически абсолютные значения на всей длине [Рис 7|Доп 4]. Теперь момент истины сравнить предсказания:
Таблица 5 Сравнение предсказаний
| OPM | DeepTMHMM |
| 1(7-29) | 1(9-27) |
| 2(39-60) | 2(40-60) |
| 3(78-96) | 3(75-95) |
| 4(106-125) | 4(106-126) |
| 5(135-157) | 5(139-157) |
| 6(177-199) | 6(180-195) |
| 7(205-219) | 7(210-225) |
На сей раз в нашем белке только одна субъединица и результаты можно начать сравнивать. До пятого отрезка отличия не превышают пять аминокислотных остатков, однако на последних двух отрезках степень различия резко возрастает. Модель DeepTMHMM по большей степени вложа в представление OPM. В заключение, DeepTMHMM - хорший сервис для предсказания трансмембранных участков, однако необходимо знать с чем работаешь, ибо сервис не настроен на предсказание комплексов, представленных более чем одной субъединицей.