Мини-обзор генома бактерии Streptococcus canis
Введение
Streptococcus canis — патогенная факультативно анаэробная грамположительная бактерия. По современным филогенетическим данным таксономия данного организма представлена в таблице 1 [1] :
Таблица 1. Систематическое положение исследуемой бактерии Streptococcus canis.
| Домен | Bacteria |
| Филлум | Bacillota |
| Класс | Bacilli |
| Порядок | Lactobacillales |
| Семейство | Streptococcaceae |
| Род | Streptococcus |
| Вид | Streptococcus canis |
Для рода Streptococcus также характерно множество видов, которые являются патогенными для человека или других групп организмов. Например, Streptococcus pyogenes вызывает ангину, скарлатину, ревматизм, гломерулонефрит [10]. Streptococcus pneumoniae является возбудителем пневмонии, менингита, отита [11]. Также для клеток рода Streptococcus характерна шаровидная, или коккоидная форма. Множество клеток соединяются друг с другом, образуя длинные цепочки, которые называются стрептококками.
S. canis в норме находится в ротовой полости человека, а также на коже псовых и кошачьих. Однако в определенных ситуациях эта бактерия может вызывать инфекции кожных покровов и мягких тканей. Кошки и собаки являются основным звеном передачи инфекции человеку. Клинические и микробиологические данные показали, что S. canis в основном участвует в инфекциях мочевыводящих путей, целлюлите, эндокардите, артрите, а также очень редко в пневмонии [6]. Возможно бессимптомное носительство бактерии.
За последние два десятилетия сообщения об опасных для жизни случаях заболеваний у людей, связанных с S. canis, а также постоянно растущая популярность собак и кошек в качестве домашних животных привлекли внимание к этому виду бактерии [2][3][4][5]. В ряде исследований изучался S. canis, но по сравнению с другими видами стрептококков работа над этим патогеном была ограничена, и многое до сих пор остается неясным.
Наиболее широко изученными факторами вирулентности S. canis являются М-подобный белок SCM, стрептолизин О, стрептолизин S, фибронектин связывающий белок (fibronectin binding protein) [6]. Также у S. canis существует особый биохимический путь — аргининдеиминазная система (ADS). ADS играет значимую роль в патогенности бактерий рода Streptococcus, обеспечивая им адаптацию к различным стрессовым условиям, таких как кислотный стресс (например, в фаголизосоме), недостаток питательных веществ и атаки иммунной системы хозяина. Ее функции тесно связаны с метаболизмом аргинина и производством аммиака, который помогает стрептококкам выживать в организме хозяина [13].
ADS состоит из трех основных ферментов, расположенных на поверхности клетки S. canis, — аргининдеиминазы (ArcA), орнитинкарбамоилтрансферазы (ArcB) и карбамат киназы (ArcC), которые опосредуют реакции превращения аргинина с образованием углекислого газа, аммиака и АТФ [13]. Аргининдеиминазная система обеспечивает выработку энергии и поддерживает pH среды за счет выделения аммиака. Этот процесс особенно важен для выживания бактерий в кислой среде.
В этом мини-обзоре был подготовлен анализ генома бактерии S. canis: встречаемость старт-кодонов, GC-состав, изучение триплетного состава белок-кодирующих генов и генов белков ADS (аргининдеиминазы (ArcA), орнитин-карбамоилтрансферазы (ArcB), карбамат киназы (ArcC)).
Материалы и методы
Для изучения и анализирования генома бактерии были применены следующие платформы и инструменты:Б
1. Данные о геноме исследуемой бактерии, включая таблицы с особенностями, последовательностями и участками CDS, были извлечены из базы данных геномов NCBI. Впоследствии были загружены и обработаны в Google-таблицах следующие файлы (см. S6 в сопроводительных материалах):
1) GCF_010993845.2_ASM1099384v2_cds_from_genomic.fna — файл, содержащий белок-кодирующие последовательности исследуемой бактерии. Он был использован для анализа длины белков, закодированных в геноме, старт-кодонов, содержания GC-пар, триплетного состава белок-кодирующих последовательностей и белков ADS.
2) GCF_010993845.2_ASM1099384v2_genomic.fna — файл полного генома бактерии;
3) GCF_010993845.2_ASM1099384v2_feature_table.txt — таблица особенностей генома бактерии.
2. С помощью командной оболочки BASH был проведен анализ количества встреч различных старт-кодонов отдельно во всех CDS (белок-кодирующих последовательностях): псевдогенах и “нормальных” генах, а также суммарного их количества для каждого триплета. С помощью конвейеров (см. S1 сопроводительных материалов) было определено количество встреч каждого старт-кодона. Затем результаты анализа были импортированы в Google-таблицы, где на основе полученных данных была составлена таблица 2, отражающая частоту встречаемости старт-кодонов в геноме S. canis.
3. С использованием языка программирования Python была построена столбчатая диаграмма (рисунок 3), отражающая частотный анализ триплетов в белок-кодирующих последовательностях. Данные для анализа были получены из файла *cds_from_genomic.fna, содержащего все белок-кодирующие последовательности исследуемого организма. С помощью скрипта (см. S2 сопроводительных материалов) был рассчитан триплетный состав бактерии в процентном соотношении относительно всех кодонов. Псевдогены не учитывались.
Также с использованием Python был проведен триплетный и аминокислотный анализ трех главных белков аргининдеиминазной системы с помощью скрипта (см. S3 сопроводительных материалов). Далее для каждого кодона было определено его процентное содержание относительно всех кодонов в генах, кодирующих этот белок.
Результаты и обсуждение
Длины белков, закодированных в геноме Streptococcus canis
На рисунке 1 представлена гистограмма длин белков S. canis, построенная по данным из таблицы белок-кодирующих последовательностей. Видно, что белки с длиной от 70 до 310 АК встречаются гораздо чаще других. Их среднее значение — 278 АК. Значительные отклоняющиеся значения в данном интервале отсутствуют, и большинство белков имеют длину, близкую к среднему значению. По мере увеличения длины свыше 310 АК количество белков в интервале карманов постепенно убывает. Минимальная длина белка составляет 22 аминокислотных остатка, максимальная — 2918 АК. При составлении гистограммы было решено объединить в единый столбец белки, длина которых более 1090 АК, в связи с их относительной малочисленностью.
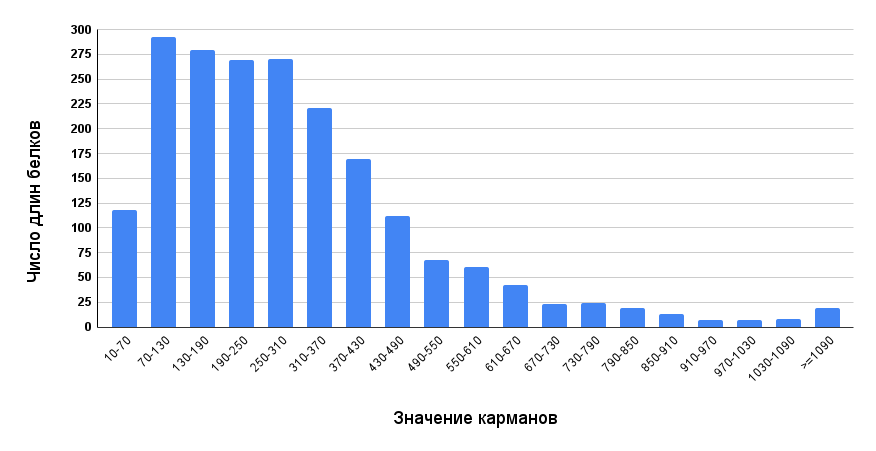
На рисунке 1 представлена гистограмма длин белков S. canis, построенная по данным из таблицы белок-кодирующих последовательностей. Видно, что белки с длиной от 70 до 310 АК встречаются гораздо чаще других. Их среднее значение — 278 АК. Значительные отклоняющиеся значения в данном интервале отсутствуют, и большинство белков имеют длину, близкую к среднему значению. По мере увеличения длины свыше 310 АК количество белков в интервале карманов постепенно убывает. Минимальная длина белка составляет 22 аминокислотных остатка, максимальная — 2918 АК. При составлении гистограммы было решено объединить в единый столбец белки, длина которых более 1090 АК, в связи с их относительной малочисленностью.
Из этой гистограммы можно сделать вывод, что абсолютное большинство белков имеют длину в диапазоне 70–310 АК. Это может быть связано с тем, что эта длина соответствует оптимальной длине для многих ферментов и транспортных белков. Самым большим белком является helicase-related protein (2918 АК), самым маленьким — peptide pheromone SHP2 (22 АК).
Интересное наблюдение заключается в том, что белки, которые содержат в своем составе более 900 АК, в основном участвуют в матричных и регуляторных процессах, а также представляют собой большие мульти субъединичные комплексы. Например, к ним относятся субъединицы ДНК-зависимой РНК-полимеразы, ДНК-зависимая ДНК-полимераза III, топоизомераза, сериновая протеаза, факторы инициации трансляции.
Встречаемость старт-кодонов в CDS
В геноме S. canis было проанализировано использование различных старт-кодонов в “нормальных” белок-кодирующих генах (Normal CDS), в псевдогенах (Pseudo CDS), а также их суммарное количество (All CDS). Результаты представлены в таблице 2.
Таблица 2. Встречаемость старт-кодонов в CDS исследуемой бактерии Streptococcus canis.
| Codons | All CDS | Pseudo CDS | Normal CDS |
|---|---|---|---|
| ATG | 1 775 | 56 | 1 719 |
| GTG | 102 | 3 | 99 |
| TTG | 86 | 3 | 83 |
| ATA | 9 | 3 | 6 |
| ATC | 4 | 4 | 0 |
| TCA | 3 | 3 | 0 |
| GTT | 3 | 3 | 0 |
| CAA | 3 | 3 | 0 |
| AAA | 2 | 2 | 0 |
| TTT | 2 | 2 | 0 |
| AGT | 2 | 2 | 0 |
| CTG | 2 | 1 | 1 |
| TAT | 2 | 2 | 0 |
| AAC | 1 | 1 | 0 |
| AGA | 1 | 1 | 0 |
| ACC | 1 | 1 | 0 |
| GAG | 1 | 1 | 0 |
| GAT | 1 | 1 | 0 |
| GGC | 1 | 1 | 0 |
| GTC | 1 | 1 | 0 |
| CAT | 1 | 1 | 0 |
| CCA | 1 | 1 | 0 |
| CCG | 1 | 1 | 0 |
| CCT | 1 | 1 | 0 |
| CTT | 1 | 1 | 0 |
| TAC | 1 | 1 | 0 |
| TTA | 1 | 1 | 0 |
В геноме S. canis было проанализировано использование различных старт-кодонов в “нормальных” белок-кодирующих генах (Normal CDS), в псевдогенах (Pseudo CDS), а также их суммарное количество (All CDS). Результаты представлены в таблице 2.
Как видно из полученных данных, старт-кодон ATG встречается наиболее часто как в белок-кодирующих генах, так и в псевдогенах. Он находится в 1775 генах (1719 в Normal CDS и 56 в Pseudo CDS). Наряду с ATG встречаются другие старт-кодоны, например, GTG (102 случая) и TTG (86 случаев). Однако они встречаются значительно реже и преимущественно наблюдаются в “нормальных” белок-кодирующих генах. Например, GTG встречается в 99 “нормальных” генах и в 3 псевдогенах, а TTG — в 83 “нормальных” генах и также в 3 псевдогенах. Остальные старт-кодоны встречаются крайне редко и преимущественно в псевдогенах.
Из данной таблицы можно сделать определенные выводы. ATG является наиболее оптимальным стартовым кодоном, который сохраняется в результате эволюции [7]. Его частое использование подтверждает ключевую роль кодона ATG в инициации трансляции у S. canis. Также стоит заметить, что самые часто встречающиеся старт-кодоны представляют собой ATG, но с точечной мутацией только в одном из положений, чаще всего в первом (GTG, TTG), что в целом специфично не только для исследуемой бактерий, но и для ряда других видов бактерий [7].
Единично встречающиеся старт-кодоны наблюдаются исключительно в псевдогенах (за исключением CTG), что, вероятно, связано с нарушением их функциональности и накоплением мутаций. Как видно из таблицы 2, псевдогены характеризуются более разнообразным и случайным распределением старт-кодонов, что указывает на потерю функциональной значимости этих последовательностей, так как на них не действует отбор. Поэтому им свойственно накапливать большое количество мутаций.
Также был составлен скрипт на Python (см. S4 сопроводительных материалов), который выводит названия белков, имеющие в своем гене исследуемый старт-кодон. Например, CTG является старт-кодоном гена белка fibronectin binding protein (FBP), являющегося одним из вирулентных белков S. canis [6]. Вероятнее всего, использование такого уникального старт-кодона связано с механизмом регуляции трансляции данного белка.
Гистограмма GC-пар в белок-кодирующих генах Streptococcus canis
На рисунке 2 изображена гистограмма содержания пар GC в различных белок-кодирующих генах. Минимальный процент пар GC составляет 23,11; максимальный — 50,41. Как видно на представленной гистограмме, большинство генов содержит от 39% до 41% пар GC, что является довольно среднестатистическим значением. Это значит, что бактерия не склонна к обитанию в экстремальных условиях, поэтому иметь большое количество пар GC в геноме не несет особого адаптивного смысла.
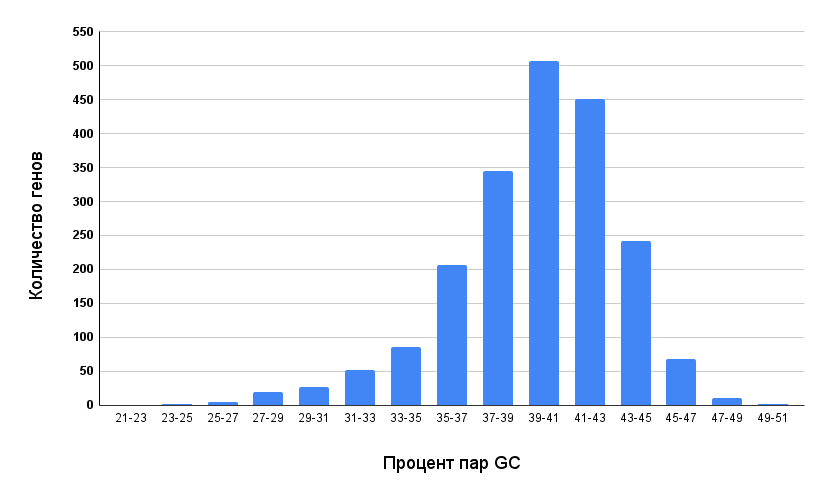
Известно, что стабильность двойной спирали ДНК относительно зависит от основного состава нуклеотидов. Выявлена линейная зависимость между содержанием GC в полимере и температурой его плавления [8]. Объяснение такой зависимости заключается в том, что пары AT и GC различаются стабильностью независимо от своих соседей. Связь АТ содержит две водородные связи, в то время как GC — 3. Тройная связь делает взаимодействие более сильным и устойчивым к высоким температурам. Это означает, что ДНК, содержащая большее количество пар GC, будет более стабильной и менее подвержена разрывам при нагревании [8].
Анализ кодонов в белок-кодирующих последовательностях Streptococcus canis
Был проведен анализ кодонного состава белок-кодирующих последовательностях в геноме исследуемой бактерии S. canis. Каждый кодон кодирует свою аминокислоту.
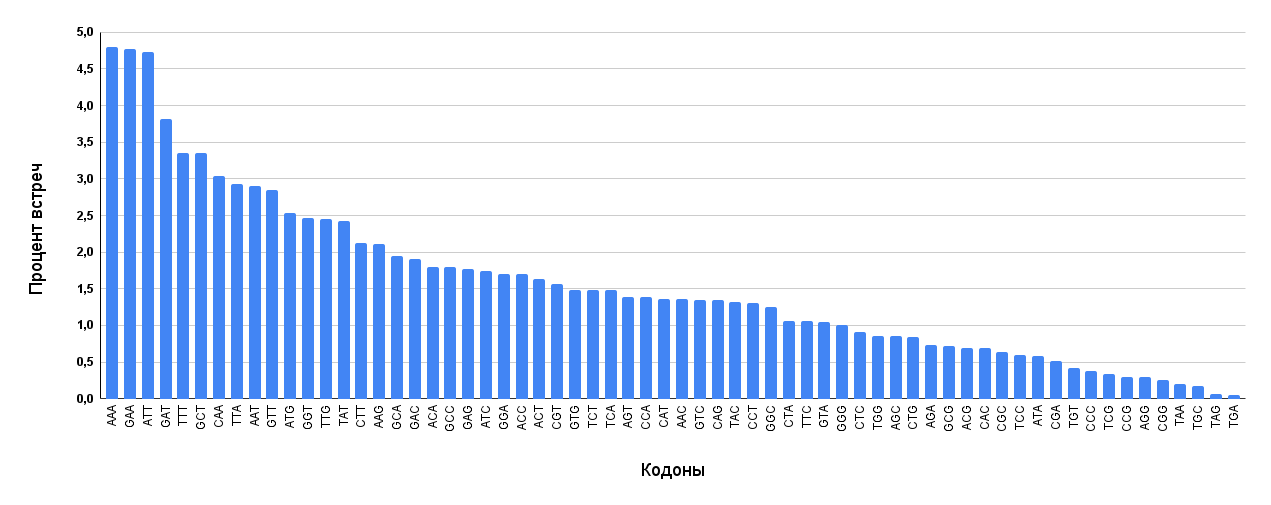
Был проведен анализ кодонного состава белок-кодирующих последовательностях в геноме исследуемой бактерии S. canis. Каждый кодон кодирует свою аминокислоту.
По рисунку 3 можно заметить, что наибольшую часть генома составляют кодоны AAA, GAA и ATT (более 4,5%), которые кодируют аминокислоты лизин (К), глутаминовая кислота (G), изолейцин (I) соответственно. Кодоны CAA, GAT, GCT, TTT, кодирующие аминокислоты глутамин (Q), аспарагиновая кислота (D), аланин (A), фенилаланин (F) соответственно (см. S5 сопроводительных материалов), встречаются реже (более 3,0%). Высокая частота кодонов, таких как AAA, ATT и GAC (около 4–5%), указывает на то, что они связаны с часто встречающимися аминокислотами в белках исследуемой бактерии. Кодоны с частотами более 3% (CAA, GAT, GCT, TTT) могут отражать аминокислоты, которые тоже широко используются, но реже, чем лидеры. Кодоны с частотами менее 0,5% свидетельствуют о редкой встречаемости соответствующих аминокислот, либо о возможных особенностях регуляции белкового синтеза, например, о том, что, возможно, такие кодоны используются для замедления трансляции. Невысокая встречаемость стоп-кодонов TAG, TGA, TAA (менее 0,25%) объяснима их функцией терминации трансляции.
Стоит заметить, что, например, глутаминовая кислота кодируется двумя кодонами: GAA и GAG. GAA является одним из самым часто встречающимся кодоном в геноме исследуемой бактерии (4,5%), однако количество GAG составляет лишь 1,75%, что является достаточно низким значением по сравнению со встречаемостью другого кодона. Лизин тоже кодируется двумя кодонами: AAA и AAG. ААА — самый часто встречающийся кодон в геноме S. canis (более 4,5%). AAG занимает около 2,25% от общего числа кодонов. Наблюдаемое распределение указывает на явное предпочтение определенным кодонам, характерное для большинства микроорганизмов [9]. Синонимичные кодоны используются не случайно и не одинаково, и для кодирования определенных аминокислот предпочтение отдается конкретным кодонам. Это может быть связано с адаптацией к числу соответствующих тРНК и особенностям эволюции вида [14]. В организме среди генов на предпочтение кодонов могут влиять уровень экспрессии и длина генов, структура мРНК, аминокислотный состав и гидрофобность кодируемого белка, а также различные этапы экспрессии белка [9].
Анализ кодонного состава генов белков аргининдеиминазной системы Streptococcus canis
Аргинидеиминаза — первый белок аргининдеиминазной системы. Она представляет собой фермент гидролазу, который превращает аргинин в цитруллин с образованием молекулы аммиака [16]. Аргининдеиминаза состоит из 411 аминокислотных остатков.
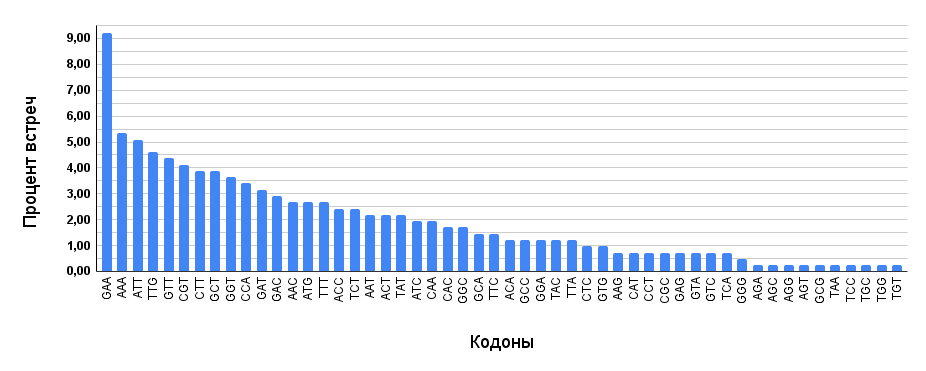
По полученным данным из диаграммы на рисунке 4, наиболее часто встречающимся кодоном оказался GAA (более 9%), кодирующий глутаминовую кислоту. Другие триплеты (AAA, ATT, GTT, TTG) встречались немного реже (более 3,75%). Они кодируют соответственно лизин, изолейцин, валин, лейцин (см. S5 сопроводительных материалов). Кодоны, кодирующие такие аминокислоты, как цистеин (C), триптофан (W), аланин (А), серин (S), аргинин (R) встречаются очень редко.
Как видно из диаграммы, соотношение встречаемости кодонов одних и тех же аминокислот указывает на кодонное предпочтение, характерное для S. canis, которое обсуждалось ранее. Первый кодон глутаминовой кислоты GAA является самым часто встречающимся (более 9%), второй кодон GAG встречается в очень малом количестве (менее 1%). Точно такая же ситуация с лизином.
Высокая частота кодонов глутаминовой кислоты указывает на доминирование заряженных остатков, что согласуется с функцией аргининдеиминазы. Она работает с аргинином — положительно заряженной аминокислотой при нейтральных значениях pH. Глутаминовая кислота — отрицательно заряженная при нейтральном pH, что, судя по всему, обеспечивает лучшее связывание аргинина в активном центре аргининдеиминазы.
Орнитин-карбамоилтрансфераза — второй по счету фермент в аргининдеиминазной системе. Он катализирует реакцию превращения цитруллина в карбамоилфосфат и орнитин [16]. В геноме S. canis закодированы две формы орнитин-карбамоилтрансферазы: одна состоит из 337 аминокислотных остатков, другая — из 332. Диаграмма была построена на основе этих двух ферментов.
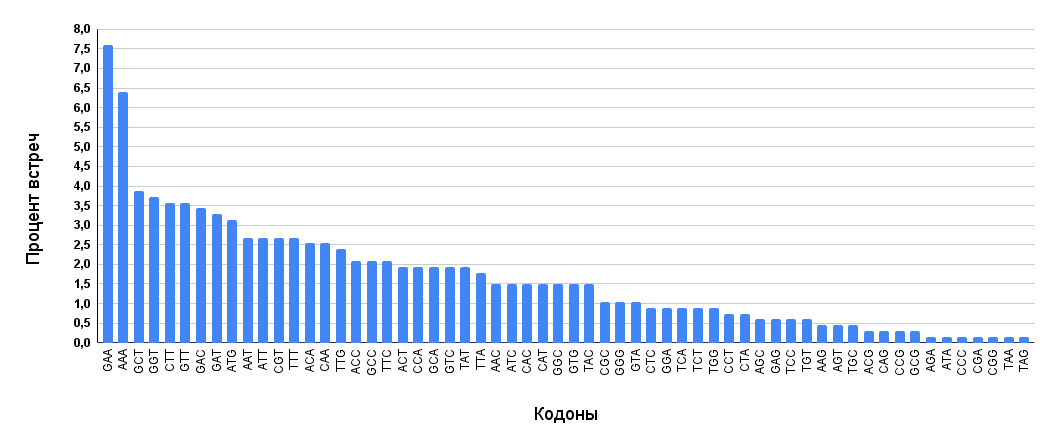
На рисунке 5 изображена диаграмма кодонов у орнитин-карбамоилтрансферазы. Так же, как у аргининдеиминазы, у орнитин-карбамоилтрансферазы наиболее часто встречающимся кодоном оказался GAA (более 9%), кодирующий глутаминовую кислоту. Однако орнитин-карбамоилтрансфераза отличилась высоким содержанием триплетов AAA, кодирующих лизин, относительно аргининдеиминазы (см. S5 сопроводительных материалов). Высокое содержание глутаминовой кислоты и лизина можно объяснить осуществляемой этим ферментом реакцией. Большое количество кодонов лизина связано с тем, что орнитин-карбамоилтрансфераза работает с карбамоилфосфатом, который является отрицательно заряженной молекулой при нейтральном pH. Глутаминовая кислота, судя по всему, участвует в стабилизации молекулы фермента.
Карбамат киназа — третий белок в аргининдеиминазной системе. Фермент катализирует превращение карбамоилфосфата и АДФ в АТФ и карбамат, который гидролизуется до аммиака и гидрокарбоната [16]. На рисунке 6 изображена диаграмма встречаемости кодонов у карбамат киназы. В геноме S. canis закодированы две формы карбамат киназы: одна состоит из 316 аминокислотных остатков, другая — из 310. Диаграмма была построена на основе этих двух ферментов.
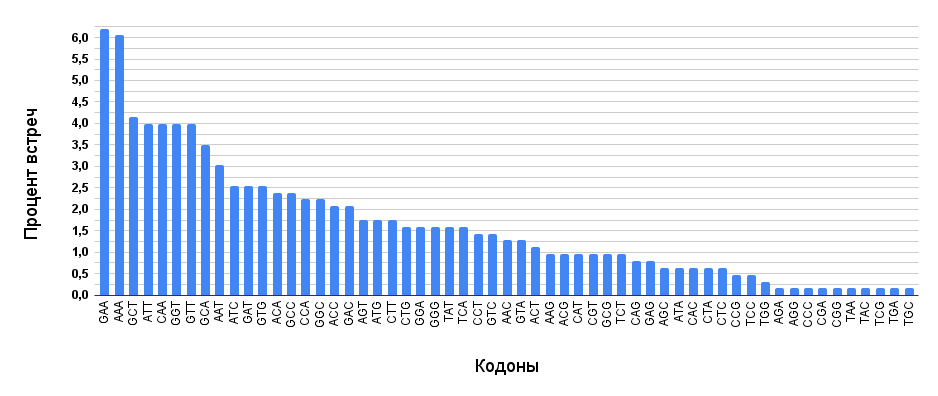
В случае карбамат киназы кодоны GCT (аланин, А), ATT (аспарагин, N), CAA (глутамин, Q) и GGT (глицин, G) находятся примерно на одном уровне по процентному содержанию в гене (4,0%). Это может быть важно для поддержания стабильного уровня синтеза карбамат киназы, поскольку равномерная частота кодонов может снижать вероятность задержек в трансляции. Аланин и глицин часто участвуют в формировании гибких участков или альфа-спиралей белков. Аспарагин и глутамин могут участвовать в образовании водородных связях, тем самым стабилизируя фермент [15].
У всех трех исследуемых белков ADS самый часто встречающийся кодон — это GAA, кодирующий глутаминовую кислоту. За ним идет кодон AAA, кодирующий лизин. При чем содержание лизина растет в следующем порядке: аргининдеиминаза, орнитин-карбамоилтрансфераза, карбамат киназа. Вероятнее всего это связано с тем, что последние два белка катализируют реакции, в ходе которых получаются или преобразуются отрицательно заряженные молекулы (орнитин-карбамоилтрансфераза работает с карбамоилфосфатом, карбамат киназа — с карбамоилфосфатом, АДФ и АТФ). Кодоны, кодирующие такие аминокислоты, как цистеин, триптофан, аланин, серин, аргинин, пролин встречаются очень редко во всех белках ADS.
Заключение
В ходе проведенной работы был проведен анализ генома патогенной бактерии Streptococcus canis. Была составлена гистограмма длин белков, таблица старт-кодонов, гистограмма содержания GC-пар, а также был сделан анализ кодонного состава всего протеома за исключением псевдогенов. В отдельности был разобран кодонный состав трех белков аргининдеиминазной системы, которая является одним из ключевых метаболических путей, влияющий на патогенность Streptococcus canis. Также было выявлено явное предпочтение кодонов одной и той же аминокислоты. Есть надежда, что результаты данного мини-исследования могут помочь с дальнейшим изучением данной бактерии.
Сопроводительные материалы
S1. Start codons. Конвейеры BASH для подсчета числа старт-кодонов CDS.
S2. Triplet count cds. Скрипт Python для подсчета встреч триплетов (в процентах) с необходимыми пояснениями.
S3. ADS proteins triplets. Скрипт Python для подсчета триплетов (в процентах) для трех белков ADS с необходимыми пояснениями.
S4. Gene name by start codon. Скрипт Python для вывода названий белков с интересующим старт-кодоном с необходимыми пояснениями.
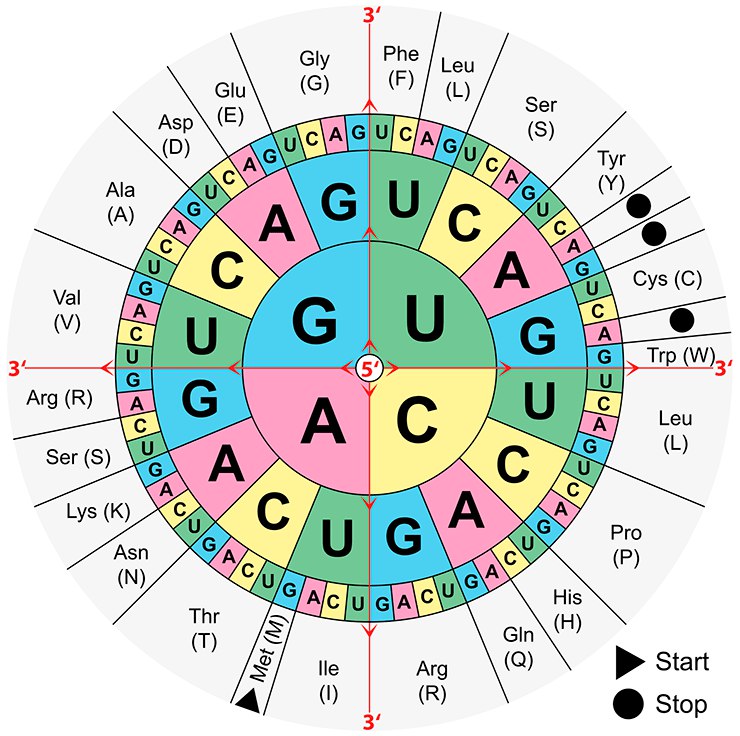
S5. Таблица генетического кода [12].
S6. СDS from genome of Streptococcus canis. Таблица CDS Streptococcus canis со всеми диаграммами, гистограммами и таблицами, которые были использованы при анализе генома Streptococcus canis.
Использованная литература
1. Schoch CL, et al. NCBI Taxonomy: a comprehensive update on curation, resources and tools. Database (Oxford). 2020: baaa062. PubMed: 32761142 PMC: PMC7408187.
2. Eibl C, Baumgartner M, Urbantke V, Sigmund M, Lichtmannsperger K et al. An outbreak of subclinical mastitis in a dairy herd caused by a novel Streptococcus canis sequence type (ST55). Animals 2021; 11:550.
3. Galpérine T, Cazorla C, Blanchard E, Boineau F, Ragnaud JM et al. Streptococcus canis infections in humans: retrospective study of 54 patients. J Infect 2007; 55:23–26.
4. Taniyama D, Abe Y, Sakai T, Kikuchi T, Takahashi T. Human case of bacteremia caused by Streptococcus canis sequence type 9 harboring the scm gene. IDCases 2017; 7:48–52.
5. Zaidi SMH, Eranki A. Streptococcus canis bacteremia in a renal transplant recipient. J Investig Med High Impact Case Rep 2019; 7:2324709619834592.
6. Davide Pagnossin, Andrew Smith, Katarina Oravcová, William Weir. Streptococcus canis, the underdog of the genus. Vet Microbiol. 2022 Oct:273:109524. doi: 10.1016/j.vetmic.2022.109524.
7. Xiongwen Cao, Sarah A Slavoff. Non-AUG start codons: Expanding and regulating the small and alternative ORFeome. Exp Cell Res. 2020 June 1;391(1):111973. doi: 10.1016/j.yexcr.2020.111973.
8. Peter Yakovchuk, Ekaterina Protozanova, Maxim D Frank-Kamenetskii. Base-stacking and base-pairing contributions into thermal stability of the DNA double helix. 2006 Jan 31;34(2):564-74. doi: 10.1093/nar/gkj454. Print 2006.
9. Sujatha Thankeswaran Parvathy, Varatharajalu Udayasuriyan, Vijaipal Bhadana. Codon usage bias. Springer. 2022 Jan;49(1):539-565. doi: 1007/s11033-021-06749-4.
10. Stephan Brouwer, Tania Rivera-Hernandez, Bodie F Curren, Nichaela Harbison-Price, David M P De Oliveira, Magnus G Jespersen, Mark R Davies, Mark J Walker. Pathogenesis, epidemiology and control of Group A Streptococcus infection. Nat Rev Microbiol. 2023 Jul;21(7):431-447. doi: 10.1038/s41579-023-00865-7.
11. Ditte Høyer Engholm, Mogens Kilian, David S Goodsell, Ebbe Sloth Andersen, Rikke Schmidt Kjærgaard. A visual review of the human pathogen Streptococcus pneumoniae. FEMS Microbiol Rev. 2017 Nov 1;41(6):854-879. doi: 10.1093/femsre/fux037.
12. Franziska Hufsky. Omeomics: die allumfassende Wissenschaft. Mai 18, 2016.
13. A Hitzmann, S Bergmann, M Rohde, G S Chhatwal, M Fulde. Identification and characterization of the arginine deiminase system of Streptococcus canis. Vet Microbiol. 2013 Feb 22;162(1):270-7. doi: 10.1016/j.vetmic.2012.08.004.
14. Parvathy ST, Udayasuriyan V, Bhadana V. Codon usage bias. Mol Biol Rep. 2022;49(1):539-565.
15. C N Pace. J M Scholtz. A helix propensity scale based on experimental studies of peptides and proteins. Biophys J. 1998 Jul;75(1):422–427. doi: 10.1016/s0006-3495(98)77529-0.
16.Silvio Hering, Antje Sieg, Bernd Kreikemeyer, Tomas Fiedler. Kinetic characterization of arginine deiminase and carbamate kinase from Streptococcus pyogenes M49. Protein Expr Purif. 2013 Sep;91(1):61-8. doi: 10.1016/j.pep.2013.07.002