Практикум 12. Семейства белковых доменов. PFAM
Описания семейства доменов
В предыдущем практикуме был выбран домен с PFAM AC PF10566, который является каталитической областью семейства бактериальных гликозилгидролаз.
| Параметр | Информация |
|---|---|
| AC pfam | PF10566 |
| ID pfam | Glycoside hydrolase 97 |
| #SEED | 44 |
| #All | 12000 |
| #SW | 3 |
| #architectures | 143 |
| #3D | 17 |
| Taxonomy | 12726 |
| #eukaryota | 59 |
| #archaea | 117 |
| #bacteria | 12378 |
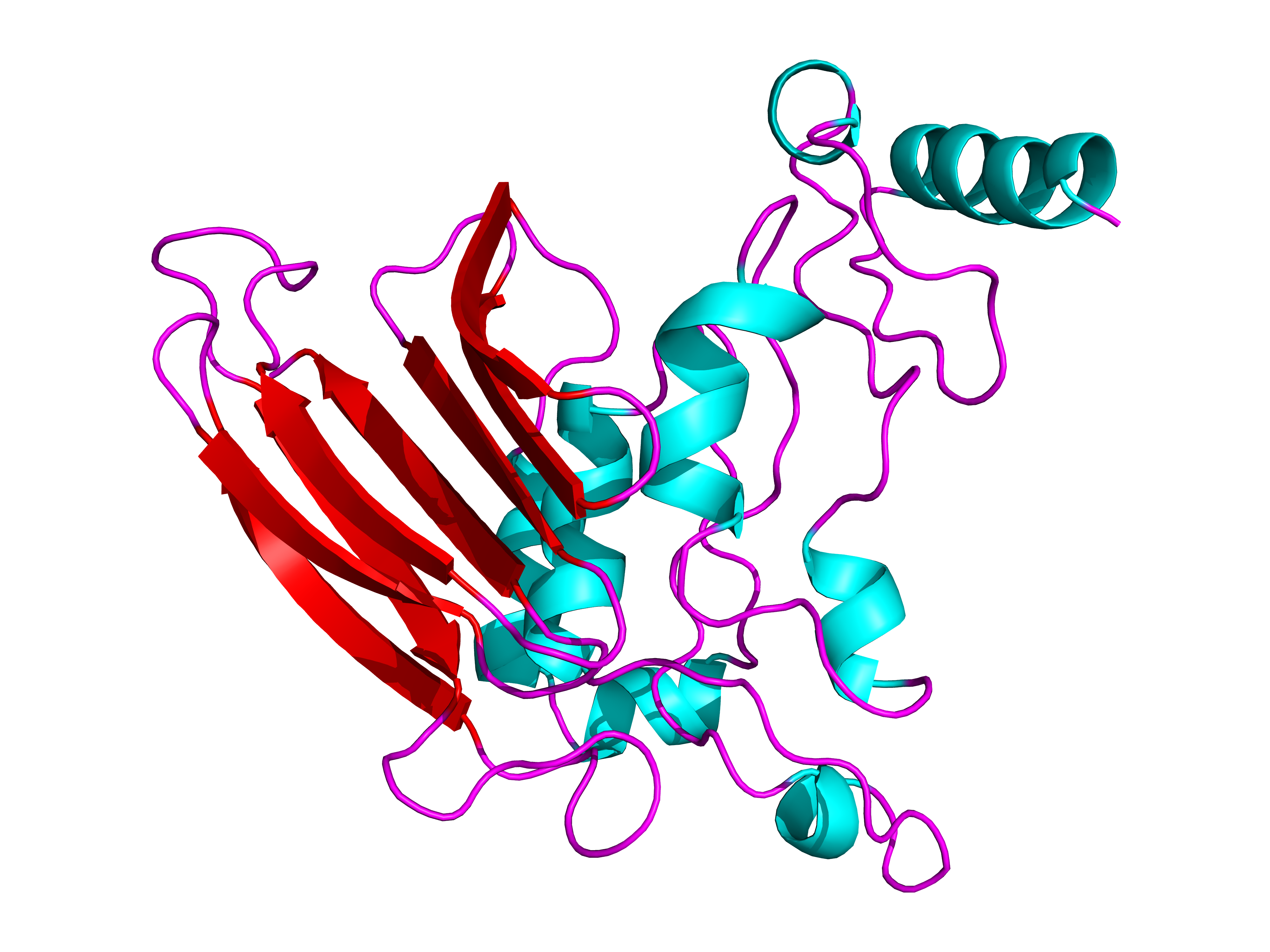
На рисунке 1 представлено строение исследуемого домена, который был предсказан в Alphafold. Как сказано в описании, каталитический домен обладает характерной (β/α)-структурой, которая является консервативной для данного семейства гликозидаз. Помимо центрального каталитического домена, эти белки содержат дополнительные структурные элементы: N- и C-концевые участки, преимущественно состоящие из β-цепей, формируют вспомогательные некаталитические домены, которые, вероятно, играют роль в стабилизации общей структуры белка. β-листы собраны в одну компактную бочонковидную структуру, α-спирали образуют отдельные кластеры.
Как видно из таблицы 1, данный домен широко распространен среди бактерий, но также очень редко встречается у архей и эукариот.
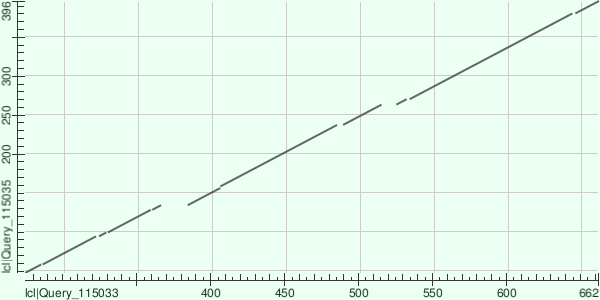
Карта локального сходства (dotplot) двух последовательностей с одним и тем же доменом, но с разной доменной архитектурой
| Информация | Protein name | |
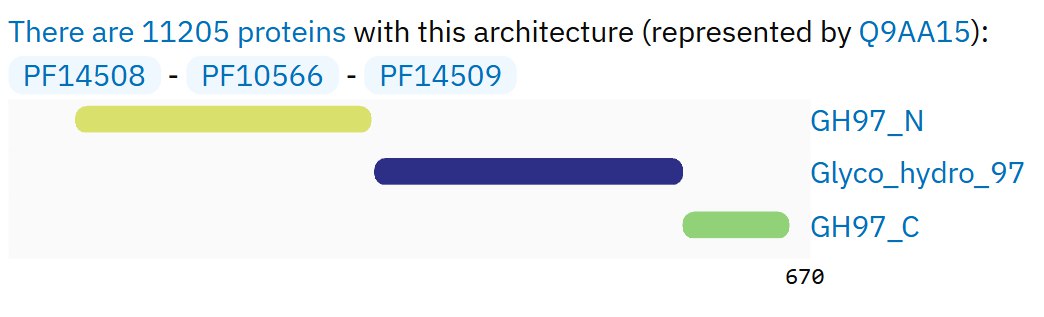
| Доменная архитектура 1 | PF14508 - PF10566 - PF14509 | |
| Белок с архитектурой 1 | Q9AA15 | Alpha-glucosidase, putative |
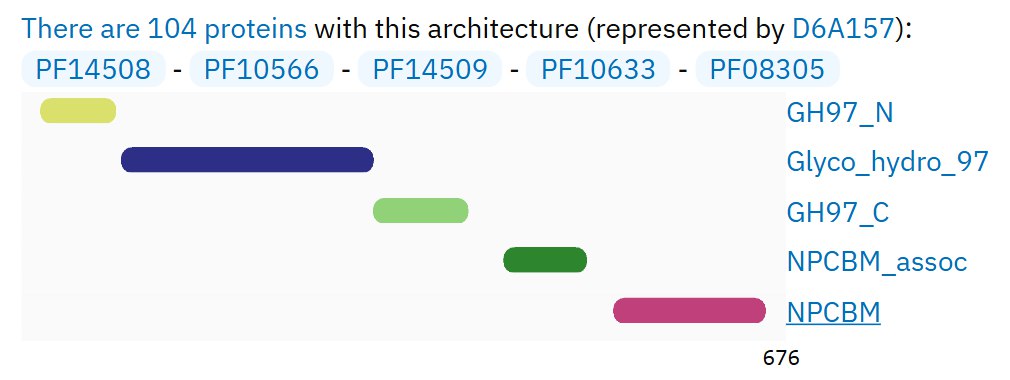
| Доменная архитектура 2 | PF14508 - PF10566 - PF14509 - PF10633 - PF08305 | |
| Белок с архитектурой 2 | D6A157 | Glycosyl hydrolase family 98 putative carbohydrate-binding module domain-containing protein |
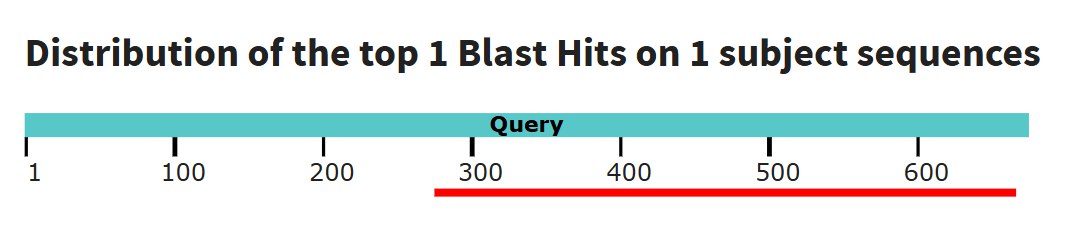
Белок Q9AA15 был выделен из бактерии Caulobacter vibrioides, D6A157 — из Streptomyces viridosporus. В UniprotKB белок D6A157 также имеет название Alpha-glucosidase. Карта локального сходства (рис. 2) демонстрирует гомологию данных белков, что проявляется в виде наличия больших участков сходства с минимальными разрывами, то есть с минимальными вставками или делециями. На рисунке 3 изображено, какие части последовательностей были локально выравнены, на рис. 4, 5 изображена доменная архитектура белков Q9AA15 и D6A157 соответственно.
В Query был поставлен Q9AA15 (ось абсцисс), а D6A157 является subject (ось ординат). Локальное выравнивание выявило сходство между участками 275-662 у Q9AA15 и 48-396 у D6A157. Участок 275-301 у Q9AA15 и 48-71 у D6A157 соответствует домену PF14508 (ID: Glycosyl-hydrolase 97 N-terminal). Исходя из названия не удивительно, что данный домен находится на N-конце обоих белков. На данном участке произошла 1 вставка/делеция у Q9AA15. У Q9AA15 данный домен намного больше, чем у D6A157, поэтому выравнилась лишь небольшая концевая часть. Участки 306-574 у Q9AA15 и 78-310 у D6A157 соответствуют исследуемому домену PF10566. Как можно заметить, в ходе эволюции у Q9AA15 в данном участке произошло 8 вставок или делеций по сравнению с D6A157. Участки 574-662 у Q9AA15 и 312-396 у D6A157 соответствуют домену PF14509 (ID: Glycosyl-hydrolase 97 C-terminal, oligomerisation). На данном участке произошла одна вставка/делеция у Q9AA15.