Практикум 8. Нуклеотидный BLAST
Задание 1. Ген, кодирующий δ-субъединицу АТФ-синтазы
В аннотированном файле генома GCA_003254725.2 (Canis lupus dingo) были идентифицированы последовательности, потенциально кодирующие субъединицу дельта АТФ-синтазы (с идентификатором белка XP_025312481.1, далее я буду работать именно с ним; и еще один участок, который помечен как псевдоген). Этот белок закодирован на 20 хромосоме. Она имеет идентификатор NC_064262. Ссылка на запись.
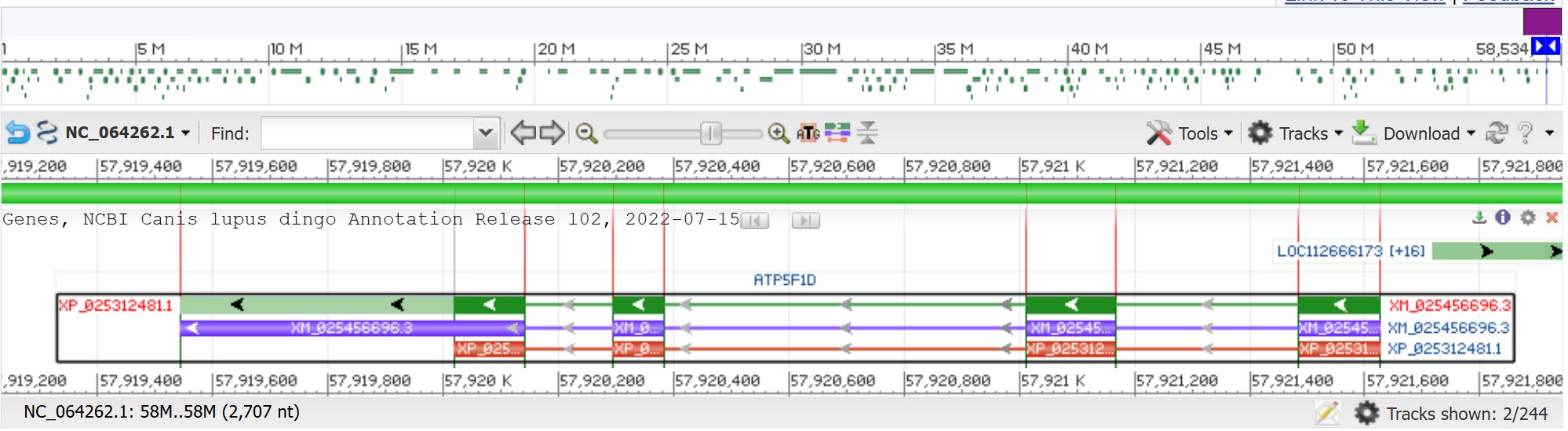
Далее в геномном браузере я нашел на этой хромосоме ген, кодирующий дельта-субъединицу АТФ-синтазы (XP_025312481.1). Идентификатор соответствующей нуклеотидной последовательности — XM_025456696.3. На рисунке 1 изображено геномное окружение XM_025456696.3. Файл гена с небольшой окрестностью.
Задание 2. BLAST для исследуемой последовательности
Для оценки консервативности кодирующей последовательности изучаемого гена был проведен поиск гомологов в геномах пауков (Araneae) из базы данных RefSeq Genomes с помощью алгоритмов BLAST: blastn и tblastn.
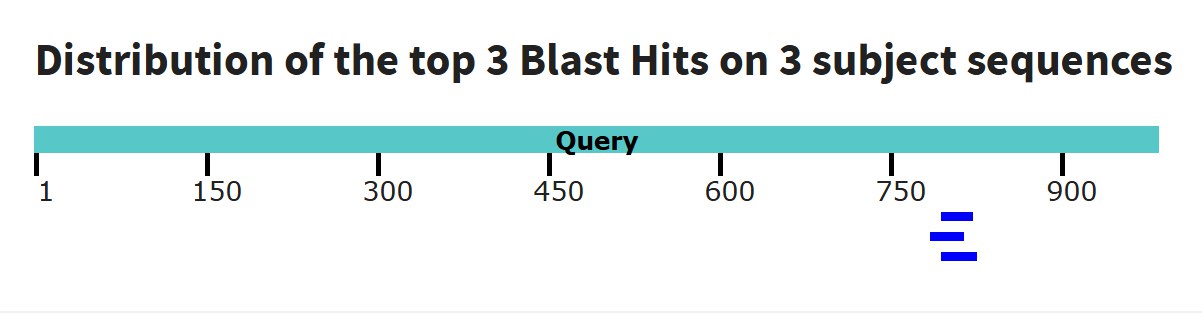
В базе данных на момент анализа присутствовало четыре геномные сборки, представляющие различные виды пауков. Параметры для blastn я оставил по умолчанию, а размер слова выставил 7. Алгоритм megablast не использовался, поскольку он оптимизирован для поиска очень сходных последовательностей, что маловероятно для столь удаленных групп организмов. Графический результат выдачи blastn показан на рис. 2.
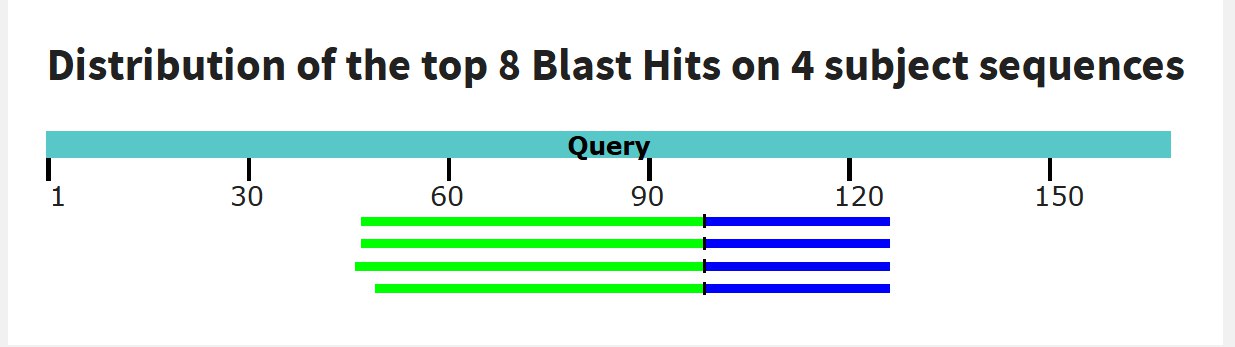
Затем я провел поиск гомологичных нуклеотидных последовательностей по tblastn. Tblastn принимает на вход белковую последовательность и ищет гомологов в сборке генома. При запуске все параметры были оставлены по умолчанию, кроме word size (я поставил 2). Результат представлен на рис. 3.
Задание 3. Поиск генов рРНК по далекому гомологу
Далее нужно было найти в геномной сборке Canis lupus dingo гомологи двух рРНК E.coli — 16S и 23S. Эти РНК являются структурной основой рибосом: 16S входит в малую субъединицу и отвечает за связывание мРНК (с последовательностью Шайна-Дольгарно), а 23S является частью большой субъединицы и катализирует образование пептидной связи.
Сначала я проиндексировал последовательности генома для работы локального BLAST. Была использована команда:
makeblastdb -in .\GCF_003254725.2_ASM325472v2_genomic.fna -dbtype nucl
Пояснения:
-in .\GCF_003254725.2_ASM325472v2_genomic.fna — имя входного файла генома исследуемого организма
-dbtype nucl — указываем, что база данных именно из нуклеотидных последовательностей
Для нахождения гомологичных последовательностей в исследуемой геномной сборке был проведен поиск с использованием алгоритма blastn, так как blastn отлично подходит для поиска далеких последовательностей. В качестве запросов (query) использовались нуклеотидные последовательности 16S и 23S рРНК E. coli в fasta-формате. Поиск выполнялся с помощью следующих команд:
blastn -task blastn -query .\16S_Ecoli.fasta.txt -db .\GCF_003254725.2_ASM325472v2_genomic.fna -out Dingo_Ecoli_16S.txt -outfmt 7
blastn -task blastn -query .\23S_Ecoli.fasta.txt -db .\GCF_003254725.2_ASM325472v2_genomic.fna -out Dingo_Ecoli_23S.txt -outfmt 7
Пояснения:
-task blastn — программа по умолчанию использует megablast
-query .\16S_Ecoli.fasta.txt — имя fasta-файла с последовательностью 16S рРНК E. coli.
-db .\GCF_003254725.2_ASM325472v2_genomic.fna — имя раннее созданной базы данных по последовательности генома Canis lupus dingo.
-out Dingo_Ecoli_16S.txt -outfmt 7 — имя выходного файла blastn в формате 7
Файл выдачи blastn по 16S рРНК E. coli. Файл выдачи blastn по 23S рРНК E. coli.
В случае поиска гомологов 16S рРНК blastn нашел 26 хитов (находок). Я их отфильтровал по значению E-value, чтобы оно было относительно маленьким для увеличения достоверности найденных предполагаемых гомологов. В итоге осталось 8 участков. Они все соответствуют участку [1496-1536] у query (16S рРНК E coli). Значит, для 16S рРНК было найдено 8 участков, которые гомологичны участку [1494-1536] у 16S рРНК.
В случае поиска гомологов 23S рРНК blastn нашел 42 хита. Я удалил из выдачи blastn те находки, у которых E-value был относительно высоким (больше 10^(-2)). Далее по достоверным находкам с помощью программы Inkscape я построил графическое изображение всех найденных 11 гомологов. Они изображены на рисунках 4-14.
Подписи ко всем рисункам:
Черной зигзагообразной линией обозначен запрос (query). В этом случае запросом всегда является последовательность 23S рРНК у E. coli. Салатовым цветом изображена находка в исследуемом геноме Canis lupus dingo. По середине указан идентификатор хромосомы или скэффолда, в котором были найдены гомологичные участки. Черными стрелками указано направление цепей (у query это всегда 5'-3', а у subject может меняться, так как гомологи были найдены как на прямой цепи, так и на обратной). Числами обозначены координаты начал и концов гомологичных участков query и subject.











Задание 4. Карты локального сходства двух геномов
Для сравнительного анализа были отобраны референсные геномы двух видов рода Streptococcus: Streptococcus pyogenes (NZ_CP049697.1) и Streptococcus ferus (NZ_LS483343.1), полученные из базы данных NCBI Nucleotide. S. pyogenes является возбудителем заболеваний животных (в том числе человека), таких как ангина, скарлатина, острая ревматическая лихорадка и другие. S. ferus участвует в образовании зубного налета, провоцируя кариес.
Сравнение геномов проводилось с использованием алгоритмов blastn, megablast и tblastx, по результатам которых были построены карты локального сходства. Они изображены на рисунках 15-17.
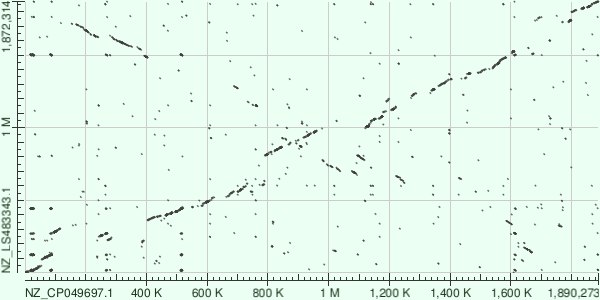
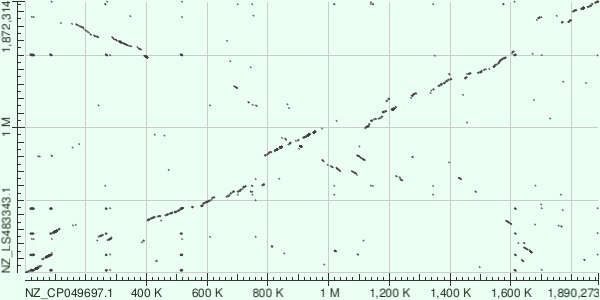
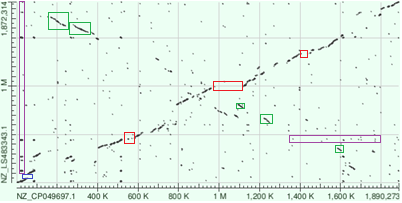
Наблюдаемые изменения в геноме по карте локального сходства blastn
На рисунке 17 изображены выделенные области, которые соответствуют различных генетическим перестройкам. Обозначения:
Красными прямугольниками выделены делеции в последовательности 1 (нижняя, Streptococcus pyogenes (NZ_CP049697.1))
Синим прямоугольником выделена делеция в последовательности 2 (верхняя, Streptococcus ferus (NZ_LS483343.1))
Зелеными прямугольниками выделены инверсии с транслокацией
Розовыми прямоугольниками выделены некторые дупликации (вертикально — в последовательности 2, горизонтально — в последовательности 1), которые так же отразились в результатх megablast.