1. Отобранные виды семейства Firmicutes
| Название | Мнемоника |
| Bacillus anthracis | BACAN |
| Clostridium tetani | CLOTE |
| Enterococcus faecalis | ENTFA |
| Finegoldia magna | FINM2 |
| Lactobacillus delbrueckii | LACDA |
| Lactococcus lactis | LACLM |
| Listeria monocytogenes | LISMO |
| Staphylococcus epidermidis | STAES |
2. Скобочная формула
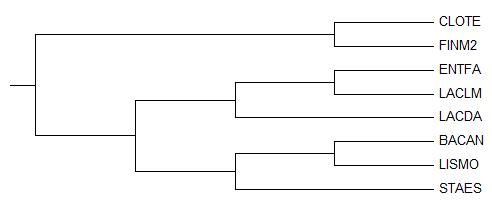
((CLOTE, FINM2), (((ENTFA, LACLM), LACDA), ((BACAN, LISMO), STAES)))
3. Изображение Филогенетического дерева
4. Нетривиальные ветви
{CLOTE, FINM2} vs {ENTFA, LACLM, LACDA, BACAN, LISMO, STAES}
{ENTFA, LACLM} vs {CLOTE, FINM2, LACDA, BACAN, LISMO, STAES}
{BACAN, LISMO} vs {CLOTE, FINM2, ENTFA, LACLM, LACDA, STAES}
{ENTFA, LACLM, LACDA} vs {CLOTE, FINM2, BACAN, LISMO, STAES}
{BACAN, LISMO, STAES} vs {CLOTE, FINM2, ENTFA, LACLM, LACDA}
1.
| Название | Мнемоника |
| Bacillus anthracis | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group |
| Clostridium tetani | cellular organisms; Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Enterococcus faecalis | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Finegoldia magna | cellular organisms; Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiales incertae sedis; Clostridiales Family XI. Incertae Sedis; Finegoldia |
| Lactobacillus delbrueckii | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Lactococcus lactis | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus |
| Listeria monocytogenes | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria |
| Staphylococcus epidermidis | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
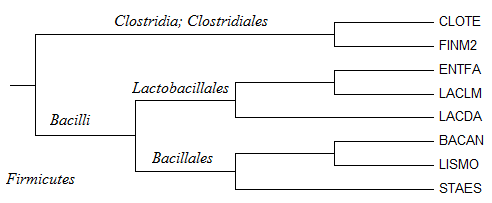
2.
Выбрал функцию - рибосомальный белок R2.
Для получения последовательностей белков составил список ID и подал его на вход сервису retrieve на сайте UniProt.
3.
Создал выравнивание отобранных белков программой mafft.
4 (дополнительное).
Выравнивание
5. fprotpars
Программа выдала лишь одно дерево:
+--------------RS2_LISMO
+--6
! ! +-----------RS2_STAES
! +--7
! ! +--------RS2_FINM2
! +--3
+--2 ! +-----RS2_LACDA
! ! +--4
! ! ! +--RS2_LACLM
! ! +--5
1 ! +--RS2_ENTFA
! !
! +-----------------RS2_CLOTE
!
+--------------------RS2_BACAN
Его скобочная формула:
(((((((RS2_LACLM, RS2_ENTFA), RS2_LACDA), RS2_FINM2), RS2_STAES), RS2_LISMO), RS2_CLOTE), RS2_BACAN)
В полученном дереве лишь клада 4 совпадает с реальностью. Все попытки как-то укоренить это дерево, чтобы оно совпало с правильным
становятся тщетными. В нем нет ветви отделяющей CLOTE и FINM2 от остальных, аналогично и клада ((BACAN, LISMO), STAES) со своими ветвями не определяема в выданном дереве.
6. fprotdist
RS2_BACAN 0.000000 0.352891 0.295251 0.397747 0.419908 0.382330 0.184069 0.242151
RS2_CLOTE 0.352891 0.000000 0.422336 0.390700 0.486929 0.421259 0.360844 0.392291
RS2_ENTFA 0.295251 0.422336 0.000000 0.551788 0.369588 0.254410 0.336172 0.436504
RS2_FINM2 0.397747 0.390700 0.551788 0.000000 0.505670 0.531117 0.493917 0.550807
RS2_LACDA 0.419908 0.486929 0.369588 0.505670 0.000000 0.370076 0.512328 0.548624
RS2_LACLM 0.382330 0.421259 0.254410 0.531117 0.370076 0.000000 0.439996 0.471129
RS2_LISMO 0.184069 0.360844 0.336172 0.493917 0.512328 0.439996 0.000000 0.335770
RS2_STAES 0.242151 0.392291 0.436504 0.550807 0.548624 0.471129 0.335770 0.000000
Аксиома ультраметричности может выполняться (даже на 6 сотых)
d(RS2_BACAN, RS2_CLOTE) < max[d(RS2_BACAN, RS2_ENTFA); d(RS2_CLOTE, RS2_ENTFA)]
0.352891 0.295251 0.422336
или нет (на 5 сотых)
d(RS2_LACDA, RS2_BACAN) > max[d(RS2_ENTFA, RS2_BACAN); d(RS2_LACDA, RS2_ENTFA)]
0.419908 0.295251 0.369588
Вывод: нет ультраметричности.
d(RS2_BACAN, RS2_CLOTE) + d(RS2_ENTFA, RS2_FINM2) = 0.352891 + 0.551788 = 0.904679
d(RS2_BACAN, RS2_ENTFA) + d(RS2_CLOTE, RS2_FINM2) = 0.295251 + 0.390700 = 0.685951
d(RS2_BACAN, RS2_FINM2) + d(RS2_CLOTE, RS2_ENTFA) = 0.397747 + 0.422336 = 0.820083
В принципе, похоже на выполнение свойства аддитивности. Первая и последняя сумма приблизительно равны с точностью 0.8 (по сравнению со второй - вообще не равны)
и больше второй.
7. fneighbor
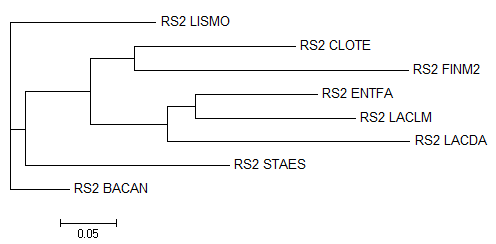
Neighbor-Joining алгоритм
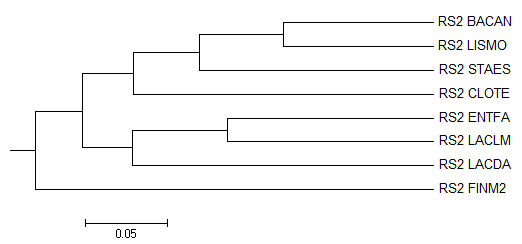
 | UPGMA алгоритм
 |
Дерево, построенное по первому алгоритму, совпадает с реальным, если его укоренить на третьем узле слева.
В дереве второго алгоритма клады ((ENTFA, LACLM), LACDA) и ((BACAN, LISMO), STAES) совпадают с правильными, но положение CLOTE не верно.
Из ошибочности второго дерева можно заключить, что для данных белков гипотеза молекулярных часов не работает.
8. TreeTop
TreeTop