| Neighbor-Joining | UPGMA |
 |
 |
|
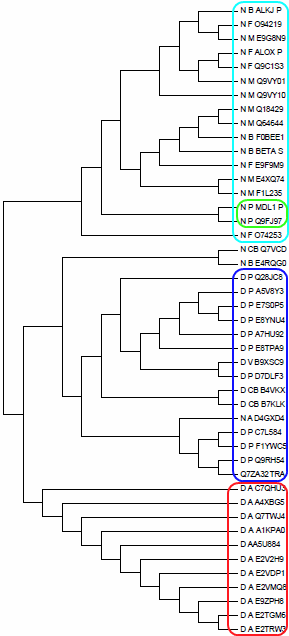
В результате использования программы fneighbor я получил неукоренненное дерево, в котором последовательности двух доменных архитектур четко разделялись (за исключение пары представителей). Далее укоренил полученное дерево таким образом, чтобы от корня отходили две клады, соответствующие двум доменным архитектурам. Скобочная струтктура |
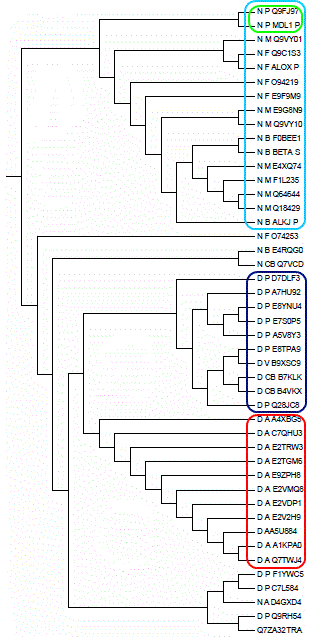
Дерево, построенное по данному алгоритму, исходно было укорененным, причем практически так же, как и я сделал в предыдущем случае (промахнулся на одну последовательность). Скобочная струтктура |
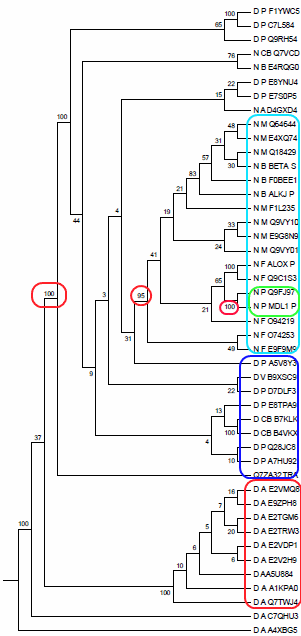
Профиль для первой архитектуры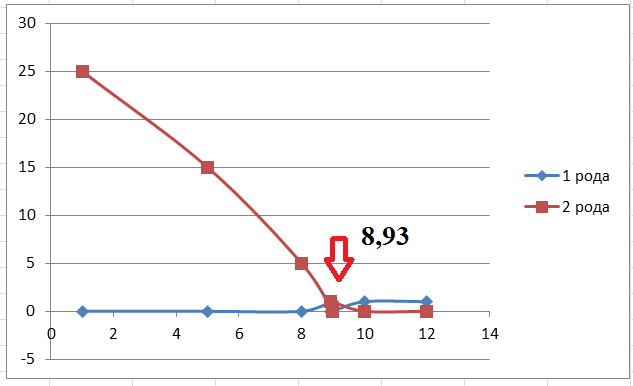
Для порога 8,93: TP = 20 (все), TN = 24, FN = 0, FP = 1 |
Профиль для второй архитектуры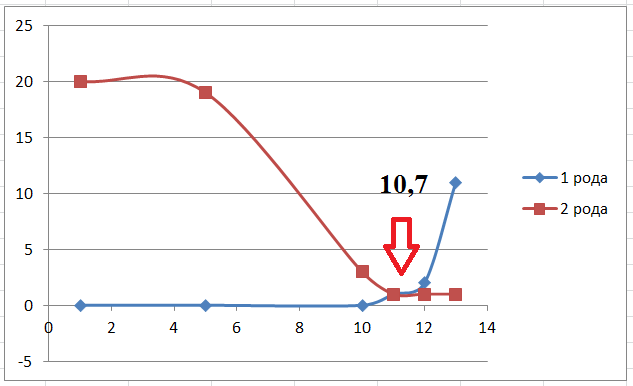
Для порога 10,7: TP = 25 (все), TN = 18, FN = 0, FP = 2 |