Задание 1
Упр.1.
С помощью программы einverted пакета EMBOSS я нашел в последовательности заданной тРНК (PDB ID 1O0B) инвертированные участки. Мне удалось найти 2 разных набора параметров, при которых выдача программы соответствовала реальным координатам стеблей. Однако таким образом получилось предсказать лишь 2 стебля из 4 (и только по одному за раз).
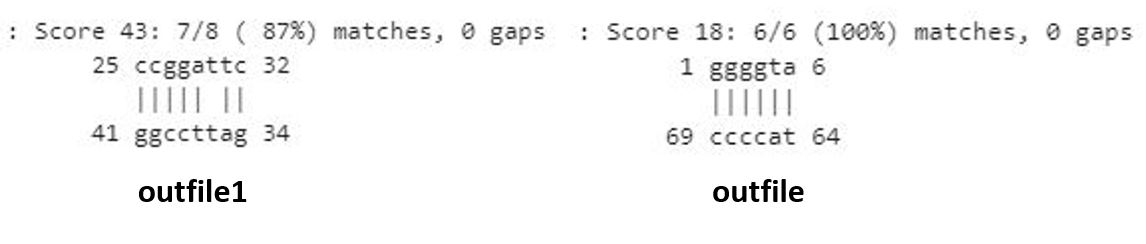
Запуск 1:
! einverted -sequence rna.seq -gap 12 -threshold 10 -match 3 -mismatch -3 -outfile outfile -outseq seqout
Запуск 2:
! einverted -sequence rna.seq -gap 50 -threshold 43 -match 7 -mismatch -6 -outfile outfile1 -outseq seqout1
Упр.2.
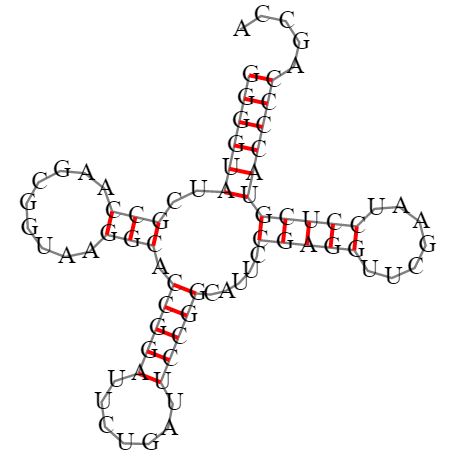
Далее вторичная структура той же тРНК бала предсказана с помощью ViennaRNA (Рис. 2). Результаты сравнения полученных предсказаний отражены в таблице:
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | [G]902/[C]971-[A]907/[U]966; 6 п.о. |
Координаты предсказаны верно | Координаты предсказаны верно |
| D-стебель | [G]910/[C]925-[C]912/[G]923; 3 п.о. |
Нет предсказания | Координаты предсказаны верно |
| T-стебель | [C]949/[G]965-[G]953/[C]961; 5 п.о. |
Нет предсказания | Координаты предсказаны верно |
| Антикодоновый стебель | [A]937/[U]933-[C]944/[A]926; 8 п.о. |
[G]936/[C]934-[G]943/[C]927; 7 п.о. |
[U]939/[A]931-[G]943/[C]927; 5 п.о. |
| Общее число канонических пар нуклеотидов в стеблях | 18 | 13 (12 предсказаны верно) | 18** (все предсказаны верно) |
** Одна из пар G-C, предсказаных по алгоритму Зукера, является согласно find_pair неканонической. Ее я в данном случае не учитывал.
Задание 2
Упр.1.
Сначала был создан скрипт с определением множеств атомов set1, set2 и set3, после этого я написал скрипт, последовательно отображающий эти множества.
Упр.2.
Для определения множеств полярных и неполярных атомов белка и различных остатков в составе ДНК я написал скрипт define_p_np.spt. Затем, используя отдельные команды select в консоли JMol (Рис. 3), я получил количество контактов разных типов.
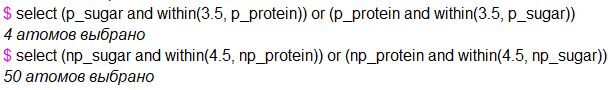
Результаты выполнения команд я представил в виде таблицы:
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 4 | 50 | 54 |
| остатками фосфорной кислоты | 33 | 34 | 67 |
| остатками азотистых оснований со стороны большой бороздки | 62 | 81 | 143 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 2 | 2 |
Полученные данные говорят о том, что в структуре 1P47 белок связывается с ДНК со стороны больной бороздки. Также можно предположить, что это взаимодействие является специфичным по потношению к последовательности ДНК (число контактов с большой бороздкой, несущей наибольшее количество информации о последовательности, превышает число контактов с оставом и с малой бороздкой). При этом неполярные взаимодействия (дисперсионные взаимодействия, гидрофобный эффект) являются более многочисленными, чем полярные.
Упр.3.
С помощью программы nucplot я получил отображение контактов между ДНК и белком (Рис. 4).
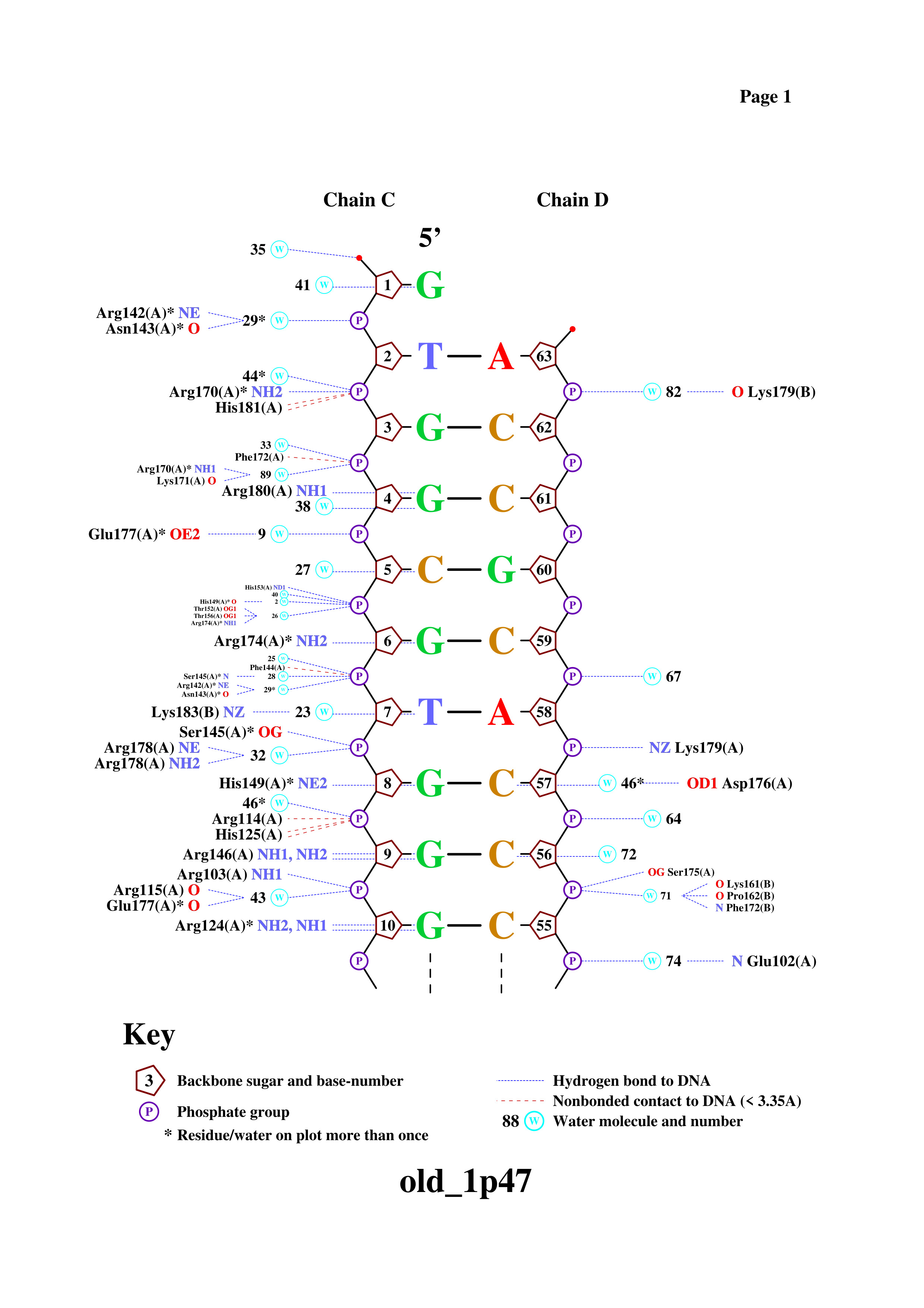
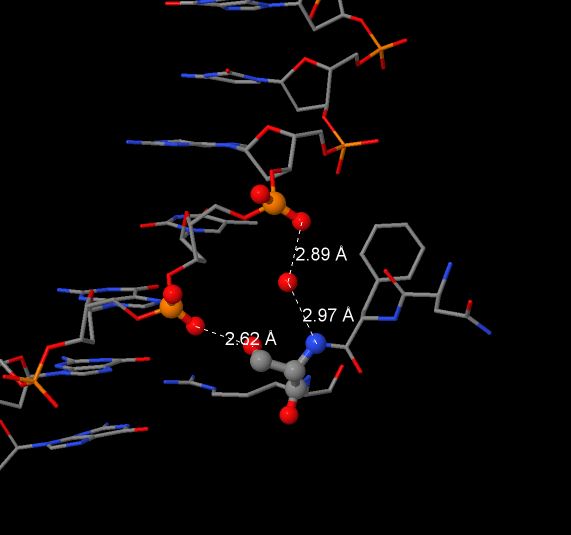
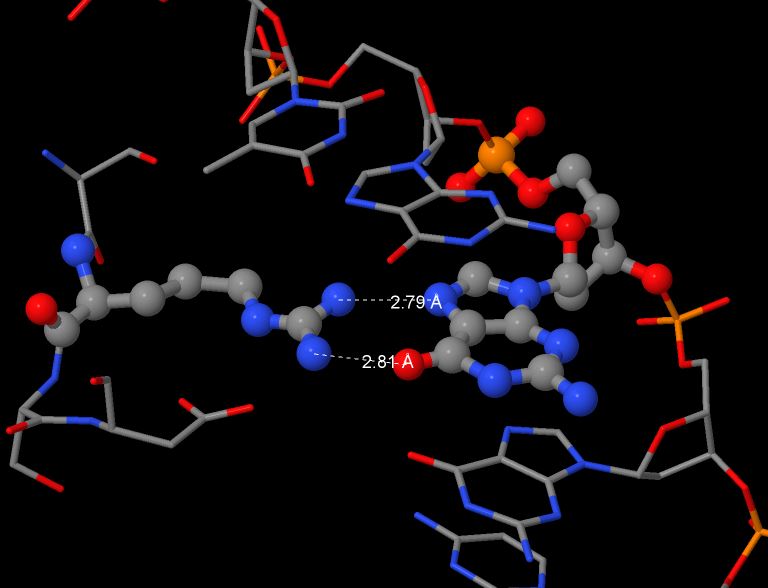
Наибольшее количество непосредственных контактов с ДНК образуют остатки Arg124 и Arg146 (2 водородные связи). Некотрые другие остатки также участвуют в образовании 2 водородных связей, однако хотя бы 1 из них является частью водяного мостика (то есть, эти остатки взаимодействуют с ДНК не напрямую, а через посредство молекулы воды). Для одного из них, Ser145, я решил получить детальное изображение (Рис. 5, водородные связи обозначены измерениями).
Судя по полученной схеме, в распознавании последовательности ДНК, вероятно, участвуют остатки Arg124, Arg146, His149, Arg174, Asp176, Arg180 и Lys183. По моему мнению, наиболее важную роль играют Arg124 и Arg146, каждый из которых образует 2 водородные связи с гуанином в составе ДНК, способствуя эффективному распознаванию этого азотистого основания. Детальное изображение я получил для остатка Arg146 (Рис. 6).