Задание №1
Поиск консервативного мотива в Jalview.
Для выполнения данного задания было выбрано семейство белков теплового шока HSP70 (PF00012).
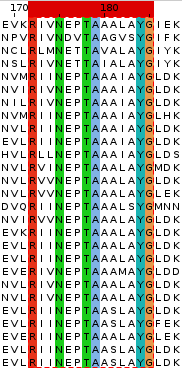
Скачав выравнивание seed, состоящее из 27 последовательностей и имеющее длину порядка 690 аминокислотных остатков, я открыл его в программе Jalview. При пороге Identity Treshold, равном 100%, нашлось несколько консервативных мотивов с высоким IC. Я выбрал один из них со следующим паттерном:
R.{2}N[ED].TA.{4}YG
Фрагмент из выравнивания с соответствующим паттерном показан на рисунке 1.
Отметим, что во второй позиции паттерна (после R) во всех последовательностях следует либо L, либо I, либо V. Это довольно схожие аминокислоты с явным гидрофобным характером. Тем не менее в паттерне эта позиция была обозначена в качестве точки, означающей любую аминокислоту. Это было сделано из-за того, что в позиции варьируют три и более аминокислоты. Или, например, после N следует либо D, либо E, отличающиеся лишь на метиленовый фрагмент (данная тонкость была учтена в паттерне, поскольку варьируют всего две буквы).
С помощью функции Find в Jalview я поискал данный паттерн в выравнивании. Он нашёлся только 27 раз (в 27 последовательностях), как и ожидалось. Иными словами, данный паттерн имеет высокое значение IC.
Далее был произведён поиск по данному мотиву в базе данных SwissProt на сайте MyHits. При этом было найдено 1074 находок! При этом абсолютное большинство из них входило в множество гомологов Hsp70 (HscA, SSB1), либо являлось непросредственно самим Hsp70. Это говорит о том, что данный паттерн прекрасно описывает белки из выбранного семейства.
Задание №2
Поиск мотивов, специфичных для клады.
Далее на основе выравнивания было построено дерево с помощью алгоритма UPGMA (Average distance), представленное на рисунке 2.
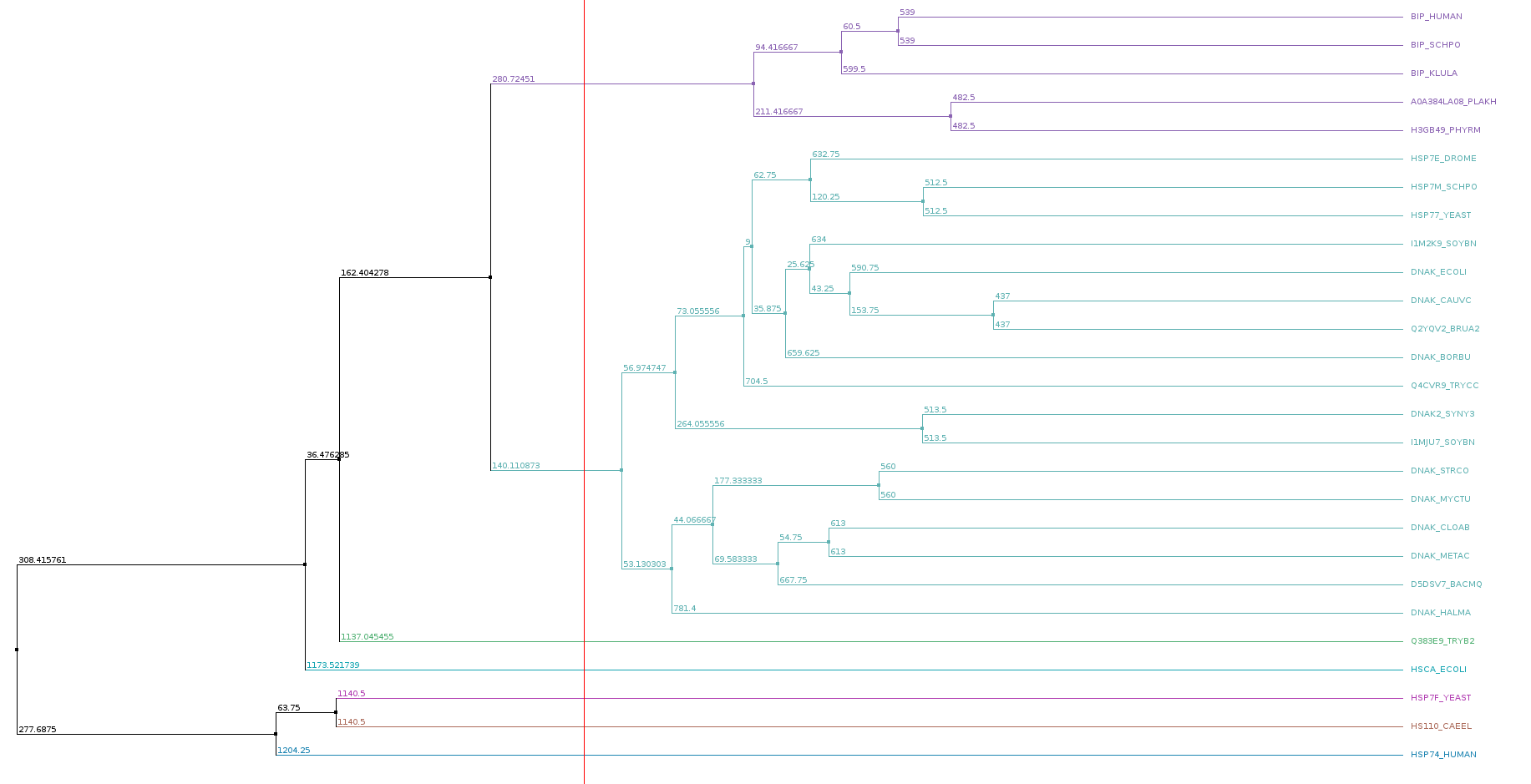
Было выделено 7 групп, из которых 5 являются тривиальными (содержат всего одну последовательность). Далее я выбрал группу из 17 последовательностей, которая выделена бирюзовым цветом на рисунке 2, и поместил соответствующее выравнивание в отдельное окно, окрасив его в соответствии с ранее перечисленными характеристиками (Identity Treshold = 100%, окраска Clustal). Файл с выравниванием в формате fasta можно скачать здесь: Project17.fasta.
Далее мною был найден мотив со следующим паттерном:
V.I.{3}QGER
Данный мотив встречался все 17 раз в выравнивании выбранной группу и также 17 раз в выравнивании всех последовательностей. То есть данный мотив встречается лишь в рамках выбранной группы, что говорит о его специфичности для выбранной клады.
Задание №3
PSI-BLAST.
В рамках данного задания был выбран AC C4Z088, принадлежащий предполагаемому белку-локализатору перегородки MinC бактерии Eubacterium] eligens (strain ATCC 27750).
Ниже представлена таблица итераций PSI-BLAST с вышеприведённым AC.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 162 | Q2P036.1 | 0.004 | Q5F5V4.1 | 0.005 |
| 2 | 188 | O25693.2 | 2,00E-09 | A7H8E6.1 | 0.050 |
| 3 | 188 | Q9ZM51.1 | 1,00E-11 | A7H8E6.1 | 0.012 |
Как видно, уже после третьей итерации число находок, проходящее порог в 0.005 E-VALUE, перестало меняться. При этом E-VALUE худшей из ''правильных'' и лучшей из ''неправильных'' находок имело различие в 9 порядков, что говорит о хорошей обособленности данного семейства с точки зрения схожести белков, входящих в него.
Задание №4
Psi-blast.
Для выполнения задания был выбран геном бактерии Saccharopolyspora spinosa (strain CCTCC M206084). Краткий рассчёт демонстрируется в ноутбуке.
Встречаемость динуклеотидов AT в геноме составила 245325, а ожидаемое число AT, рассчитанное по формуле Len_genome * prob_A * prob_T, оказалось равным 234393.4.
Полученное в ходе теста chisquare p-value оказалось равным 4e-56. Следовательно, мы можем поверить в то, что верна альтернативная гипотеза (ожидаемое и наблюдаемое значения встречаемости AT значимо различны).