Предсказание вторичной структуры тРНК (PDB ID 1EHZ). Поиск ДНК-белковых контактов (PDB ID 1D5Y)
Предсказание вторичной структуры тРНК
Для того чтобы сравнить работу различных алгоритмов по выявлению вторичной структуры тРНК по её первичной структуре, воспользуемся результатами, полученными в предыдущем задании с помощью программ find_pair и analyze пакета 3DNA, программой einverted из пакета EMBOSS и программой RNAfold из пакета Viena Rna Package, которая реализует алгоритм Зукера. Результаты сравнения представлены в таблице 1.
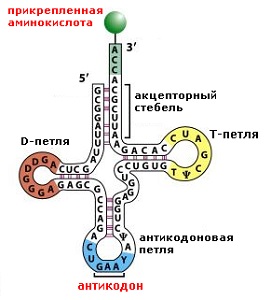
На схематичном изображении вторичной структуры тРНК, представленном выше, мы можем увидеть взаимное расположение стеблей и их названия.
| Таблица 1. Реальная и предсказанная вторичная структура тРНК из файла 1EHZ.pdb | |||
| Стебли | Результаты программ find_pair и analyze (реальная структура) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-1-7-3' 5'-66-72-3' Всего 7 пар |
- | 5'-1-7-3' 5'-66-72-3' Всего 7 пар |
| D-стебель | 5'-10-13-3' 5'-22-25-3' Всего 4 пары |
- | 5'-10-13-3' 5'-22-25-3' Всего 4 пары |
| T-стебель | 5'-49-53-3' 5'-61-65-3' Всего 5 пар |
5'-49-53-3' 5'-61-65-3' Всего 5 пар |
5'-49-53-3' 5'-61-65-3' Всего 5 пар |
| Антикодоновый стебель | 5'-27-31-3' 5'-39-43-3' Всего 5 пар |
5'-22-31-3' 5'-39-48-3' Всего 5 пар |
5'-27-31-3' 5'-39-43-3' Всего 5 пар |
| Общее число канонических пар нуклеотидов в стеблях | 18 | 12 | 18 |
Для программы einverted я использовала входные параметры: gap penalty = 12; minimum score threshold = 5; match score = 3; mismatch score = -4.
Из таблицы видно, что алгоритм Зукера является более эффеткивным в поиске вторичной структуры тРНК.
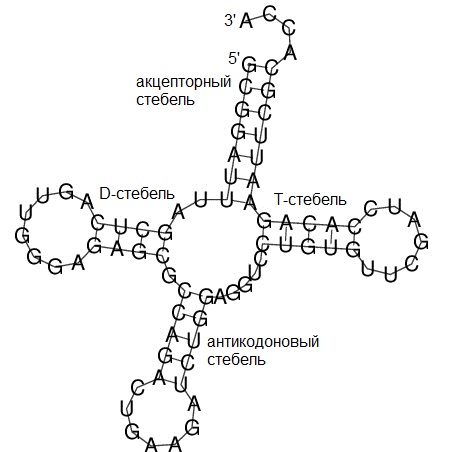
Ниже представлено изображение вторичной структуры тРНК, полученное при выполнении программы RNAfold. Это предсказание было первым по счёту. Видно, что все стебли были правильно предсказаны, единственно программа не указывает дополнительные водородные связи, стабилизирующие третичную структуру тРНК.
Поиск ДНК-белковых контактов
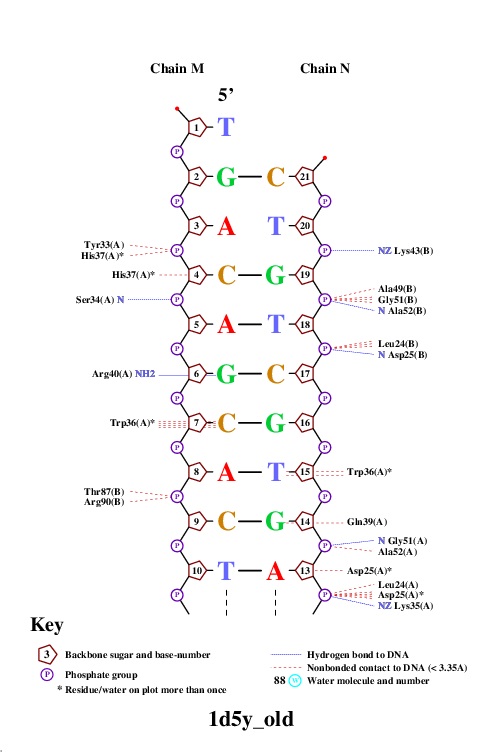
Для программы Jmol я создала скрипт define.spt, в котором определила множество атомов кислорода 2'-дезоксирибозы (set1), множество атомов кислорода в остатке фосфорной кислоты (set2) и множество атомов азота в азотистых основаниях (set3). Затем я написала другой скрипт script.spt, который выдаёт последовательное изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов set1, затем set2 и set3. Также в нём дополнительно описаны множества полярных и неполярных атомов азотистых оснований, смотрящих в сторону большой и малой бороздок (set4 - set7 соответственно). Они необходимы для описания ДНК-белковых контактов.
Если атомы кислорода (О) и азота (N) считать полярными, а атомы углерода (C), фосфора (P) и серы (S) - неполярными, то можно оценить количество ДНК-белковых контактов разной природы. Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5Å. Тогда для заданной ДНК-белковой структуры (PDB ID 1D5Y) результаты будут выглядить следующим образом (таблица 2).
| Таблица 2. Контакты разного типа в комплексе 1D5Y.pdb | |||
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 1 | 9 | 10 |
| остатками фосфорной кислоты | 10 | 9 | 19 |
| остатками азотистых оснований со стороны большой бороздки | 2 | 13 | 15 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 0 | 0 |
Из таблицы видно, что атомы азотистых оснований со стороны малой бороздки вовсе не образуют контакты с белком, что может быть связано с их стерической недоступностью. Также остатки 2'-дезоксирибозы чаще образуют неполярные контакты, как и остатки азотистых оснований со стороны большой бороздки, когда остатки фосфорной кислоты, наоборот, охотнее образуют полярные контакты.
Затем с помощью программы nucplot я получила популярную схему ДНК-белковых контактов, представленную ниже.
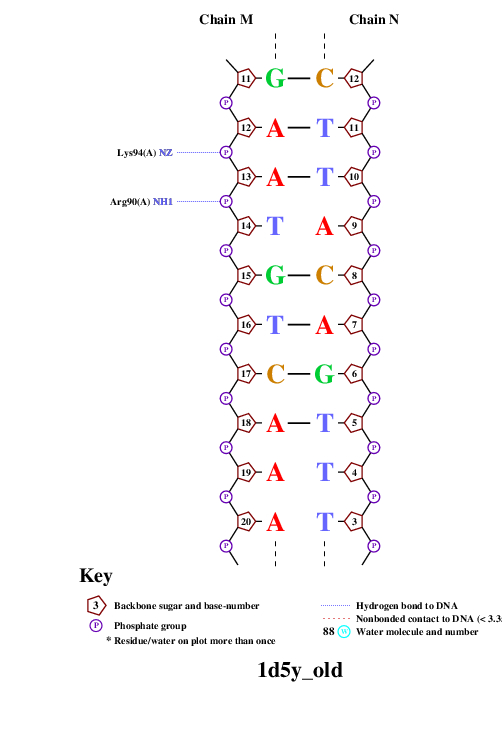
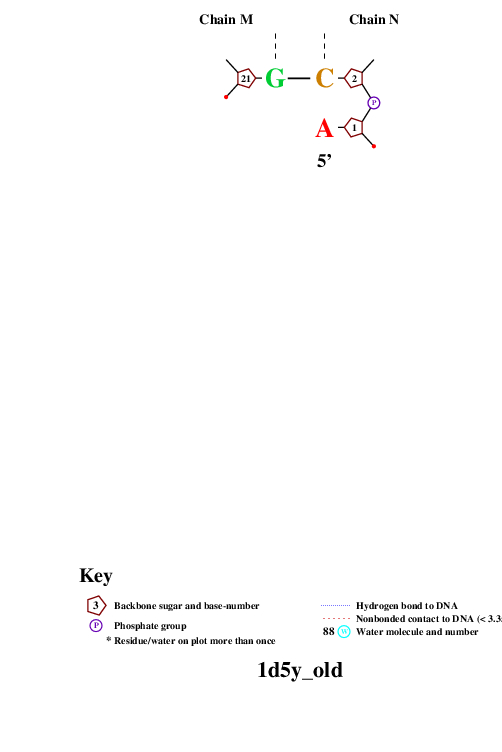
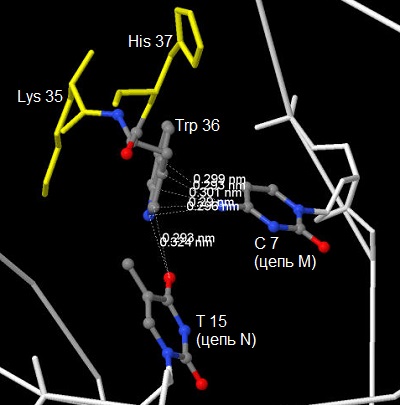
На данной схеме Trp 36 (цепь A) и Asp 25 (цепь A) обладают наибольшими числами контактов с ДНК (7 и 6 соответственно).
Я считаю, что из всех аминокислотных остатков, образующих контакты с ДНК, наиболее важным для распознавания её последовательности является Trp 36 (цепь A), так как он образует 5 контактов с цитозиновым азотистым основанием (№7) из M цепи и 2 контакта с тиминовым азотистым основанием (№15) из N цепи. Большинство остальных аминокислотных остатков, показанных на схеме, образуют контакты лишь с остатками фосфорной кислоты или остатками 2'-дезоксирибозы, выполняя роль стабилизации структуры, но не отвечая за специфичное связывание. Ниже представлены изображения, полученные с помощью программы Jmol, иллюстрирующие контакты Trp 36 и Asp 25 с ДНК.
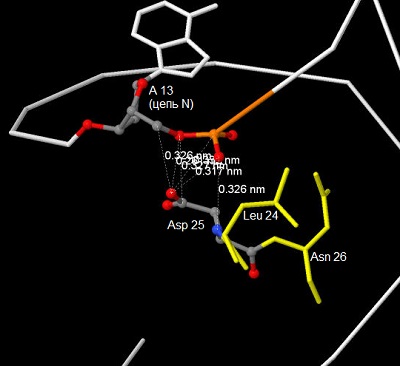
Жёлтым цветом выделены аминокислотные остатки, соседние с рассматриваемыми Asp 25 и Trp 36. Цепь ДНК изображена белым остовом, за исключением нуклеотидов, участвующих в контактах. Пунктирными линиями с цифрами обозначены контакты и подписаны расстояния между контактирующими атомами. Также на картинках подписаны названия азотистых оснований и цепи ДНК, к которым они принадлежат.