Банки нуклеотидных последовательностей
Характеристика качества сборки генома Candida albicans
Для работы я выбрала грибок Candida albicans из семейства Saccharomycetes отдел Ascomycota, который является возбудителем кандидоза (молочницы). В норме этот грибок живёт в желудочно-кишечном тракте у 80% людей, не вызывая воспалений. Однако, для людей с ослабленным иммунитетом возможно увеличение количества грибковых клеток, что приводит к кандидозу.
На сайте NCBI я обнаружила 36 сборок генома, из которых лишь одна GCA_000149445.2 была наиболее полной, столько же проектов по секвенированию организма и 35 образцов.
Для выбранной мной сборки использовался образец SAMN02953609 (BioSample ID: 2953609). В GenBank он имеет ID gb|AAFO00000000.1. Это штамм Candida albicans WO-1, который имеет следующее систематическое положение по номенклатуре NCBI: cellular organisms; Eukaryota; Opisthokonta; Fungi; Dikarya; Ascomycota; Saccharomyceta; Saccharomycotina; Saccharomycetes; Saccharomycetales; Debaryomycetaceae; Candida/Lodderomyces clade; Candida; Candida albicans (таксономический ID 294748).
Проект по секвенированию данного организма PRJNA16373 (BioProject ID: 16373) был опубликован 4 января 2009 года в журнале Nature в статье под названием "Evolution of pathogenicity and sexual reproduction in eight Candida genomes", что переводится как "Эволюция патогенности и полового размножения в геномах восьми видов из рода Candida". Но зарегистрирован этот проект был 28 марта 2006 года двумя организациями: University of Iowa, Department of Biological Sciences, USA, Iowa City и Broad Institute. Он был выполнен в Broad Institute с 10-кратным покрытием и с использованием секвенирования всего генома методом дробовика, суть которого заключается в фрагментации ДНК на мелкие участки случайным образом, которые затем секвенировали обычными методами. Таким образом, перекрывающиеся случайные фрагменты ДНК можно собрать в одну большую последовательность. Также проект можно найти в базе данных GOLD (Genomes Online Database) по идентификатору Gp0002578. Помимо этого, есть публикации в PubMed и PMC (PubMed Central). Можно найти результаты секвенирования в базах данных BioSample и Assembly. В результате секвенирования было распознано 5 хромосом: номера 3 (1,768,732 пн), 5 (1,198,695 пн), 6 (1,043,947 пн), 7 (958,737 пн), R (2,299,365 пн), определено 5752 последовательностей белка. Общая длина синтезированной ДНК равна 14,472,953 пн, а общая длина гэпов в сборке равна 56,540 пн.
Число контигов оказалось равным 86, скэффолдов - 17 (22, если учитывать 5 хромосом). Подробные данные о названиях и длинах контигов и скэффолдов можно найти в таблице. Гэпы между скэффолдами отсутствуют. Общее количество хромосом и плазмид в сборке равно 9. Ниже представлены характеристики качества сборки (таблица 1).
| Таблица 1. Параметры сборки | |||||
| Количество | N50 | L50 | Самый длинный (его длина) | Самый короткий (его длина) | |
| Контиг | 86 | 537,130 пн | 10 | AAFO01000007 (1,293,053 пн) | AAFO01000076 (2,103 пн) |
| Скэффолд | 17 | 1,768,732 пн | 3 | CH672346.1 (3,194,068 пн) | GG670284.1 (5,039 пн) |
Ссылка на последовательность AAFO01000058 контига с длиной в 537,130 пн.
Описание ключей, используемых в таблицах особенностей формата GenBank
- source
Указывает на биологический источник участка генетического материала определённой протяжённости; Ключ является обязательным; Разрешено указывать этот ключ более одного раза на одну последовательность; Каждая запись должна иметь как минимум один такой ключ, охватывающий всю последовательность, или несколько ключей, которые вместе охватывают всю последовательность.
source 1..86
/organism="Candida albicans WO-1"
/mol_type="genomic DNA"
/strain="WO-1"
/db_xref="taxon:294748"
Любая область последовательности, функция которой заключается в регуляции транскрипции или трансляции.
regulatory 95..100
/gene="sod"
/regulatory_class="ribosome_binding_site"
Область биологического интереса, идентифицированная как ген, которой было присвоено название; Ключ описывает интервал ДНК, соответствующий генетическому признаку или фенотипу; По определению он не строго привязан к позиции концов интервала, так как ключ предназначается для обозначения области, где ген расположен.
gene 95..746
/gene="sod"
Кодирующая последовательность; Последовательность нуклеотидов, которая соответствует последовательности аминокислот в белке, включая стоп-кодон; Ключ включает последовательность аминокислот, транслированную с последовательности нуклеотидов.
CDS 109..717
/gene="sod"
/EC_number="1.15.1.1"
/codon_start=1
/transl_table=11
/product="superoxide dismutase"
/db_xref="GI:44011"
/db_xref="GOA:P28763"
/db_xref="InterPro:IPR001189"
/db_xref="UniProtKB/Swiss-Prot:P28763"
/protein_id="CAA45406.1"
/translation="MTYELPKLPYTYDALEPNFDKETMEIHYTKHHNIYVTKLNEAVS
GHAELASKPGEELVANLDSVPEEIRGAVRNHGGGHANHTLFWSSLSPNGGGAPTGNLK
AAIESEFGTFDEFKEKFNAAAAARFGSGWAWLVVNNGKLEIVSTANQDSPLSEGKTPV
LGLDVWEHAYYLKFQNRRPEYIDTFWNVINWDERNKRFDAAK"
мРНК (матричная РНК); Включает 5' нетранслируемую область (5'UTR) кодирующей последовательности гена (CDS = coding sequences, экзоны) и 3' нетранслируемую область (3'UTR).
mRNA join(1603..1891,2322..2438,2538..2633,2801..2843,
2918..3073,3167..3247,3874..3972,4082..4637)
/gene="CCT"
1) Область на 5'-конце зрелого транскрипта (предшествующая старт-кодону), которая не транслируется в белок.
2) Область на 5'-конце вирусной РНК (предшествующая первому старт-кодону), которая не транслируется в белок.
5'UTR 1603..1712
/gene="CCT"
1) Область на 3'-конце зрелого транскрипта (после стоп-кодона), которая не транслируется в белок.
2) Область на 3'-конце вирусной РНК (после последнего стоп-кодона), которая не транслируется в белок.
3'UTR 4310..4637
/gene="CCT"
Сайт нековалентного связывания белка в нуклеиновых кислотах.
protein_bind 166..175
/gene="Mlp84B"
/note="matches consensus at only 9 of 10 positions"
/bound_moiety="MEF2"
Область генома, которая кодирует части сплайсированных мРНК, рРНК и тРНК; может содержать 5'UTR (5' нетранслируемую область), все CDS (всю кодирующую последовательность гена) и 3'UTR (3' нетранслируемую область).
exon 1310..2135
/gene="Mt-PK"
/note="Form I,V,VI,VII,VIII mRNA"
Область генома, содержащая повторяющиеся элементы.
repeat_region 5153..5434
/gene="Mt-PK"
/rpt_family="Alu-J"
/rpt_type=dispersed
Описание одного из массовых геномных проектов
100K Pathogen Genome Project был запущен в июле 2012 года Бартом Веймером (Bart Weimer). Целью данного проекта является секвенирование геномов 100,000 инфекционных микроорганизмов, чтобы создать базу данных бактериальных последовательностей генома, что ускорит диагностику болезней, связанных с порчей продуктов питания, и сократит количество вспышек инфекционных заболеваний. Изначальными партнёрами-учредителями данного проекта являются Калифорнийский университет в Де́йвисе, американская компания Agilent Technologies, производящая измерительное, электронно-медицинское оборудование и оборудование для химического анализа, и американское Управление по санитарному надзору за качеством пищевых продуктов и медикаментов (USFDA). В качестве соисполнителей выступили Центры по контролю и профилактике заболеваний США (CDC) и Министерство сельского хозяйства США (USDA). В данном проекте осуществляется высокопроизводительное секвенирование следующего поколения (next-generation sequencing (NGS)) для исследования геномов целевых микроорганизмов. Полногеномное секвенирование проводится для небольшого числа микроорганизмов с целью дальнейшего их использования в качестве эталонных геномов. Большинство бактериальных штаммов будет секвенировано и собрано как "эскизы" геномов. Однако, в рамках проекта также производятся закрытые геномы для различных кишечных патогенов. Эта стратегия даёт возможность совместной работы по всему миру для определения наборов генетических биомаркеров, связанных с важными патогенными особенностями. Этот пятилетний проект в результате приведёт к созданию свободной и общедоступной базы данных с информацией о последовательности генома каждого патогена. Завершённые последовательности будут храниться в общественной базе данных Национальных институтов здравоохранения США (NIH) в Национальном центре биотехнологической информации США (NCBI). Используя эту базу данных, учёные смогут разрабатывать новые методы контроля болезнетворных бактерий в пищевой цепи.
Подробную информацию о проекте вы можете найти на сайте NCBI или на официальном сайте проекта.
Составление таблицы митохондриальных генов организма из типа Euglenozoa
Сначала я осуществила поиск в базе данных Nucleotide полных митохондриальных геномов организмов из типа Euglenozoa с помощью запроса:
"mitochondrion"[TITL] AND "Euglenozoa"[Organism]
В результате я получила 8 ссылок, из которых 6 - это секвенированные линейные РНК. Лишь 2 ссылки указывали на кольцевые молекулы митохондриальной ДНК из организма Diplonema papillatum. Тогда следующий запрос выдаст все полные митохондриальные геномы таксона и число находок (2):
("mitochondrion"[TITL] AND ("complete sequence"[TITL] OR "complete genome"[TITL])) AND
"Euglenozoa"[Organism]
Поиск в базе данных RefSeq не дал результатов по запросу:
("mitochondrion"[TITL] AND ("complete sequence"[TITL] OR "complete genome"[TITL])) AND
"Euglenozoa"[Organism] AND srcdb_refseq[PROP]
Обе находки относятся к базе данных GenBank. Я выбрала находку с ID=JQ302962.1
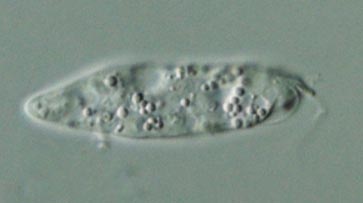
Diplonema papillatum относится к роду свободноживущих протистов, которые отличаются от представителей рода Rhynchopus отсутствием полностью жгутиковых дисперсионных стадий. Для представителей рода Diplonema характерно наличие двух коротких жгутиков одинаковой длины с двумя субапекальными щелями. Интересно, что были зарегистрированы случаи заражения моллюсков данными протистами и случаи внезапного разложения аквариумных растений, если в воде присутствовали представители рода Diplonema. Протисты были изначально описаны в 1914 году. Позднее они были заново обнаружены в 1960-х годах и ошибочно отнесены к классу Эвгленовых. Ниже представлена фотография рассматриваемого организма.
В выбранной находке содержится 5763 пн, но всего один ген "nad7", расположенный с 2534 нуклеотида по 2715 нуклеотид включительно:
2521 cctccag agtgtcctag ctgtattcat ggcttacagg atgacagcta 2581 cgacctctca gggcatccat gctgtggaag gtccaaaggg agagctgcac atcagcctca 2641 ctgtcaccaa cagcagcatg tgacggtgca gagtgcgtcc tgcagaccta ggacacctcc 2701 taggactaca tgcac
С помощью программы JalView я построила выравнивание последовательностей кольцевых молекул ДНК из обеих находок. Вы можете посмотреть изображение выравнивания. По большей части длины хромосомы A4005 и A3216 схожи, но сильные различия начинаются именно в том месте, где находится ген. С 2520 нуклеотида по 2742 нуклеотид включительно последовательности существенно отличаются, поэтому мне кажется маловероятным, что эти нуклеотиды кодируют белок.
Считается, что данный ген кодирует субъединицу 7 НАДН-дегидрогеназы:
LQSVLAVFMA YRMTATTSQG IHAVEGPKGE LHISLTVTNS SMWRCRVRPA DLGHLLGLHA
Так как ген всего лишь один, то всю информацию о нём я разместила на этой странице в таблице 1.
| Таблица 1. Информация о гене из митохондрии Diplonema papillatum | |
| Таксономический идентификатор | 91374 |
| Название организма | Diplonema papillatum |
| Штамм | ATCC 50162 |
| Короткое название гена | nad7 |
| Полное название гена | - |
| Описание белка | NADH dehydrogenase subunit 7 |
| Расположение гена | ДНК митохондрии |
| Хромосома | A4005 |
| Идентификатор генома | JQ302962 (GenBank) |
| Координаты гена в хромосоме | 2534..2715 |
| Ориентация в геноме | plus |
| Идентификатор белка | AFW98722 (GenBank) |
| Классификация фермента | 1.6.5.3 |
| Дата секвенирования | 29 августа 2013 год |
| Идентификатор статьи | 23324603 (PubMed) |
| Название статьи | RNA-level unscrambling of fragmented genes in Diplonema mitochondria. |