Работа с нуклеотидным BLAST
Определение таксономии и функции прочтенной нуклеотидной последовательности из задания 4
Для выполнения задания я воспользовалась программой Standard Nucleotide BLAST, введя полученную последовательность в виде файла DNA.fasta. И получила следующий результат:
Из результата работы программы можно сделать следующие выводы:
- Для аннотации достаточно и 10 находок, так как все они указывают на один и тот же организм и один и тот же белок.
- Последовательность ДНК, с которой я работаю, принадлежит виду Modiolus modiolus (двустворчатый моллюск), который имеет систематику: Eukaryota; Metazoa; Lophotrochozoa; Mollusca; Bivalvia; Pteriomorphia; Mytiloida; Mytiloidea; Mytilidae; Modiolinae; Modiolus.
- Данная последовательность содержит ген "CO1" митохондриальной ДНК Modiolus modiolus, котрый кодирует субъединицу 1 цитохромоксидазы С (cytochrome c oxidase subunit 1).
В программе JalView я построила выравнивание моей последовательности ДНК (на рисунке выделена в рамку) с 10 находками BLAST. Cверху от неё - последовательности с геном CO1, кодирующим субъединицу 1 цитохромоксидазы С (cytochrome c oxidase subunit 1), с противоположной ориентацией в геноме; сснизу - последовательности с геном COI, кодирующим тоже субъединицу 1 цитохромоксидазы С (cytochrome c oxidase subunit 1), но имеющим положительную ориентацию в геноме.
Почему-то в названии находок с геном CO1 присутствует слово "isolate", а в находках с геном COI вместо этого слова написано "voucher". Я склонна думать, что это указывает на ориентацию гена в ДНК. Так как моя последовательность больше всего схожа с находкой Modiolus modiolus isolate Mo45 cytochrome c oxidase subunit 1 (CO1) gene, partial cds; mitochondrial (самая верхняя на изображении выравнивания), то я считаю правильным назвать исследуемый ген как CO1.
Сравнение списков находок нуклеотидной последовательности разными алгоритмами BLAST
Для последовательности ДНК из задания 4 я решила сравнить находки, полученные разными алгоритмами BLAST. Чтобы их было возможно адекватно оценивать, я для всех запусков программы указывала одинаковые параметры, отличные от тех, что указаны по умолчанию. Сходства искались среди организмов из одного семейства, но из других родов. На следующих двух изображениях представлены параметры для запуска программы BLASTN (Standard Nucleotide BLAST).
Ниже представлен результат работы программы BLASTN (Standard Nucleotide BLAST). Число находок 21. Этот алгоритм программы сравнивает мою нуклеотидную последовательность, которую я даю на вход, с нуклеотидными последовательностями из базы данных Nucleotide collection (nr/nt), строит локальные парные выравнивания и выводит выравнивания с наибольшим весом.
Следующие параметры я использовала при запуске программы BLASTX (Translated BLAST).
Ниже представлен результат работы программы BLASTX (Translated BLAST). Число находок 16. Этот алгоритм прграммы переводит мою нуклеотидную последовательность, которую я даю на вход, в 6 аминокислотных последовательностей и сравнивает их с аминокислотными последовательностями из базы данных Non-reductant protein sequences (nr). Затем программа также строит все возможные парные выравнивания и выдаёт находки с наибольшим весом выравнивания.
Следующие параметры я использовала при запуске программы TBLASTX (Translated BLAST)
Ниже представлен результат работы программы TBLASTX (Translated BLAST). Число находок 21. Этот алгоритм прграммы переводит мою нуклеотидную последовательность, которую я даю на вход, в 6 аминокислотных последовательностей и сравнивает их с аминокислотными последовательностями, полученными в результате трансляции нуклеотидных последовательностей из базы данных Nucleotide collection (nr/nt) в соответствующие белковые последовательности. Затем программа также строит все возможные парные выравнивания аминокислотных последовательностей и выдаёт находки с наибольшим весом выравнивания.
Из таблицы 1 видно, что алгоритмы BLASTN и TBLASTX выдали одинаковое число находок, причём списки их находок тоже совпадают. Отличается лишь порядок находок в этих списках, т.к. разные алгоритмы по-разному оценивают и строют выравнивания.
| Таблица 1. Число находок различными алгоритмами BLAST | |||
| BLASTN | BLASTX | TBLASTX | |
| Число находок | 21 | 16 | 21 |
Что же касается алгоритма BLASTX, то он выдал меньше находок. Ниже представлено изображение тех находок, которые данная программа не обнаружила.
Сравнение алгоритмов BLASTN, BLASTX и TBLASX демонстрирует, что в зависимости от того, какая у вас цель, вам слудует воспользоваться тем или иным алгоритмом BLAST. Для поиска схожих нуклеотидных последовательностей с целью определения таксономии и функции подойдёт алгоритм BLASTN. Если вы заранее знаете или предполагаете, что ваша нуклеотидная последовательность содержит ген, то вы можете воспользоваться алгоритмом BLASTX для определения названия кодируемого белка и положения гена в нуклеотидной последовательности. В данном случае желательно на вход подавать небольшую последовательность в формате FASTA, содержащую предполагаемый ген, т.е. для ДНК с большим количеством интронов и экзонов программа будет долго работать и вероятно выдаст ошибочные результаты. К тому же невозможно будет обнаружить участки, с которых синтезируются тРНК или рРНК. Алгоритм TBLASTX позволяет искать схожие нуклеотидные последовательности, как и BLASTN, но при этом участки с различными нуклеотидами будут считаться как одинаковые участки, если в результате мутаций они продолжили кодировать одинаковые аминокислоты. Следом идут последовательности, в которых мутации в геномах привели к замене аминокислот на другие аминокислоты, но со схожими функциями.
В моём случае тот факт, что алгоритм BLASTX выдал меньше находок, можно объяснить тем, что ненайденные пять находок в реальности кодируют белки сильно отличающиеся от гипотетических белков, которые может кодировать моя нуклеотидная последовательность.
Проверка наличия гомологов трех белков в геноме организма Amoeboaphelidium protococarum
В соответствии с заданием я решила проверить, присутствуют ли в геноме Amoeboaphelidium protococarum гены, кодирующие такие белки, как теломеразная обратная транскриптаза (Telomerase reverse transcriptase), митохондриальная цитратсинтаза (Citrate synthase, mitochondrial) и cубъединица RPB1 ДНК-зависимой РНК-полимеразы II (DNA-directed RNA polymerase II subunit RPB1).
Теломеразная обратная транскриптаза - Telomerase reverse transcriptase - TERT_HUMAN - O14746 (UniProt ID) - каталитическая субъединица фермента, который необходим для репликации терминальных концов хромосом у большинства эукариот. В нормальных соматических клетках он находится в практически неактивном состоянии. Каталитический комплекс удлинняет теломеры, действуя как обратная транскриптаза. Он добавляет простую нуклеотидную последовательность повторов к концам хромосом путём копирования шаблонной последовательности с теломеразной РНК компоненты фермента.
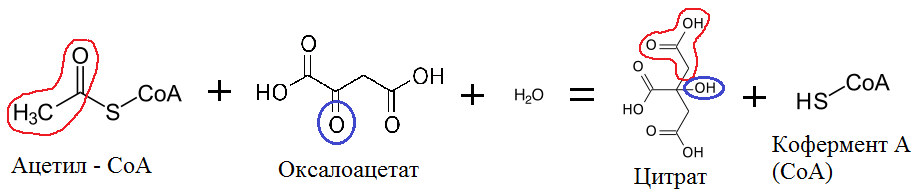
Митохондриальная цитратсинтаза - Citrate synthase, mitochondrial - CISY_HUMAN - O75390 (UniProt ID) - митохондриальный фермент, существующий почти во всех живых клетках и участвующий в первом шаге цитратного цикла, или цикла Кребса. Этот фермент кодируется в ядерной ДНК, синтезируется цитоплазматическими рибосомами, после чего отправляется в митохондриальный матрикс. Там он катализирует реакцию:
Субъединица RPB1 ДНК-зависимой РНК-полимеразы II - DNA-directed RNA polymerase II subunit RPB1 - RPB1_HUMAN - P24928 (UniProt ID) - самая большая субъединица РНК-полимеразы II, которая совместно с другими субъединицами полимеразы образует ДНК-связывающий домен полимеразы, в котором матрица ДНК транскрибируется в РНК. ДНК-зависимая РНК-полимераза II катализирует транскрипцию ДНК в РНК с использованием четырёх различных трифосфатов рибонуклеозидов в качестве субстратов. Таким образом в клетке синтезируются предшественники мРНК, малые ядерные РНК и множество малых некодирующих молекул РНК.
Последовательности выше указанных белков я скачала с сайта UniProt, записав их для удобства все в один файл.
Сначала я создала локальную базу данных, содержащую геном Amoeboaphelidium protococarum, записанный в файле X5.fasta:
makeblastdb -in X5.fasta -dbtype nucl
Затем я воспользовалась программой tblastn, которая находит гомологи белка в формальной трансляции нуклеотидного банка:
tblastn -query prots.fasta -db X5.fasta -out proteins.out
Для Теломеразной обратной транскриптазы (Telomerase reverse transcriptase) было обнаружено 3 находки:
Лучшая из них выглядит следующим образом:
> scaffold-17
Length=2125590
Score = 105 bits (263), Expect = 8e-23, Method: Compositional matrix adjust.
Identities = 151/568 (27%), Positives = 248/568 (44%), Gaps = 43/568 (8%)
Frame = +1
Query 452 LRQHSSPWQVYGFVRACLRRLVPPGLWGSRHNERRFLRN-TKKFISLGKHAKLSLQELTW 510
L +S P V FVR L ++ +G N F+ N ++L K +
Sbjct 610942 LNDYSRPHDVKQFVRCVLIKVFRCNFFGGMENLNAFVDNAVGMLLNLRKFESMPEASFIV 611121
Query 511 K-MSVRDCAWLRRSPGV--GCVPAAEHRLREEILAKFLHWLMSVYVVELLRSFFYVTETT 567
K + WLR V EH+ + + WL++ +V +LL++ F++T+T+
Sbjct 611122 KGIQSSRIMWLRSKLNT*PKVVNKLEHQKL**LCSSLFQWLLNRFVSDLLKACFFITDTS 611301
Query 568 FQKNRLFFYRKSVWSKLQSIGIRQHLKRVQLRELSEAEVRQHREARPALLTSRLRFIPKP 627
KNR+F+YR +W ++ + ++ +K L + + R+ S +R IPK
Sbjct 611302 HCKNRVFYYRFDLWRRM--VEVQSSIK--NLHPIDMG*INTGRK-----FMS*IRLIPKE 611454
Query 628 DG-LRPIVNMDYVVGARTFRREKRAERLTSRV-KALFSVLNYERARRPGLLGASVLGLDD 685
+G R I N+ R+ K L S L S NY LL VL DD
Sbjct 611455 NGSFRRINNL------RSVNNNK*MYGLLSDA*CILLSEKNYG*I---DLLKDIVLSNDD 611607
Query 686 IHRAWRTFVLRVRAQDPPPE-LYFVKVDVTGAYDTIPQDRLTEVIASIIKPQNTYCVRRY 744
I+ + F +R +A+ + LYFVK DVT AYD+I + +L V+ I + + + Y
Sbjct 611608 IYARLK*FKMRNKARF*RGD*LYFVKSDVT*AYDSINRQKLFSVLE*IF**DSEFIIHGY 611787
Query 745 AVVQKAAHGHVRKAFKSHVSTLTDLQPYMRQFVAHLQETSPLRDAVVIEQSSSLNEASSG 804
+ VS + +F L ++ + + V I++ + +
Sbjct 611788 *R*LQLCLLR*F*KLYHKVSIRAE*HQTFPEFCKELAKS--IANKVFIDKV**KKVSGAD 611961
Query 805 LFDVFLRFMCHHAVRIRGKSYVQCQGIPQGSILSTLLCSLCYGDME-NKLFAGIrrdgll 863
+F + + + ++ YVQ +GIPQGSI+S+LLCSL Y + N+LF RR L
Sbjct 611962 VFKAIEQLIYDNILQFEDGYYVQEEGIPQGSIVSSLLCSLLYSHLALNELFTFTRRSDSL 612141
Query 864 lrlvddfllVTPHLTHAKT----FLRTLVRGVPEYGCVVNLRKTVVNFPVEDEALGGTAF 919
L D L +LT K +L + G P+YG +N +KT N DE
Sbjct 612142 LIKFIDDFL---YLTFDKA*A*GYLSRI*IGFPDYGVHMNPKKTATNCLDMDEH------ 612294
Query 920 VQMPAHGLFPWCGLLLDTRTLEVQSDYSSYARTSIRASLTFNRGFKAGRNMRRKLFGVLR 979
P + +CG L+ L V D + Y +++ +L+ N G KL +R
Sbjct 612295 -SAPLQEV-SFCGHLIQVGDLSVSIDMNRYIGSNLSDALSVNYDKNPGDRAIDKLVQYVR 612468
Query 980 LKCHSLFLDLQVNSLQTVCTNIYKILLL 1007
K ++ D +N + TV NI++ +L+
Sbjct 612469 PKSLCMYFDCSLNGV*TVAMNIFENMLI 612552
Я бы назвала этот результат отрицательным, так как крайне малы проценты полных совпадений (Identities) и функциональных совпадений (Positives). К тому же при длине теломеразной обратной транскриптазы в 1132 аминокислотных остатка мы видим очень малый процент перекрывания (50.2 %). Хоть в данном выравнивании и присутствуют консервативные участки, но утверждать о гомологии из-за этого нельзя.
Для теломеразной обратной транскриптазы я построила множественное выравнивание, взяв последовательности белка из разных организмов. В результате я увидела, что данный белок не консервативен, а весьма вариабелен. Поэтому возможно, что scaffold-17 в действительности кодирует теломеразную обратную транскриптазу, только она в значительной степени отличается от таковой из организма человека. К тому же стоит отметить, что наиболее консервативные участки всё же присутствуют в нашем образце. Тогда такой результат стоит назвать условно положительным.
Для Митохондриальной цитратсинтазы (Citrate synthase, mitochondrial) было обнаружено 4 находки:
Лучшая из них выглядит следующим образом:
> scaffold-693
Length=1268102
Score = 565 bits (1457), Expect = 2e-180, Method: Compositional matrix adjust.
Identities = 262/377 (69%), Positives = 315/377 (84%), Gaps = 3/377 (1%)
Frame = +1
Query 90 GIRFRGFSIPECQKLLPKAKGGEEPLPEGLFWLLVTGHIPTEEQVSWLSKEWAKRAA-LP 148
GIRFRG +IPEC + LPKA GG EPLPEGLF+LL+TG +PT+EQV +S++WA RA+ LP
Sbjct 1243882 GIRFRGMTIPEC*EKLPKANGG*EPLPEGLFYLLLTGEVPTKEQVDEVSRDWANRASSLP 1244061
Query 149 SHVVTMLDNFPTNLHPMSQLSAAVTALNSESNFARAYAQGISRTKYWELIYEDSMDLIAK 208
HV ++D P LHPMSQ S AVTA+ +S FA+AY QG+ ++KYWE YEDSMDLIAK
Sbjct 1244062 KHVEDIID*CPVTLHPMSQFSIAVTAMQHDSKFAQAYQQGVHKSKYWEYAYEDSMDLIAK 1244241
Query 209 LPCVAAKIYRNLYREGSGIGAIDSNLDWSHNFTNMLGY-TDHQFTELTRLYLTIHSDHEG 267
LP VA++IYRN++++G + AID DWS+NF NMLG+ D QF EL RLYLTIHSDHEG
Sbjct 1244242 LPVVASRIYRNVFKDGK-VAAIDKTKDWSYNFANMLGFGKDAQFVELMRLYLTIHSDHEG 1244418
Query 268 GNVSAHTSHLVGSALSDPYLSFAAAMNGLAGPLHGLANQEVLVWLTQLQKEVGKDVSDEK 327
GNVSAHT+HLVGSALSDPYLSFAA +NGLAGPLHGLANQEVL W+ Q+++E+G +VSDE+
Sbjct 1244419 GNVSAHTTHLVGSALSDPYLSFAAGLNGLAGPLHGLANQEVLRWILQMKEEIGTNVSDEQ 1244598
Query 328 LRDYIWNTLNSGRVVPGYGHAVLRKTDPRYTCQREFALKHLPNDPMFKLVAQLYKIVPNV 387
+RDY W TL SG+V+PGYGHAVLRKTDPRYTCQREFALKHLP DP+FK+V+QLY IVPNV
Sbjct 1244599 VRDYCWKTLKSGQVIPGYGHAVLRKTDPRYTCQREFALKHLPTDPLFKMVSQLYNIVPNV 1244778
Query 388 LLEQGKAKNPWPNVDAHSGVLLQYYGMTEMNYYTVLFGVSRALGVLAQLIWSRALGFPLE 447
L EQGK KNP+PNVDAHSGVLLQ+Y + E +YTVLFGVSRALG L+QL+W RALG P+E
Sbjct 1244779 LTEQGKTKNPFPNVDAHSGVLLQHYNLKEQEFYTVLFGVSRALGCLSQLVWDRALGLPIE 1244958
Query 448 RPKSMSTEGLMKFVDSK 464
RPKS++T+ + K D K
Sbjct 1244959 RPKSLTTDTIKKMFDGK 1245009
Score = 74.3 bits (181), Expect = 7e-14, Method: Compositional matrix adjust.
Identities = 31/64 (48%), Positives = 51/64 (80%), Gaps = 0/64 (0%)
Frame = +3
Query 26 ASASSTNLKDILADLIPKEQARIKTFRQQHGKTVVGQITVDMMYGGMRGMKGLVYETSVL 85
++ ++T+LK+ L++L+P + +K R++HG+ +G +TVDM YGGMRG+KGL++ETS+L
Sbjct 1243623 STTTATSLKNRLSELVP*K*EEVKRVRKEHGEKSLGNVTVDMAYGGMRGIKGLIWETSLL 1243802
Query 86 DPDE 89
D DE
Sbjct 1243803 DADE 1243814
Находка, представленная выше, предполагает, что мРНК, с которой транслируется митохондриальная цитратсинтаза, образуется в результате сплайсинга. Проценты совпадений в выравнивании достаточно велики, чтобы можно было предположить гомологию. Известно, что активный центр этого фермента содержит 3 ключевых остатка: His301, His347 и Asp402. Из выравнивания видно, что все три аминокислотных остатка сохранили своё относительное расположение, к тому же они находятся в консервативном блоке. Всего митохондриальная цитратсинтаза содержит 466 аминокислотных остатка. Длина выравнивания составляет 441 аминокислотный остаток. Следовательно процент перекрывания равен 94.6 %. Из всего вышеуказанного можно сделать вывод, что данный результат положителен, и в геноме Amoeboaphelidium protococarum есть гомолог человеческой митохондриальной цитратсинтазы. Отсюда, видимо, можно предположить, что у данного организма скорее всего есть митохондрия.
Для Субъединицы RPB1 ДНК-зависимой РНК-полимеразы II (DNA-directed RNA polymerase II subunit RPB1) было обнаружено 5 находок:
Лучшая из них имеет следующие параметры:
> scaffold-300 Length=293202 Score = 1495 bits (3870), Expect = 0.0, Method: Compositional matrix adjust. Identities = 799/1547 (52%), Positives = 1079/1547 (70%), Gaps = 65/1547 (4%) Frame = +1
Субъединица RPB1 ДНК-зависимой РНК-полимеразы II состоит из 1970 нуклеотидных остатка. Длина выравнивания равна 1547 аминокислотных остатка. Следовательно процент перекрывания равен 78.5 %. Хотя проценты совпадений довольно велики, но в выравнивании содержится много гэпов. В данном белке ключевые позиции занимают остатки: Cys71, Cys74, Cys81, His84, Cys111, Cys114, Cys154, Cys184, Asp495, Asp497, Asp499. Из выравнивания видно, что ключевые остатки находятся в консервативных блоках. Причём остатки Cys71, Cys74, Cys81, His84 связываются с одним катионом цинка, остатки Cys111, Cys114, Cys154, Cys184 связываются с другим катионом цинка, остатки Asp495, Asp497 и Asp499 связываются с одним катионом магния и выполняют каталитическую функцию, помимо этого остатки Asp495 и Asp497 также связываются со вторым катионом магния совместно с субъединицей RPB2. Так как сайты, выполняющие каталитическую функцию, расположены в длинном консервативном блоке выравнивания, то я считаю, что данный результат положителен, то есть в геноме Amoeboaphelidium protococarum найден гомолог субъединицы RPB1 ДНК-зависимой РНК-полимеразы II с той же функцией. Однако, количество различий в выравнивании говорит о том, что гены, кодирующие данный белок разошлись очень давно, и с тех пор в них произошло много мутаций, но функция сохранилась прежней. Поэтому данный белок вариабелен.
Посмотреть изображение выравнивания.
Для подтверждения своей точки зрения я построила множественное выравнивание в JalView субъединиц RPB1 ДНК-зависимой РНК-полимеразы II из различных организмов. Участок, который сильно выделяется среди остальных последовательностей, скорее всего удаляется в ходе сплайсинга. Посмотреть изображение множественного выравнивания. Последовательность полученная из генома Amoeboaphelidium protococarum выделена красной рамочкой.
Поиск гена белка, закодированного в скэффолде Amoeboaphelidium protococarum
Для справки: cellular organisms; Eukaryota; Opisthokonta; Nucleariidae and Fonticula group; Aphelidea; Amoeboaphelidium; Amoeboaphelidium protococarum.
Сначала я получила информацию о длинах скэффолдов с помощью программы infoseq пакета EMBOSS:
infoseq X5.fasta -only -name -length
Я выбрала scaffold-514 длиной в 59281 пар нуклеотидов (пн). Затем с помощью команды seqret я получила последовательность нужного скэффолда:
seqret X5.fasta:scaffold-514 -out 514.fasta
Затем я запустила программу BLASTN с ограничением по таксону: Opisthokonta (taxid:33154), максимальным числом находок: 50 и порогом ожидания: 0.001. Остальные параметры использовались по умолчанию. В результате я получила следующие находки (на картинке представлены первые 20 находок):
Мы видим, что программа выдала много предсказанных и неохарактеризованных белков. Однако, все достоверные находки указывают на то, что в скэффолде-514 присутствует участок, с которого, возможно, траскрибируется мРНК, которая затем транслируется в бета-актин (beta actin). Лучшая такая находка:
>AF025305.1 Danio rerio beta actin mRNA, complete cds
Length=1143
Score = 1144 bits (619), Expect = 0.0
Identities = 961/1130 (85%), Gaps = 8/1130 (1%)
Strand=Plus/Minus
Query 9243 AGAAGCACTTTCTGTGCACAATAGATGGTCCAGACTCATCGTACTCCTGCTTGCTGATCC 9302
||||||| || | ||| || || |||||||||||||||||||||||||||||||| ||||
Sbjct 1132 AGAAGCATTTGCGGTGGACGATGGATGGTCCAGACTCATCGTACTCCTGCTTGCTAATCC 1073
Query 9303 ACATCTGTTGGAAGGTGGACAAGCTAGCCAGGATGGAGCCACCGATCCAGACAGAGTACT 9362
||||||| ||||||||||||| | |||||||||||||| ||||||||||||||||| |
Sbjct 1072 ACATCTGCTGGAAGGTGGACAGGGAGGCCAGGATGGAGCCTCCGATCCAGACAGAGTATT 1013
Query 9363 TACGCTCTGGAGGAGCAACAACCTTGATCTTCATGCTGCTAGGAGCCAGAGCAGTGATTT 9422
||||||| || || |||| | |||||||||||| ||||||| ||||| | ||||| |
Sbjct 1012 TACGCTCCGGTGGGGCAATGATCTTGATCTTCATTGTGCTAGGGGCCAGGGATGTGATCT 953
Query 9423 CCTTCTGCACTCTGTCAGCAATACCAGGGTACATGGTAGTACCACCAGACAAGACAATGT 9482
||||||||| ||||||||||| |||||||||||||| |||||||||||||| ||| |||
Sbjct 952 CCTTCTGCATCCTGTCAGCAATGCCAGGGTACATGGTGGTACCACCAGACAATACAGTGT 893
Query 9483 TAGCATACAGATCCTTACGAATATCAACATCACACTTCATGATGGAGTTGTAGGTAGTCT 9542
| |||||||| |||||||| || || || ||||||||||||||||||||| |||| ||||
Sbjct 892 TGGCATACAGGTCCTTACGGATGTCGACGTCACACTTCATGATGGAGTTGAAGGTGGTCT 833
Query 9543 CGTGGACACCAGC-AGCTTCCATACCCAAGAATGAAGGCTGGAACAGAGCTTCTGGGCAA 9601
|||||| ||| || || ||||||||||||||| || ||||||||||| || ||||||||
Sbjct 832 CGTGGATACC-GCAAGATTCCATACCCAAGAAGGATGGCTGGAACAGGGCCTCTGGGCAC 774
Query 9602 CGGAAACGCTCATTACCAATGGTGATGACTTGACCGTCAGGCAGCTCGTAACTCTTCTCC 9661
| ||| | |||||| |||||||||||||| || ||||||||||||||||| |||||||||
Sbjct 773 CTGAACCTCTCATTGCCAATGGTGATGACCTGTCCGTCAGGCAGCTCGTAGCTCTTCTCC 714
Query 9662 AGAGCAGAGGAGGTGGAAGCAGTC-TG--CATCTCCTACTCAAAGTCCAGAGCAACATAG 9718
|| | ||||||| |||||||| || |||||||| ||||||||| || || ||||||
Sbjct 713 AG-G--GAGGAGGAGGAAGCAGCGGTGCCCATCTCCTGCTCAAAGTCAAGGGCCACATAG 657
Query 9719 CACAGCTTCTCCTTGATGTCACGAACAATTTCACGCTCAGCAGTGGTAGTGAAAGAGTAA 9778
|| |||||||||||||||||||| |||||||| | |||||| ||||| ||||| |||
Sbjct 656 CAGAGCTTCTCCTTGATGTCACGGACAATTTCCCTCTCAGCTGTGGTGGTGAAGCTGTAG 597
Query 9779 CCACGCTCAGTCAGAATCTTCATCAGGTAGTCAGTCAAGTCACGGCCAGCCAGGTCCAGA 9838
|| | ||| ||||| |||||||| ||||||||||||| |||||||||||||| |||||||
Sbjct 596 CCTCTCTCGGTCAGGATCTTCATGAGGTAGTCAGTCAGGTCACGGCCAGCCAAGTCCAGA 537
Query 9839 CGCAGAATAGCGTGAGGCAGAGCATAACCTTCATAGATAGGCACAGTGTGGGAGACACCA 9898
|| || || || || ||||| || ||||| || ||||| ||||||||||||| |||||||
Sbjct 536 CGGAGGATGGCATGGGGCAGGGCGTAACCCTCGTAGATGGGCACAGTGTGGGTGACACCA 477
Query 9899 TCACCAGAGTCCAAGACAATACCAGTGGTACGACCGGAAGCATACAGAGACAGCACAGCC 9958
||||||||||||| || ||||||||||||||||| || |||||||| ||||||||||||
Sbjct 476 TCACCAGAGTCCATCACGATACCAGTGGTACGACCAGAGGCATACAGGGACAGCACAGCC 417
Query 9959 TAGATAGCAACGTAGAAGGCAGGCACATTGAAGGTCTCGAACATGATCTGAGTCATCTTC 10018
| ||| |||||||| | ||| || ||||||||||||||||||||||| ||||||||
Sbjct 416 TGGATGGCAACGTACATGGCGGGGGTGTTGAAGGTCTCGAACATGATCTGTGTCATCTTT 357
Query 10019 TCACGGTTAGCCTTTGGGTTCAGAGGAGCCTCAGTACACAGGACTGGGTGCTCTTCTGGA 10078
|| | ||| ||||| |||||||| || ||||| || |||||| |||||||| |||||
Sbjct 356 TCCCTGTTGGCCTTGGGGTTCAGGGGGGCCTCTGTGAGCAGGACGGGGTGCTCCTCTGGG 297
Query 10079 GCAACACGCAGCTCATTGTAAAAGGTATGGTGCCAGATCTTCTCCATGTCGTCCCAGTTG 10138
|||||||||||||||||||| ||||| || ||||||||||||||||| || |||||||||
Sbjct 296 GCAACACGCAGCTCATTGTAGAAGGTGTGATGCCAGATCTTCTCCATATCATCCCAGTTG 237
Query 10139 GTCACAATACCGTGCTCAATAGGATACTTCAGGGTGAGGATACCACGCTTGGACTGAGCC 10198
|| ||||||||||||||||| || ||||||||||| |||||||| | |||| |||||||
Sbjct 236 GTGACAATACCGTGCTCAATTGGGTACTTCAGGGTCAGGATACCTCTCTTGCTCTGAGCC 177
Query 10199 TCATCACCAACATAAGAGTCCTTCTGGCCCATACCAACCATGACACCTTGATGACGAGGA 10258
||||||||||| || ||| ||||| |||||||||||||||||||| ||||| | ||
Sbjct 176 TCATCACCAACGTAGCTGTCTTTCTGTCCCATACCAACCATGACACCCTGATGTCTGGGG 117
Query 10259 CGACCAACAATGGATGGGAAGACAGCTCTTGGAGCATCATCACCAGCAAAGCCAGCCTTG 10318
||||| || ||||||||||||||||||| ||||||||||| ||||| || || || |||
Sbjct 116 CGACCCACGATGGATGGGAAGACAGCTCGGGGAGCATCATCTCCAGCGAATCCGGCTTTG 57
Query 10319 CACATACCTGAACCGTTGTCAATAACCAATGCAGCAACTTCGTCTTCCAT 10368
|||||||| || |||||||||| ||||| ||| |||| ||| || |||||
Sbjct 56 CACATACCGGATCCGTTGTCAACAACCAGTGCGGCAATTTCATCATCCAT 7
При запуске программы BLASTX с теми же изменёнными параметрами мы получим следующие находки (на картинке представлены первые 20 находок):
Очень много гипотетических белков, но те, которые ими не являются, представляют собой аминокислотные последовательности белка фимбрина (fimbrin), участвующего в перекрёстном связывании актина и играющего важную роль в формировании филоподий. Как известно, Amoeboaphelidium protococarum в состоянии споры формируют филоподии, содержащие хорошо развитые актиновые микрофиламенты. К тому же фимбрин является высококонсервативным белком. Он управляет формированием плотно связанных актиновых филаментов, которые принимают участие в динамических процессах, включая цитокинез и вторжение в клетку хозяина. Лучшая находка:
>XP_004345335.1 fimbrin [Capsaspora owczarzaki ATCC 30864]
KJE96233.1 fimbrin [Capsaspora owczarzaki ATCC 30864]
Length=627
Score = 702 bits (1812), Expect = 0.0, Method: Compositional matrix adjust.
Identities = 364/619 (59%), Positives = 478/619 (77%), Gaps = 4/619 (1%)
Frame = -1
Query 3595 SKYFEVGRKFTQFKDSEIDLFIKQFQSFDKDGNGHIDKSELHKVCQELGEAISTEDLSKK 3416
S + KF +F +++ F++ F+S D DG+G ID +EL V + LGE + E++ +
Sbjct 2 SDFQAAASKFPEFSADDVEKFVQTFKSMDADGSGSIDAAELGAVLRSLGEKATPEEVRAQ 61
Query 3415 IAEVDQNNNNTVEFDEFLQVIAKIRERRVGSDKGFGALYQK*ARLVKMGGATEASAHSIN 3236
I EVD N + T+EF+EFL +I+++R + SD GFG + K +++V +GG++++ AHS +
Sbjct 62 IQEVDTNRSGTIEFNEFLGIISRLRAGKASSDAGFGKTFTKQSKVVTVGGSSDSIAHSFS 121
Query 3235 EDEHEQFVLHINAALKNDADV*NKLPINPLEFGDLYEKCKDGLVLCKLINDSVPDTIDER 3056
EDE E FV HIN L DAD+ + P++ + ++E KDGL+LCKLIN SVPDTIDER
Sbjct 122 EDEKESFVDHINMELGTDADIGKRFPLDSHDMS-IFEAVKDGLLLCKLINYSVPDTIDER 180
Query 3055 VLNKGNKLNTF*KTENNNVVVNSAKAIGCSVVNIGAQDliegreililgLVW*IIKIGLF 2876
VLN KLN F EN NV +NSAKAIGC+VVN+GAQDL+EGR L+LGL+W IIKIGL
Sbjct 181 VLNIKAKLNQFEIVENQNVCINSAKAIGCNVVNVGAQDLMEGRVHLVLGLIWQIIKIGLL 240
Query 2875 AKVDLKFHPELFRLLEQGETLDDLFKLPVD*ILLRWFNYHLKKAGWNRKVTNFTSDIKDS 2696
++++L HPEL+RLLE+GETLDDL KLPV+ IL+RW NYHLK AG +++ NF SDIKDS
Sbjct 241 SRINLSNHPELYRLLEEGETLDDLLKLPVEQILIRWVNYHLKNAGSKKRIANFGSDIKDS 300
Query 2695 ENYIVLLNQLEPSQCSRAALNEKDLKQRAE*MLVNADKLE--CRKYVTPKAIVEGNQKln 2522
E Y +LL+QL+P++C+ A LNE DL +RAE +L NADKL+ CRK+VTPKAIV GN KLN
Sbjct 301 EAYTILLSQLDPNRCTTAPLNESDLHKRAELVLQNADKLDPPCRKFVTPKAIVAGNPKLN 360
Query 2521 lafvanlfnNYPGLEPLTETEKAALDDWLFNSQGDREARAFALWLNSLNVDPFVNNLYED 2342
LAFVANLFN +PGL PL+E EKAA+D+ LF +GDREARAFALWLNSL ++PFVNNLYED
Sbjct 361 LAFVANLFNFHPGLAPLSEEEKAAIDEALFGGEGDREARAFALWLNSLGIEPFVNNLYED 420
Query 2341 LRDGIILL*AFDKVHPGCVEWKRVNKGKGL-SKFKAVENTNYVVELGKHFKYSLVGIQGA 2165
L+DG++LL AFDK+ PG V+W +VN+ + + SKFK +ENTNY + +GK K+SLVG+ G
Sbjct 421 LKDGLVLLRAFDKISPGSVQWSKVNQNQPITSKFKRLENTNYAIVVGKSLKFSLVGVGGQ 480
Query 2164 DIFDGNKKLTLAIVW*LMRDNVI*ILKSVAKDGKEVTEQDMVNWANSVPGRVGKKSSMSG 1985
DI DGNK LTLA+VW +MR +V+ ILKS++KDG++++E +MV WAN+ + G+ S M
Sbjct 481 DIEDGNKTLTLALVWQMMRFHVLSILKSISKDGRDISEDEMVAWANNTVKKGGRDSVMDS 540
Query 1984 FKDSSLKTSLFFLDVLAGIKKGIVDYNLVTAGDNDDDAKLNAKYAISIARKLGATIFVLP 1805
FKD L +S+FFLD++ GIKKGIV+Y++V AG +D + K NAKY+ISIARKLGA IFVLP
Sbjct 541 FKDPKLASSIFFLDLMNGIKKGIVNYDIVAAGSDDAERKSNAKYSISIARKLGACIFVLP 600
Query 1804 EDLMEVKPKMILTFVGALM 1748
ED+MEVKPKMILTFVGALM
Sbjct 601 EDIMEVKPKMILTFVGALM 619
Число совпадений и процент перекрывания позволяют мне предположить, что в скэффолде-514 есть ген, кодирующий фимбрин.
Я проделала те же операции для скэффолда-698, и с помощью команды BLASTX мне удалось обнаружить ген, кодирующий субъединицу гамма эукариотического трансляционного фактора инициации 2 (eukaryotic translation initiation factor 2 subunit gamma (eIF2-γ)). Эта субъединица совместно с субъединицами eIF2-α и eIF2-β образует эукариотический трансляционный фактор инициации 2 (eIF2), который стабилизирует формирование функциональной рибосомы рядом со старт-кодоном. Он связывается с гуанозин трифосфатом (GTP) и отвечает за доставку заряженной метионином инициирующей тРНК к P-сайту пред-инициирующего комплекса. Как только инициирующая тРНК свяжется со старт-кодоном AUG в P-сайте, так трансляционный фактор инициации 2 гидролизирует ГТФ (GTP) до ГДФ (GDP) и сам диссоциирует. Это позволяет большой субъединице рибосомы связаться и начать элонгацию (удлинение полипептидной цепи). Лучшая находка:
>XP_016608020.1 eukaryotic translation initiation factor 2 subunit gamma [Spizellomyces
punctatus DAOM BR117]
KNC99980.1 eukaryotic translation initiation factor 2 subunit gamma [Spizellomyces
punctatus DAOM BR117]
Length=452
Score = 676 bits (1743), Expect = 0.0, Method: Compositional matrix adjust.
Identities = 339/448 (76%), Positives = 382/448 (85%), Gaps = 4/448 (1%)
Frame = -1
Query 2799 VNVDVSKLTPLSPEVISN*ATINIGTIGHVAHGKSTVVKAISGV*TVRHKTELERNITIK 2620
+++DV++L PLSPEVIS ATINIGTIGHVAHGKSTVVKAISGV TVR K ELERNITIK
Sbjct 4 IDIDVTQLNPLSPEVISRQATINIGTIGHVAHGKSTVVKAISGVQTVRFKNELERNITIK 63
Query 2619 LGYANAKIYECDQ--CPRPLKYKSFRSDKEVQFKCEEPNCGGTMHLKRHVSFVDCPGHDI 2446
LGYANAKI++CD CPRP Y+S+RSDKE F C+ C G M L RHVSFVDCPGHDI
Sbjct 64 LGYANAKIFKCDSEACPRPGCYRSYRSDKEEGFPCDRVGCMGRMRLLRHVSFVDCPGHDI 123
Query 2445 LMATMLNGAAVMDAAMLLIAGNESCP*P*TSEHLAAIEIMKLNHVIILQNKIDLISE**A 2266
LMATMLNGAAVMDAA+LLIAGNESCP P TSEHLAAIEIMKL H++ILQNK+DLI E A
Sbjct 124 LMATMLNGAAVMDAALLLIAGNESCPQPQTSEHLAAIEIMKLQHILILQNKVDLIKESAA 183
Query 2265 AE*YKSILKFINGTVAQKAPIVPISA*MKYNIDAVNEYICKKVPVPVRDFTAPARLIVIR 2086
E ++SILKF+ GTVA API+PISA +KYNIDA+NEYI KK+P+P+RDFTA RLIVIR
Sbjct 184 EEHHESILKFVKGTVADSAPIIPISAQLKYNIDAINEYIVKKIPIPIRDFTADPRLIVIR 243
Query 2085 SFDVNKPGTEY*DLKGGVAGGSIL*GCLKLGDEIEVRPGIVSKDSEGKFRVRPILSRIVS 1906
SFDVNKPG E DLKGGVAGGSIL G LK+GDEIEVRPGIVSKDS+GK R +PI SRIV+
Sbjct 244 SFDVNKPGAEVADLKGGVAGGSILCGVLKIGDEIEVRPGIVSKDSDGKVRCKPIYSRIVT 303
Query 1905 LHTE*NHLQFAVPGGLIGVGTQIDPTVCRSDRLVGQVLGARGTLPEIYTELEITYFLLRQ 1726
L E N L+FAVPGGLIGVGT+IDPT+CR+DRLVGQVLGA G LP IYTELEI YFLLR+
Sbjct 304 LLAETNELKFAVPGGLIGVGTRIDPTLCRADRLVGQVLGAVGKLPAIYTELEINYFLLRR 363
Query 1725 LLGVRSTTENK*LTKVTKLAKNETLMVNIGSTTAGCKVLNVKADMAKIYLTVPCCTEIGE 1546
LLGV+S E+K TKV KL+++E LMVNIGST+ G +VL+VKADMAKI L P CTEIGE
Sbjct 364 LLGVKS--EDKKQTKVQKLSRHEVLMVNIGSTSTGGRVLSVKADMAKILLNTPACTEIGE 421
Query 1545 KVALSRRIDRHWRLIGWGKITRGVTIEP 1462
K+ALSRRI++HWRLIGWG I RGVTIEP
Sbjct 422 KIALSRRIEKHWRLIGWGTIRRGVTIEP 449
Таким образом, в скэффолде-514 я предполагаю наличие гена, кодирующего бета-актин, и гена, кодирующего фимбрин. А для скэффолда-698 я предполагаю наличие гена, кодирующего субъединицу гамма эукариотического трансляционного фактора инициации 2 (eIF2-γ).