EMBOSS. Выравнивание геномов
Упражнения с пакетом программ EMBOSS
- (seqret) Несколько файлов в формате fasta собрать в единый файл
Исходные данные: tubulin.fasta и actin.fasta.
Команда:
seqret "*.fasta" both.fasta
Результат: both.fasta.
Исходные данные: both.fasta.
Команда:
seqretsplit both.fasta
Результат: am501548.1.fasta и a18572.1.fasta.
Исходные данные: both.fasta.
Команда:
transeq -table 0 both.fasta proteins.fasta
Результат: proteins.fasta.
Исходные данные: tubulin.fasta.
Команда:
transeq -frame 6 tubulin.fasta proteins6.fasta
Результат: proteins6.fasta.
Исходные данные: align.fasta.
Команда:
seqret align.fasta msf::align.msf
Результат: align.msf.
Сравнение геномов
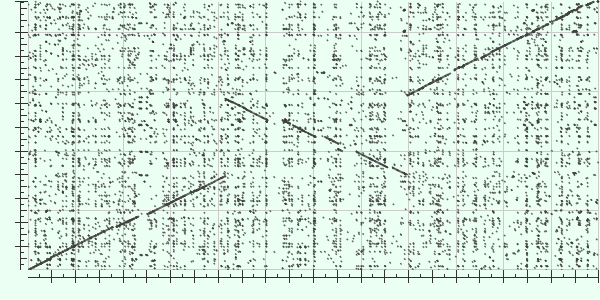
С помощью программы blast2seq алгоритма BLASTN я построила карту локального сходства бактерий Bifidobacterium longum subsp. infantis штамм 157F в качестве query (ось X) и Bifidobacterium longum штамм 35624 в качестве subject (ось Y).
Bifidobacterium longum - Грам-положительные палочковидные бактерии, живущие в желудочно-кишечном тракте человека. Они синтезируют молочную кислоту, которая, как пологается, предотвращает рост болезнетворных организмов. Сами же бактерии не являются патогенными, и их часто добавляют в продукты питания. Данный вид включает следующие подвиды: Bifidobacterium longum subsp. infantis, Bifidobacterium longum subsp. longum и Bifidobacterium longum subsp. suis. Штамм взятый в качестве запроса (35624), как правило, тоже относят к подвиду infantis, но даже из карты локального сходства мы видим, что их разница значительна. Ниже представлено изображение Bifidobacterium longum BB536.
В таблице 1 представлены параметры построенного геномного выравнивания.
| Таблица 1. Параметры парного выравнивания геномов в blast2seq | ||||
| Max score | Total score | Query cover | E-value | Ident |
| 1.910e+05 | 4.330e+06 | 86% | 0.0 | 99% |
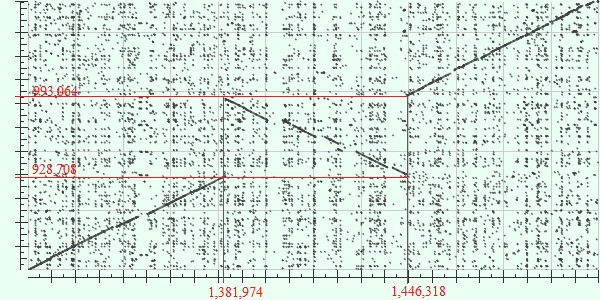
Стоит отметить, что геном Bifidobacterium longum subsp. infantis штамма 157F состоит из 2,400,312 пн, а геном Bifidobacterium longum штамма 35624 состоит из 2,264,056 пн.
На примере полученной карты локального сходства опишем крупные эволюционные события. Их можно разбить на дв группы: инверсии и вставки (делеции).
1) Инверсии: На изображении карты я выделила красным цветом инверсию. Цифры указывают номера нуклеотидов в последовательностях геномов. Я предполагаю, что первичной была именно инверсия, после которой уже произошли вставки (делеции), из-за чего вместо прямой зеркально отражённой линии на выделенном участке мы видим разрывную линию.
2) Вставки (делеции): На следующем изображении я синим цветом отметила наиболее крупные делеции по оси Y (соответственно, вставки по оси X). А красным цветом отмечена единственная делеция по оси X (вставка по оси Y).
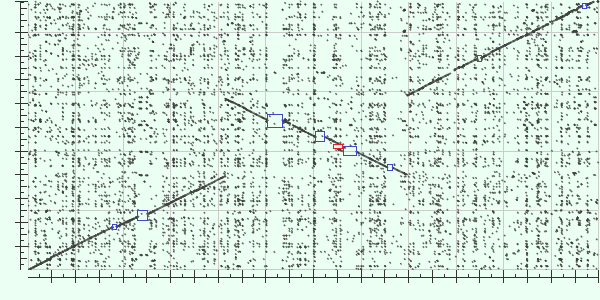
Для поиска гомологов выбранных бактерий с полной сборкой генома я использовала следующий запрос в базе данных Nucleotide:
"Bifidobacterium longum"[Organism] AND "complete genome"[TITLE]