Укоренение в среднюю точку
Для бактерий из предыдущего задания я реконструировала филогенетическое дерево методом "Neighbor Joining Using % Identity" по выравниванию последовательностей белка энолазы - фермента, катализирующего предпоследний, девятый этап гликолиза. Вы можете ознакомиться с моим проектом JalView или отдельно с выравниванием. Ниже представлено изображение полученного дерева с помошью программы MEGA.
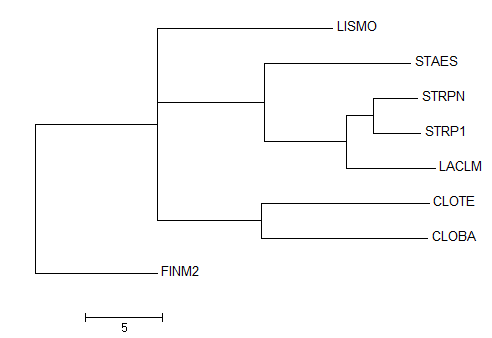
Его скобочная формула.
Далее я воспользовалась программой retree пакета PHYLIP. Я выполнила укоренение в среднюю точку (опция "Midpoint root the tree") и получила файл outtree со скобочной формулой. Затем с помощью программы MEGA я получила изображение дерева, укорененного в среднюю точку, представленное ниже.
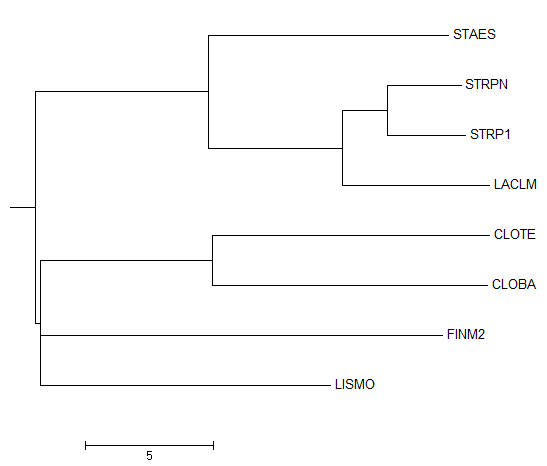
В результате мы получили неразрешенное дерево (есть узел, из которого выходит более двух ветвей). Его существенным отличием от эталонного является отсутствие нетривиальной ветви:
(CLOBA, CLOTE, FINM2, STRP1, STRPN, LACLM) vs (LISMO, STAES).
Это весьма существенно, так как бактерии с мнемониками LISMO и STAES относятся к одной таксономической группе - порядку Bacillales. Поэтому данное укоренение является ошибочным.
Также хочу отметить, что все ветви на изображении имеют разную длину. Дело в том, что метод "Neighbor-Joining" реконструирует длины ветвей в соответствии с принимаемой на вход матрицей расстояний между таксонами, которая строится, исходя из числа несовпадений в парных выравниваниях. Таким образом, длины ветвей пропорциональны числу произошедших мутаций. Метод "Neighbor-Joining" отвергает гипотезу о "молекулярных часах".