Построение дерева по нуклеотидным последовательностям и дерева с паралогами и ортологами
Построение дерева по нуклеотидным последовательностям
Для бактерий из предыдущих заданий я добыла последовательности 16S рибосомальной РНК из базы полных геномов NCBI. В папке с соответствующим таксономическим названием бактерии расположен файл с расширением .frn, в котором находится перечень последовательностей рРНК в fasta формате. Довольно часто в таком файле содержится более одной 16S рРНК, в таком случае я стремилась выбрать наиболее распространенный вариант. Таким образом, я сформировала файл 16S_rRNAs.fasta, в котором собрала восемь последовательностей 16S рРНК из геномов выбранных ранее бактерий.
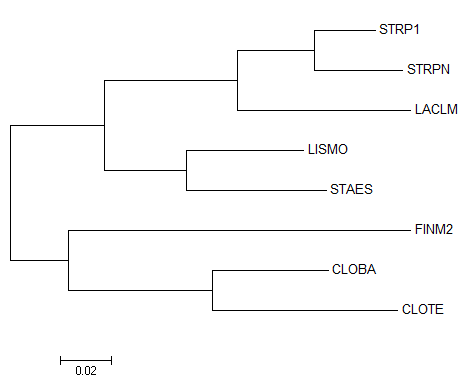
Далее с помощью сервера Musccle я построила множественное выравнивание 16S_rRNAs_align.fasta. Затем с помощью программы MEGA я построила филогенетическое дерево методом Maximum Likelihood (максимального правдоподобия). Ниже представлено его изображение.
Далее мне стало интересно получить укорененное дерево. Для этого я добавила к файлу 16S_rRNAs.fasta последовательность 16S рРНК бактерии Escherichia coli в качестве внешней группы, сделала выравнивание и построила дерево тем же методом Maximum Likelihood (максимального правдоподобия). В новом дереве я выделила поддерево, не содержащее только тривиальную ветвь E. coli. В результате я получила следующее изображение:
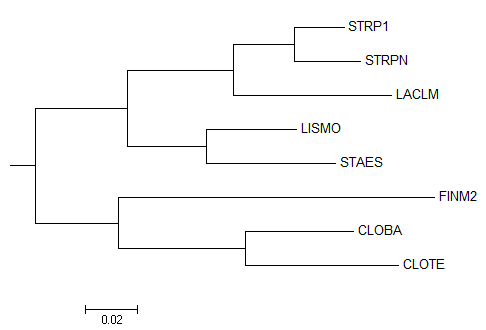
Я вполне довольна результатом, потому что построенное дерево по топологии и укоренению в точности совпадает с эталонным. Такие же результаты я получила при построении деревьев методами Neighbor-Joining и Minimum Evolution (минимальной эволюции).
Построение и анализ дерева, содержащего паралоги
Cначала нужно было найти в протеомах выбранных бактерий всех возможных гомологов белка CLPX_BACSU. Для этого я сохранила к себе файл CLPX_BACSU.fasta и создала файл proteomes.fasta, в котором собрала протеомы всех восьми бактеий. Далее я создала базу данных для blastp:
makeblastdb -in proteomes.fasta -out database.fasta -dbtype prot
Затем я провела поиск гомологов по новой базе данных database.fasta программой blastp, установив порог на E-value 0,001:
blastp -query CLPX_BACSU.fasta -db database.fasta -evalue 0.001 -out homologues.txt
На выходе я получила файл homologues.txt со списком из 36 белков - гомологов CLPX_BACSU. По полученным мнемоникам белков я создала файл proteins.fasta c их последовательностями. Затем с помощью сервера Musccle я построила выравнивание этих белков proteins_aln.fasta. После чего с помощью программы MEGA по методу Maximum Likelihood я построила следующее дерево:
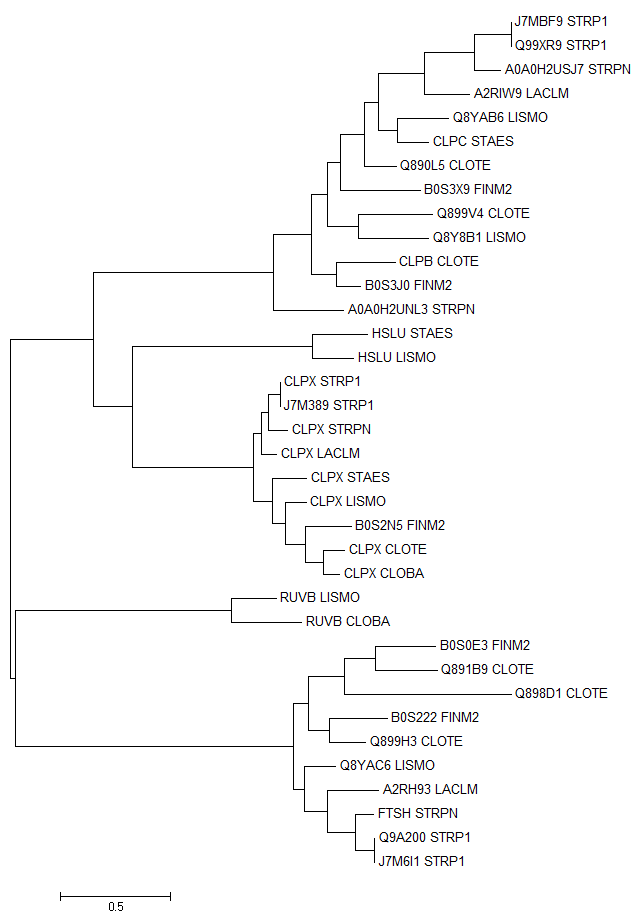
При детальном анализе белки J7MBF9_STRP1 и Q99XR9_STRP1 оказались идентичными друг другу. Они взяты из разных штаммов одного вида и по ошибке названы по-разному в базе данных Uniprot, хотя их аминокислотные последовательности полностью совпадают. Аналогичная ситуация с белками J7M389_STRP1 и CLPX_STRP1, и Q9A200_STRP1 и J7M6I1_STRP1. Поэтому я решила удалить по одному варианту и заново построить дерево:
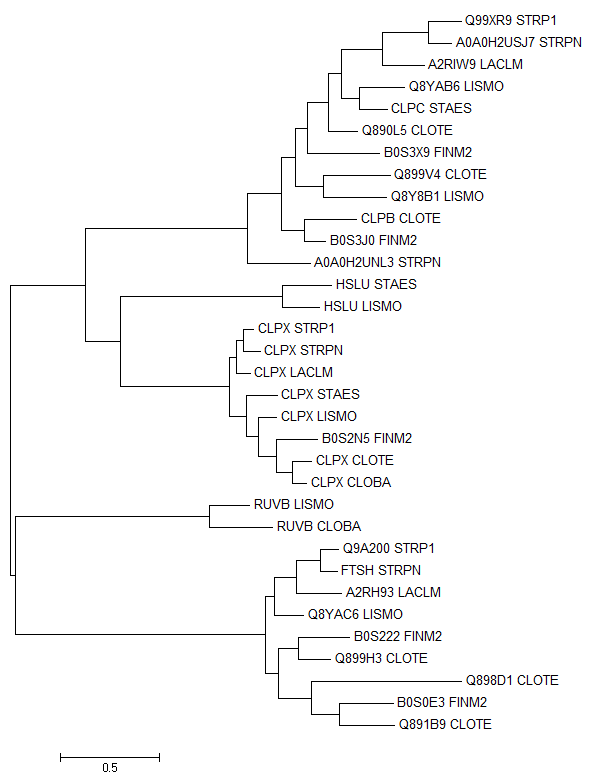
Теперь приступим к анализу полученного дерева:
Два гомологичных белка будем называть ортологами, если они из разных организмов и разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования.
Два гомологичных белка из одного организма, образовавшихся в результате дупликации гена, будем называть паралогами.
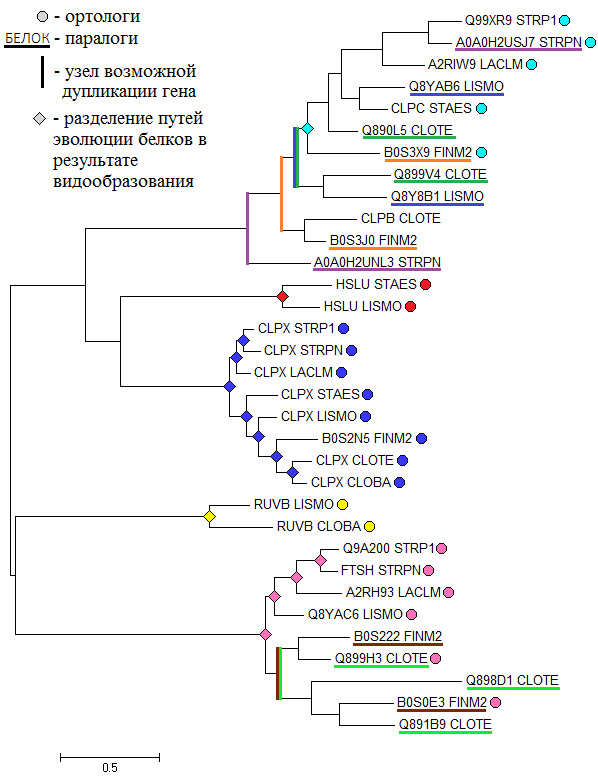
Полагая, что дерево реконструировано верно, ортологами можно назвать белки:
- CLPX_STRP1, CLPX_STRPN, CLPX_LACLM, CLPX_STAES, CLPX_LISMO, B0S2N5_FINM2, CLPX_CLOTE, CLPX_CLOBA - ATP-dependent Clp protease ATP-binding subunit ClpX (АТФ-связывающая субъединица ClpX из АТФ-зависимой Clp-протеазы)
- HSLU_STAES и HSLU_LISMO - ATP-dependent protease ATPase subunit HslU (АТФазная субъединица HslU из АТФ-зависимой протеазы)
- A0A0H2USJ7_STRPN, A2RIW9_LACLM, CLPC_STAES, Q99XR9_STRP1 и B0S3X9_FINM2 - ATP-dependent Clp protease ATP-binding subunit (АТФ-связывающая субъединица из АТФ-зависимой Clp-протеазы)
- B0S0E3_FINM2, Q9A200_STRP1, Q899H3_CLOTE, FTSH_STRPN, Q8YAC6_LISMO и A2RH93_LACLM - ATP-dependent zinc metalloprotease FtsH (АТФ-зависимая цинк металлопротеаза FtsH)
- RUVB_CLOBA и RUVB_LISMO - Holliday junction ATP-dependent DNA helicase RuvB (АТФ-зависимая ДНК хеликаза RuvB для структур Холлидэя)
А паралогами можно назвать белки:
- Q890L5_CLOTE и Q899V4_CLOTE - Negative regulator of genetic competence mecB/clpC (отрицательный регулятор генетической компетентности, способности клеток к трансформации)
- Q899H3_CLOTE, Q891B9_CLOTE и Q898D1_CLOTE - ATP-dependent zinc metalloprotease (АТФ-зависимая цинк металлопротеаза)
- B0S0E3_FINM2 и B0S222_FINM2 - ATP-dependent zinc metalloprotease FtsH (АТФ-зависимая цинк металлопротеаза)
- B0S3X9_FINM2 и B0S3J0_FINM2 - аннотированы как разные белки (ATP-dependent protease Clp ATP-binding subunit ClpC и Chaperone protein ClpB соответственно), но их парное выравнивание имеет большой процент совпадений. Поэтому можно предположить, что данные белки являются давно разошедшими паралогами. Интересно, что белок B0S3J0_FINM2 длиннее белка B0S3X9_FINM2 на 55 аминокислотных остатка, которые образуют петли во вторичной структуре, не относящиеся к каталитическим центрам.
- Q8Y8B1_LISMO и Q8YAB6_LISMO - также аннотированы как разные белки (ATP-dependent protease и Endopeptidase Clp ATP-binding chain C), но имеют хорошее парное выравнивание.
- A0A0H2USJ7_STRPN и A0A0H2UNL3_STRPN - ATP-dependent Clp protease, ATP-binding subunit (АТФ-связывающая субъединица из АТФ-зависимой Clp-протеазы)
Ниже представлено изображение дерева, на котором отмечены паралоги, ортологи, а также обозначены такие возможные эволюционные события, как дупликация гена и разделение путей эволюции белков в результате видообразования.