Ферменты и метаболические пути. База данных KEGG
Цель данного задания состоит в том, чтобы проверить, являются ли члены разных ортологических рядов из базы данных KEGG гомологичными белками, и проанализировать их филогенетические отношения.
Выбор пары ортологических рядов
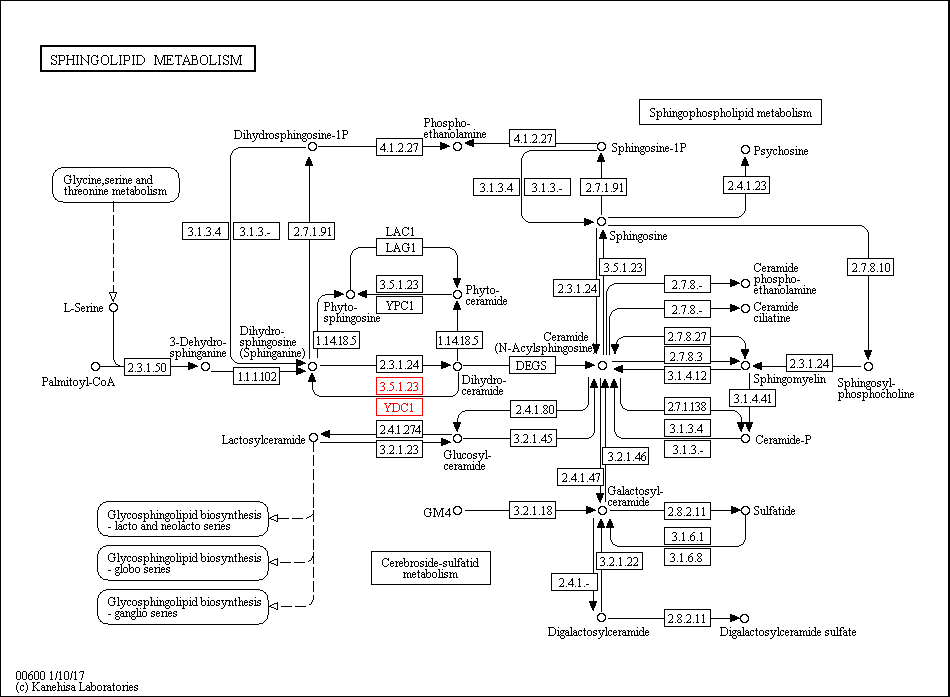
Для работы я выбрала метаболизм сфинголипидов (Sphingolipid metabolism), а в нем выбрала реакцию R06518 (EC:3.5.1.23). Ниже представлено изображение карты метаболизма сфинголипидов, на которой красным цветом выделена выбранная реакция.
На следующем изображении вы можете видеть выбранную реакцию, в которой N-ацетилсфинганин гидролизуется до жирной кислоты и сфинганина (дигидросфингозина).
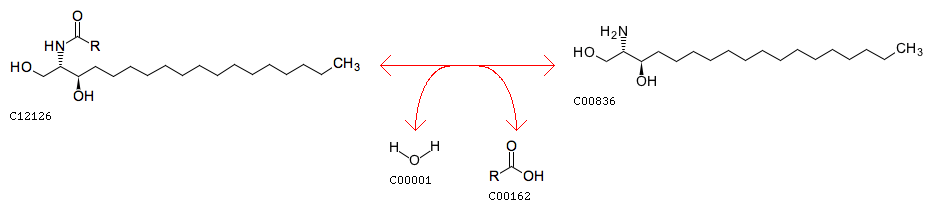
N-Acylsphinganine + H2O <=> Fatty acid + Sphinganine
Данная реакция катализируется двумя ортологическими рядами, информация о которых приведена в таблице 1.
| Таблица 1. Информация об ортологических рядах | ||||
| Идентификатор | Название | Описание | Число белковых последовательностей | Число генов |
| K12348 | ASAH1 | acid ceramidase | 65 (UniProt 58) | 261 |
| K12349 | ASAH2 | neutral ceramidase | 310 (UniProt 300) | 2099 |
Хоть в ортологическом ряде K12349 число белковых последовательностей больше 200, но я решила попробовать поработать с выбранными рядами, так как в сумме я имею число белков меньшее 400.
Получение совместного множественного выравнивания
На странице с описанием ортологического ряда K12348 я нажала на кнопку "UniProt" в разделе "Genes". Появилась таблица с перечнем идентификаторов белков в базе данных UniProt, относящихся к указанному ортологическому ряду. Далее я скопировала эту таблицу в программу Microsoft Excel. Затем я скопировала второй столбец таблицы в буфер обмена, открыла сайт UniProt, выбрала в верхнем меню "Retrieve/ID mapping", вставила второй столбик моей таблицы в поле "1. Provide your identifiers", а в поле "2. Select options" задала параметры "From: UniProtKB AC/ID" и "To: UniProtKB" и нажала на кнопку "Go". Сервер выдал мне таблицу со всеми нужными белковыми последовательностями, я выделила все и сохранила в файл K12348.fasta. Аналогичным образом я получила файл K12349.fasta.
Затем полученные файлы я обработала соответствующими скриптами fasta8.py и fasta9.py. В результате я получила файлы K12348.fasta и K12349.fasta соответственно.
Затем я подала эти файлы, заранее объединив в один файл input.fasta, серверу Musccle, который построил мне множественное выравнивание align_KO_0.fasta. Полученное выравнивание я открыла в программе JalView и раскрасила по Clustalx. Результат представлен в файле проекта align_KO_0.jvp.
Проверка гомологичности белков в выравнивании
Далее я удалила из выравнивания белки, которые очень плохо выровнены с остальными: I4B0X9_TURPD|9, R7W9Q8_AEGTA|9, Q5B5D5_EMENI|9, A0A0D1EC83_USTMA|9, F4PKN3_DICFS|8, G4YVT8_PHYSP|9, C3XXG7_BRAFL|8, V4AZF3_LOTGI|8, G4ZWQ2_PHYSP|9, A0A061HE08_9BASI|9, C1E7W0_MICCC|8, Q22AK2_TETTS|8, YQV_CAEEL|8, F4R3F3_MELLP|9, D7MR59_ARALL|9, F4KHQ8_ARATH|9, A0A087SGW8_AUXPR|9, F6EGG1_HOYSD|9, A6R789_AJECN|9, D5URW8_TSUPD|9, A0A0G2ZZK0_9DELT|9, A0A0G3A862_9DELT|9, A0A127PXU1_9BURK|9, M8BP98_AEGTA|9, E1ZPU0_CHLVA|9, A0A0H5PBC5_NOCFR|9 и др.
Далее я удалила из выравнивания короткие белки, которые содержат много гэпов в тех участках, где в других последовательностях выравнивания консервативные колонки: A0A109SKZ1_9MYCO|9, A0A1D6LTQ1_MAIZE|9, R7WEE3_AEGTA|9, B9HP93_POPTR|9, R0GV69_9BRAS|9, D7LFF4_ARALL|9, I0YQP2_COCSC|9, B9H5E5_POPTR|9, B7QN43_IXOSC|9, E0W4J0_PEDHC|9, T1FZQ7_HELRO|8, Q4TG63_TETNG|8, G4ZWQ4_PHYSP|9, D0N5N6_PHYIT|9, D0N5N0_PHYIT|9, G6DRM9_DANPL|9, B0XDM1_CULQU|9, G4VN86_SCHMA|9, B4GNM1_DROPE|9, B8NH00_ASPFN|9, A0A109SKZ3_9MYCO|9, C3XYV2_BRAFL|9, E9EH73_METAQ|9, M2MZQ8_BAUCO|9, G0SDM7_CHATD|9, K5W3P0_PHACS|9, S7RLR7_GLOTA|9, B0DCM2_LACBS|9, B7FYD1_PHATC|9, A9RDV4_PHYPA|9, N1R3N3_AEGTA|9, A8J8C7_CHLRE|9, D8U3H2_VOLCA|9 и др.
В результате я получила подкорректированное выравнивание align_KO_2.fasta. Результат представлен в файле проекта align_KO_2.jvp.
Гомологичность белков в выравнивании. Как я ни старалась удалять "плохие" последовательности из файла проекта, их все равно осталось так много, что программа отказывается экспортировать мне изображение выравнивания. Однако если открыть файл проекта, то будет видно, что во многих консервативных областях последовательности из ортологического ряда K12348 сильно отличаются от последовательностей ортологического ряда K12349 (обозначения |8 и |9 в названиях соответственно). Поэтому я считаю неуместным вообще говорить о множественном выравнивании. Если выделить белки ряда K12348 в отдельную группу и окрасить независимо от остальных, то мы увидим много совпадений. Аналогично будет для белков ряда K12349. Что доказывает мою точку зрения. Хочу заметить, что были в изначальном наборе белков, такие которые были схожи и с белками K12348 и с белками K12349. Однако их было мало и они были удалены, так как в целом плохо выравнивались с обеими группами.
Построение филогенетического дерева
Я считаю, что в данном случае нельзя строить дерево, так как оно не будет достоверно отражать филогению выбранных ортологических рядов.