Геномное окружение. База данных Gene Ontology (GO)
Получение информации о КОГе белка
База данных COGs (clusters of orthologous groups) была создана благодаря сравнению предсказанных и известных белков, взятых из полных геномов, чтобы выделить группы ортологов. Она используется для филогенетической классификации новых белков на основании сходства их последовательностей с тем или иным КОГом. Каждый КОГ (кластер ортологических групп) содержит белки или группы паралогов из хотя бы трех линий, выполняющих одну и ту же функцию в клетке. КОГи относятся к одной или нескольким (реже) крупным функциональным категориям, информацию о которых можно найти на сайте.
База данных Conserved Domain Database (CDD) состоит из коллекции хорошо аннотированных шаблонов множественных выравниваний для древних доменов и белков в полную длину. Сервис CDD позволяет быстро аннотировать белки через RPS-BLAST, определяя консервативные домены в введенной последовательности белка.
Для работы возьмем белок пептидогликан гликозилтрансферазу с идентификатором YP_003180359.1 (сейчас WP_012809424.1 в базе данных RefSeq и C8W8H7_ATOPD в UniProt) из генома бактерии Atopobium parvulum.
Я открыла сервис CDD, ввела в поле последовательность белка в fasta формате, взятую по ссылке из UniProt, и нажала на кнопку "Submit". В разделе "Full results" я получила следующий список хитов ("успехов").
В колонке "Accession" оказалось 5 КОГов, информация о которых приведена в таблице 1.
| Таблица 1. Информация о КОГах белка пептидогликан гликозилтрансферазы | ||||
| Идентификатор | E-value | Координаты | Функциональная категория |
Название КОГа |
| COG0772 | 6.62e-75 | 61-420 | D | Bacterial cell division protein FtsW, lipid II flippase бактериальный белок клеточного деления FtsW, флиппаза на липид II * |
| COG0768 | 6.09e-72 | 471-951 | DM | Cell division protein FtsI penicillin-binding protein 2 белок клеточного деления FtsI пенициллин-связывающий белок 2 |
| COG0744 | 1.08e-32 | 595-928 | M | Membrane carboxypeptidase (penicillin-binding protein) мембранная карбоксипептидаза (пенициллин-связывающий белок) |
| COG5009 | 4.84e-26 | 631-940 | M | Membrane carboxypeptidase penicillin-binding protein мембранная карбоксипептидаза пенициллин-связывающий белок |
| COG4953 | 6.33e-08 | 552-893 | M | Membrane carboxypeptidase penicillin-binding protein PbpC мембранная карбоксипептидаза пенициллин-связывающий белок PbpC |
Замечание. Данные о функциональной категории я взяла из последнего релиза базы данных.
* Флиппазы - трансмембранные белки, способствующие переносу молекул фосфолипидов между двумя сторонами липидного бислоя клеточной мембраны. Данная флиппаза переносит молекулы липида II из клетки через внутреннюю мембрану. После чего трансгликозилазы полимеризуют молекулы липида II с образованием линейной гликановой (полисахаридной) цепи.
Категория D - Cell cycle control, cell division, chromosome partitioning (контроль клеточного цикла, клеточное деление, разделение хромосом).
Категория M - Cell wall/membrane/envelope biogenesis (биогенез клеточной стенки/мембраны/оболочки).
Визуализация геномного окружения
База данных Search Tool for the Retrieval of Interacting Genes/Proteins (STRING) - биоинформатический ресурс, используемый для поиска и предсказания белок-белковых взаимодействий. Особенно он бывает полезен для работы с белками, не изученными экспериментально.
Я получила изображение белок-белковых взаимодействий для КОГа с наилучшим E-value (COG0772) с помощью сервиса STRING. На главной странице я нажала кнопку "Search", после чего в левом меню перешла в раздел "Protein families ("COGs")", в появившемся поле "Protein Family Name" я ввела идентификатор COG0772 и нажала на кнопку "Search". Остальные настройки остались по умолчанию:
- минимальная требуемая оценка взаимодействий (minimum required interaction score) = средняя степень достоверности (medium confidence) (4.000);
- максимальное число отображаемых взаимодействий со входной последовательностью (max number of interactions to show) = не более чем 10 взаимодействующих белков (no more than 10 interactors).
Ниже представленно полученное изображение. На нем узлы символизируют КОГи, а ребра - предсказанные функциональные связи.
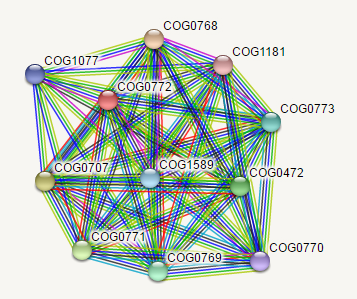
Ребра графа максимально могут быть покрашены в семь цветов, обозначающих разные типы доказательств в предсказании связей.
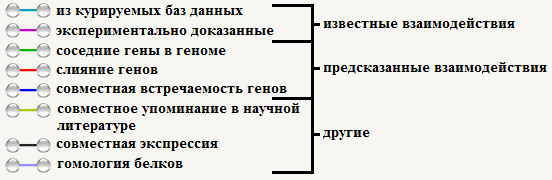
Ниже представлено изображение таблицы, соответствующей полученному графу. В последней колонке дана оценка достоверности взаимосвязей (максимум = 1). По точкам можно понять, на основании чего, программа выдала такой результат.

Из таблицы видно, что экспериментально доказанных взаимодействий (Experiments) немного (4 из 10), чуть больше достоверных взаимодействий, взятых из различных баз данных (Databases), (5 из 5). И лишь первая находка сочетает все типы доказательств. Тем не менее вывод программы о взаимосвязах основывался в большей степени на соседстве генов в геномах (Neighborhood) и на совместном упоминании белков или их генов в научной литературе (Textmining).
Затем я переключилась в раздел "Evidence" -> "Neighborhood", где представлено следующее изображение геномного окружения.
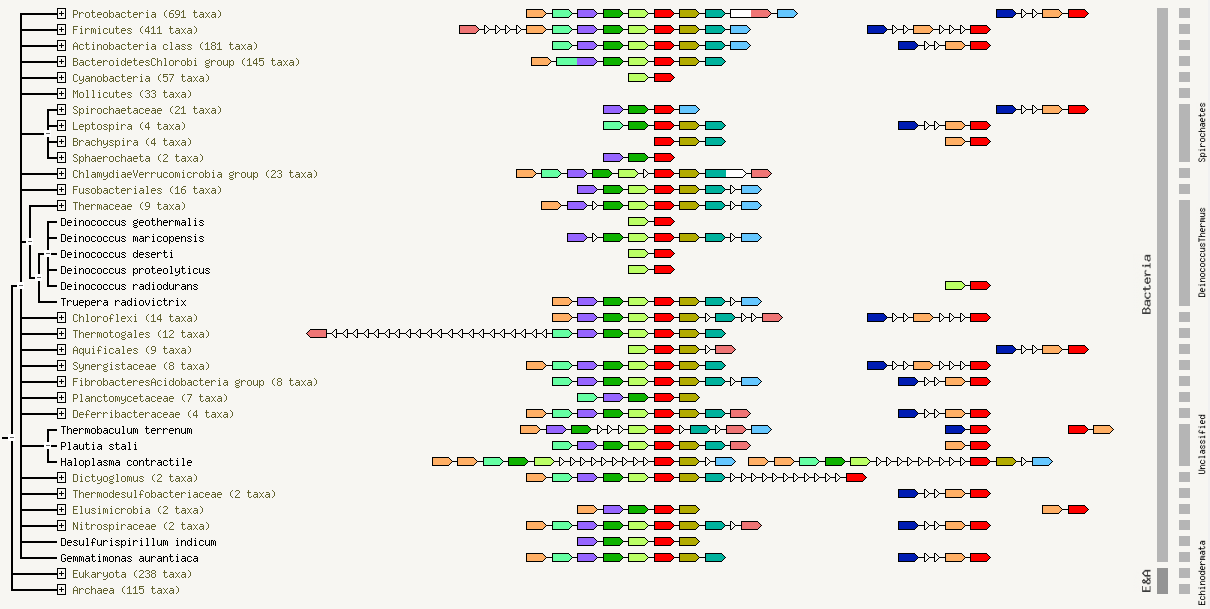
Цвет гена на рисунке соответствует цвету КОГа, к которому относится данный белок. Белые треугольнички - это гены белков, относящихся к другим КОГам. Направление стрелочки указывает на направление транскрипции гена. Если стрелочка окрашена двумя цветами, то это означает, что различные части гена могут иметь различные эволюционные истории. Это может быть следствием слияния (сшивки) генов или деления гена.
Я считаю, что в данной группе белков наблюдается консервативное геномное окружение, которое хоть и имеет некоторые вариации между разными таксономическими группами организмов, но все же имеет тенденцию сохраняться. В данном случае корректно говорить, что у нас есть две консервативные группы, образованные КОГами:
- COG0768, COG0769, COG0770, COG0472, COG0771, COG0772, COG0707, COG0773, COG1181, COG1589;
- COG1077, COG0768, COG0772.
Отнесение альфа субъединицы АТФ-синтазы из бактерии Escherichia coli штамм K-12 к терминам GO
База данных Gene Ontology (GO) содержит три структурированные онтологии, которые описывают генные продукты с точки зрения связанных с ними биологических процессов, клеточных компонентов и молекулярных функций не зависимо от вида.
На странице сервиса AmiGO я ввела индентификатор (Accession number P0ABB0) белка из базы данных UniProt, ограничила число выравниваний до 20 и запустила BLAST.
Наилучшая находка относилась к гену atpA из бактерии Escherichia coli штамм K-12. Именно этот ген в геноме E. coli кодирует альфа субъединицу АТФ-синтазы. Однако их соответствующие белковые последовательности оказались не полностью идентичными с E-value равным 2.0e-255. В последовательности исследуемого белка, взятого из UniProt присутствует вставка длиной в 18 аминокислот, которой нет в других приведенных последовательностях. Изображение парного выравнивания с лучшей находкой приведено ниже.

Мне кажется, что данная вставка является результатом ошибки авторов, аннотировавших последовательность альфа субъединицы АТФ-синтазы (P0ABB0) в базе данных UniProt. Поэтому можно считать что найдена та же самая последовательность.
Далее я перешла на страницу белка-находки, нажала на кнопку "5 terms associations" и вышла на страницу с перечисленными терминами GO, относящимися к наилучшей находке, а следовательно и к исследуемому белку. Информация о найденных терминах GO приведена в таблице 2.
| Таблица 2. Термины GO, отнесенные к белку с идентификатором UniProt P0ABB0 (ATPA_ECOLI) | ||||
| Аспект | Идентификатор | Название термина | Перевод названия термина | Код типа достоверности |
| Биологический процесс (Biological process) |
GO:0015986 | ATP synthesis coupled proton transport | АТФ синтез в сочетании с протонным транспортом | IMP |
| Клеточный компонент (Cellular component) |
GO:0016020 | Membrane | Мембрана | IDA |
| Клеточный компонент (Cellular component) |
GO:0045261 | Proton-transporting ATP synthase complex, catalytic core F(1) | Протон-транспортирующий АТФ синтазный комплекс, каталитическое ядро F(1) | IMP |
| Молекулярная функция (Molecular function) |
GO:0005515 | Protein binding | Cвязывание с белками | IPI |
| Молекулярная функция (Molecular function) |
GO:0046961 | Proton-transporting ATPase activity, rotational mechanism | Протон-транспортирующая АТФазная активность, механизм вращения | IDA |
Поясню, что означают указанные коды типа достоверности из таблицы 2. Для этого обратимся к таблице 3.
| Таблица 3. Описание кодов достоверности, использованных в таблице 2 | ||
| Код типа достоверности |
Расшифровка кода типа достоверности |
Объяснение |
| IMP | Inferred from Mutant Phenotype Выведен из мутантного фенотипа |
Данный код достоверности охватывает те случаи, когда функция, процесс или клеточная локализация продукта гена определяется, исходя из соответствующих различий между двумя различными аллелями ("диким" и "мутантным") в соответствующем гене. Следует осторожно принимать при оформлении аннотаций, так как может быть трудным определить нормальную функцию гена, имея лишь данные про появление функции в мутантном варианте, хотя иногда это возможно. |
| IDA | Inferred from Direct Assay Выведен из прямого анализа |
Данный код достоверности используется для обозначения того, что был проведен прямой анализ (эксперимент), направленный на определение функции, процесса или компонента, обозначенного термином GO. Поэтому кураторы должны быть осторожны, потому что эксперимент, считающийся прямым анализом для термина одной онтологии, может отличаться от такового для термина из другой онтологии. |
| IPI | Inferred from Physical Interaction Выведен из физического взаимодействия |
Данный код достоверности охватывает взаимодействия между объектом интереса и другими молекулами (например, белки, ионы или комплексы). Часто бывает трудно по доказательствам, представленным в статьях определить, есть ли прямое взаимодействие или нет. Единственные методы, которые предоставляют прямое доказательство связи двух белков, - это их выделение и предварительная очистка. Поэтому допустимо существование взаимодействий, даже если еще не известно, прямые они или нет. |