Поиск сигналов
Определение биологической роли транскрипционного фактора SaeR в бактерии Staphylococcus epidermidis ATCC 12228
Ссылка на задание, выполненное напарницей - Кузнецовой Ксенией.
Влияние метилирования на связывание транскрипционного фактора SaeR со своим сайтом
Сперва я получила файлы motif1.fasta и motif2.fasta с последовательностями лучших мотивов (NP_763718.1|SE0163 и NP_764036.1|SE0481 соответственно) вместе с окрестностями ± 50 нуклеотидов по бокам от них. Информация бралась из генома бактерии Staphylococcus epidermidis ATCC 12228
С помощью программы fuzznuc из пакета EMBOSS я нашла все сайты метилирования, пересекающиеся с предсказанными ранее сайтами связывания транскрипционного фактора SaeR, в бактерии Staphylococcus epidermidis ATCC 12228. В последовательностях motif1.fasta и motif2.fasta программа искала сайты из файла MT-sites.txt. Команда выглядит следующим образом:
fuzznuc -sequence motif1.fasta -pattern @MT-sites.txt -outfile fuzznuc1.out
fuzznuc -sequence motif2.fasta -pattern @MT-sites.txt -outfile fuzznuc2.out
Полученные результаты приведены в таблице 1.
| Таблица 1. Результаты поиска сайтов метилирования | ||
| Файл с мотивом | motif1.fasta | motif2.fasta |
| Мотив | GTTAATAAATAGTTAA | GTTAAGAAATAGTTAA |
| Upstream взят для гена | SE0163 | SE0481 |
| Длина участка | 116 | 116 |
| Выходной файл | fuzznuc1.out | fuzznuc2.out |
| Найдено сайтов метилирования | 87 | 89 |
| Найдено однонуклеотидных сайтов | 42 | 50 |
| Найдено динуклеотидных сайтов | 11 | 7 |
| Число пересечений с сайтом связывания ТФ SaeR (без однонуклеотидных сайтов метилирования) |
13 (6) | 11 (5) |
В таблицах 2 и 3 подробнее указаны сайты метилирования, пересекающиеся с сайтами связывания транскрибционного фактора SaeR. Напомню, что в обоих файлах с последовательностями мотивов их относительные координаты 50-66.
| Таблица 2. Сайты метилирования для motif1.fasta | |
| Относительные координаты | Сайт |
| 46-50 | ACGGT |
| 48-53 | GGTAAT |
| 51-54 62-65 |
AATT |
| 55-57 | GAT |
| 66-71 | GAAATC |
| 50, 53, 54, 57, 61, 64, 65 |
T |
| Таблица 3. Сайты метилирования для motif2.fasta | |
| Относительные координаты | Сайт |
| 48-53 | GGTAAT |
| 51-54 62-65 |
AATT |
| 55-57 | GAT |
| 66-68 | GAT |
| 50, 53, 54, 57, 64, 65 |
T |
Если отбросить все неспецифические сайты метилирования, то остаются по 5-6 сайтов, которые, возможно действительно, участвуют в регуляции транскрипции. Чтобы это поточнее узнать, необходимо посмотреть, какие ДНК-метилтрансферазы кодируются в геноме бактерии Staphylococcus epidermidis ATCC 12228.
В базе данных REBASE я нашла геном исследуемой бактерии. На рисунке ниже представлено схематичное изображение кольцевой хромосомы нашей бактерии с указанием генов, кодирующих белки системы рестрикции-модификации.
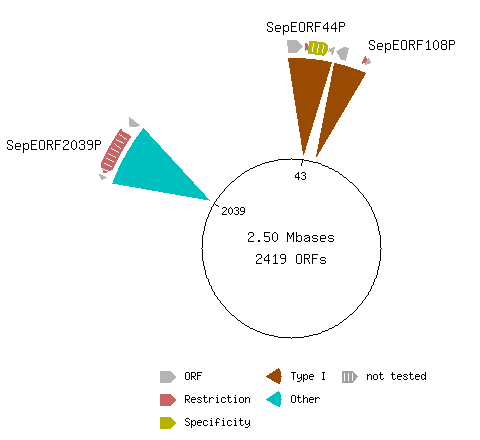
К сожалению, в геноме нашей бактерии отсутствуют гены, кодирующие ДНК-метилтрансферазы. Поэтому следует вывод, что метилирование не влияет на связывание транскрипционного фактора SaeR со своим сайтом.
asershova@gmail.com - ершова