Определение вторичной структуры
Перед нами стоит задача сравнить предсказания вторичной структуры белка двух программ DSSP и Stride с реально известной структурой. Для этого я выбрала структуру β-лактамазы TEM1 (PDB ID = 1XPB), в которой присутствуют как α-спирали, так и β-листы. Для получения предсказания алгоритмом Stride я воспользовалась онлайн-сервисом и получила файл Stride.html. А для предсказания алгоритмом DSSP я воспользовалась версией, установленной на kodomo, введя в командную строку:
mkdssp -i 1xpb.pdb -o dssp.txt
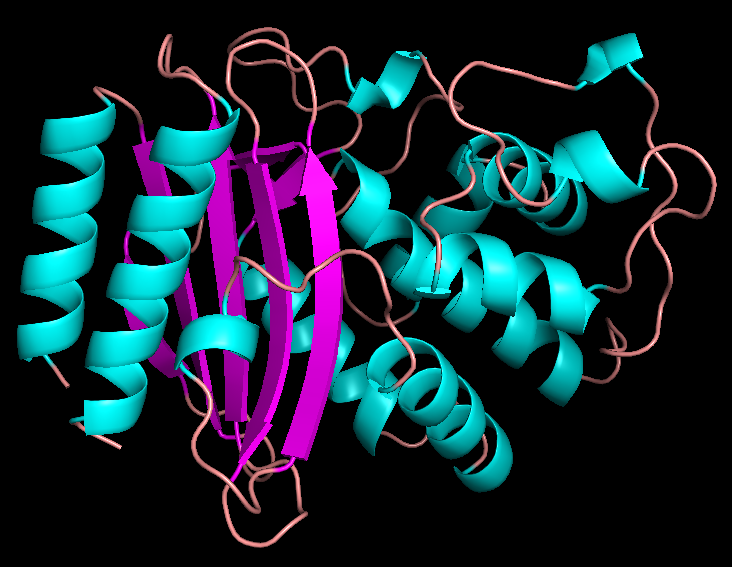
В результате я получила файл dssp.txt. Ниже представлено изображение выбранного белка с раскраской по элементам вторичной структуры.
Для сравнения алгоритмов предсказания мною были выбраны две спирали (длинная - α и короткая - 310) и два β-листа (большой из 5 β-тяжей и маленький из 2 коротких β-тяжей). Они отчетливо видны на структуре, представленной на следующей картинке.
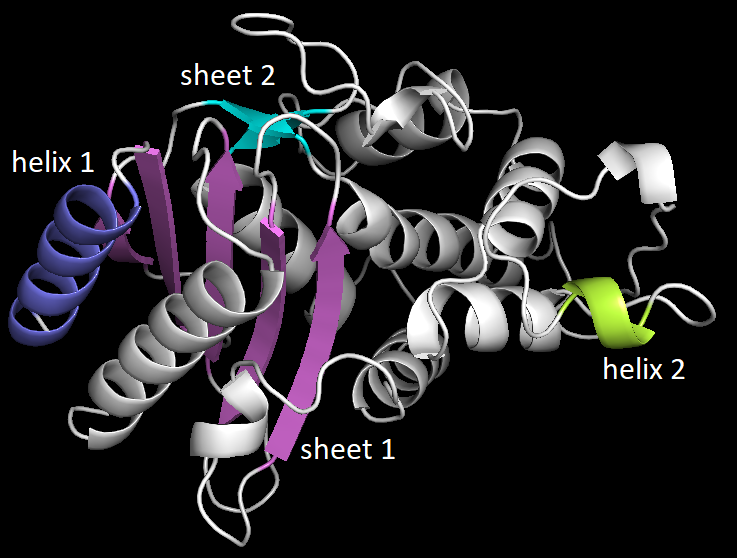
Далее в виде таблицы я оформила сравнение координат выбранных структур по аннотации PDB и по предсказаниям. Жирным шрифтом выделены несовпадения границ.
| Таблица 1. Сравнение границ элементов вторичной структуры, полученных программами, с границами из PDB файла | |||
| Элемент | PDB | Stride | DSSP |
| α-спираль 1 | Pro 27 - Leu 40 | Pro 27 - Leu 40 | Pro 27 - Leu 40 |
| 310-спираль 2 | Thr 109 - His 112 | Thr 109 - Lys 111 | Thr 109 - His 112 |
| β-лист 1 | Ile 55 - Phe 60 Arg 43 - Asp 50 Arg 259 - Thr 266 Arg 244 - Gly 251 Phe 230 - Ala 237 |
Ile 55 - Phe 60 Arg 43 - Asp 50 Arg 259 - Thr 266 Arg 244 - Gly 251 Phe 230 - Ala 237 |
Ile 55 - Phe 60 Arg 43 - Asp 50 Arg 259 - Thr 266 Arg 244 - Gly 251 Phe 230 - Ala 237 |
| β-лист 2 | Arg 65 - Pro 67 Thr 180 - Met 182 |
Phe 66 - Pro 67 Thr 180 - Thr 181 |
Phe 66 - Pro 67 Thr 180 - Thr 181 |
Из таблицы 1 видно, что протяженные элементы, как α-спираль 1 и β-лист 1 одинаково хорошо предсказываются обеими программами. Однако короткие 310-спирали и β-листы порой имеют смещенные на один аминокислотный остаток границы по сравнению со вторичной структурой, аннотированной в PDB файле. На данной конкретной структуре более точной оказалась программа DSSP. Хочу отметить, что в выдаче программ Stride и DSSP предсказан короткий (2 остатка) β-тяж, который не является таковым в PDB файле. Все остальные элементы обнаружены весьма точно.
Ниже представлено сравнение предсказанной и нативной вторичной структуры по аминокислотным остаткам последовательности. Примечание: в описании PDB α-спирали и 310-спирали отмечаются одной буквой H, а β-тяжи - буквой E, другие элементы не уточняются.
SEQ 1 HPETLVKVKDAEDQLGARVGYIELDLNSGKILESFRPEERFPMMSTFKVL 50 STR HHHHHHHHHHHHHH EEEEEEEETTTT EEEEETTTT EE GGGGGGH DSSP HHHHHHHHHHHHHHTSEEEEEEEETTT EEEEESTTS EE GGGHHHH PDB HHHHHHHHHHHHHH EEEEEEEE EEEEE EEE HHHHHHH REM REM . . . . . SEQ 51 LCGAVLSRVDAGQEQLGRRIHYSQNDLVEYSPVTEKHLTDGMTVRELCSA 100 STR HHHHHHHHHHH TTTTEE GGG TTTTTTGGGTTTTTEEHHHHHHH DSSP HHHHHHHHHHTTS TT EE GGG SS TTGGGGTTT EEHHHHHHH PDB HHHHHHHHHH HHH HHHH HHHHHHH REM REM . . . . . SEQ 101 AITMSDNTAANLLLTTIGGPKELTAFLHNMGDHVTRLDRWEPELNEAIPN 150 STR HHHH HHHHHHHHHHH HHHHHHHHHHH TTTT TTTGGG TTT DSSP HHHS HHHHHHHHHHHT HHHHHHHHHHTT SS S TTGGG TT PDB HHH HHHHHHHHHHH HHHHHHHHHH HHH REM REM . . . . . SEQ 151 DERDTTMPAAMATTLRKLLTGELLTLASRQQLIDWMEADKVAGPLLRSAL 200 STR TTTTEE HHHHHHHHHHHHH TTTTHHHHHHHHHHHHHTTTTTTTGGGG DSSP TTEE HHHHHHHHHHHHHSSSS HHHHHHHHHHHHT S IIIIIHH PDB EEEHHHHHHHHHHHHH HHHHHHHHHHHH HHHH REM REM . . . . . SEQ 201 PAGWFIADKSGAGERGSRGIIAALGPDGKPSRIVVIYTTGSQATMDERNR 250 STR TTTTEEEEEEEETTTTTEEEEEEEETTTT EEEEEEEE HHHHHH DSSP TT EEEEEEEE STT EEEEEEEESTT EEEEEEEES S HHHHHH PDB EEEEEEEE EEEEEEEE EEEEEEEE HHHHHH REM REM . SEQ 251 QIAEIGASLIKHW 263 STR HHHHHHHHHHHH DSSP HHHHHHHHHHHT PDB HHHHHHHHHHH