 |
Занятие 13. SCOP и CATH.
-
Запись PDB 1KNP содержит информацию о структуре белка NADB_ECOLI, состоящего из одной цепи А.
Для этой цепи была проведена классификация согласно CATH и SCOP. И CATH, и SCOP определили
три домена. Ниже в таблицах представлена информация о доменах из SCOP и о соответстствующих
доменах из CATH.
Классификация доменов.
|
SCOP |
CATH |
|---|
| Положение в своей структуре (цепь, координаты) |
5-237;354-422 |
5-244;349-424 |
| Классификация по БД |
класс: α/β белки [51349]
укладка: FAD/NAD(P)-связывающий домен [51904]
(FAD/NAD(P)-binding domain)
суперсемейство: FAD/NAD(P)-связывающий домен [51905]
(FAD/NAD(P)-binding domain)
семейство: сукцинат дегидрогеназа/N-концевой домен фумарат редуктазы флавопротеина
(Succinate dehydrogenase/fumarate reductase flavoprotein N-terminal domain) [51934]
SCOP_ID этого домена из записи 1knp: [72784]
|
3.50.50.60.40.1.1.2.2
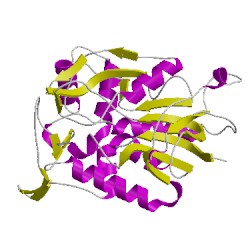
класс: α β
архитектура: 3-уровневый (ββα) сэндвич
(3-Layer(bba) Sandwich)
топология: FAD/NAD(P)-связывающий домен
(FAD/NAD(P)-binding domain)
|
| Сколько семейств содержится в данном суперсемействе |
8 |
61 |
| Сколько суперсемейств имеет данную укладку (топологию для CATH)? |
1 |
1 |
Как видно, SCOP и CATH определили практически аналогичные домены. Есть совсем небольшое различие (несколько а.о.) на границах домена,
однако функция и "строение" (структура, расположение по последовательности) у них одинаково.
|
SCOP |
CATH |
|---|
| Положение в своей структуре (цепь, координаты) |
238-353 |
245-348 |
| Классификация по БД |
класс: α+β белки [53931]
укладка: сукцинат дегидрогеназа/N-концевой домен фумарат редуктазы флавопротеина [56424]
(Succinate dehydrogenase/fumarate reductase flavoprotein, catalytic domain)
суперсемейство: сукцинат дегидрогеназа/фумарат редуктаза флавопротеина, каталитический
домен [56425]
(Succinate dehydrogenase/fumarate reductase flavoprotein, catalytic domain)
семейство: сукцинат дегидрогеназа/фумарат редуктаза флавопротеина, каталитический
домен [56426]
(Succinate dehydrogenase/fumarate reductase flavoprotein, catalytic domain)
SCOP_ID этого домена из записи 1knp: [72785]
|
3.90.700.10.4.1.1.1.3
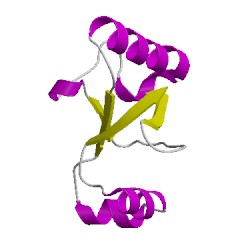
класс: α β
архитектура: α β комплекс
(Alpha-Beta Complex)
топология: флавоцитохром C3; цепь А, домен 1
(Flavocytochrome C3; Chain A, domain 1)
суперсемейство: флавоцитохром C3; цепь А, домен 1
(Flavocytochrome C3; Chain A, domain 1)
|
| Сколько семейств содержится в данном суперсемействе |
1 |
4 |
| Сколько суперсемейств имеет данную укладку (топологию для CATH)? |
1 |
1 |
Аналогичная ситуация видна и для второго домена.
|
SCOP |
CATH |
|---|
| Положение в своей структуре (цепь, координаты) |
423-533 |
425-533 |
| Классификация по БД |
класс: только α белки [46456]
укладка: Spectrin repeat-like [46965]
суперсемейство: сукцинат дегидрогеназа/N-концевой домен фумарат редуктазы флавопротеина
[46977]
(Succinate dehydrogenase/fumarate reductase flavoprotein C-terminal domain)
семейство:сукцинат дегидрогеназа/N-концевой домен фумарат редуктазы флавопротеина
[46978]
(Succinate dehydrogenase/fumarate reductase flavoprotein C-terminal domain)
SCOP_ID этого домена из записи 1knp: [72783]
|
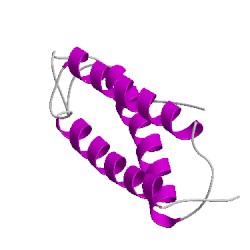
Классификация этого домена в CATH находится на стадии разработки.
|
| Сколько семейств содержится в данном суперсемействе |
1 |
- |
| Сколько суперсемейств имеет данную укладку (топологию для CATH)? |
16 |
- |
А вот C-концевой домен CATH выделил, однако классификация по нему еще не сделана, поэтому про разницу между ними ничего сказать нельзя
(кроме того, что домен, определенный SCOPб на 2 а.о. длинее).
-
Две структуры
1TIG
и 1DCJ
содержат по единственному домену. Они по классификации CATH принадлежат одной и той же топологии
(Фактор инциации трансляции IF3 - Translation Initiation Factor IF3),
но к разным суперсемействам (Translation Initiation Factor If3 для 1TIG и SirA-like для 1DCJ). Эта топология состоит в архитектуре
"2-уровневый сэндвич" (2-Layer Sandwich) класса α β.
Полные классификации обоих доменов:
1tigA00 - 3.30.110.10.1.1.1.1.1
 1dcjA00 - 3.30.110.40.1.1.1.1.1
1dcjA00 - 3.30.110.40.1.1.1.1.1

В программе PyMol было построено совмещение структур с помощью программы super:
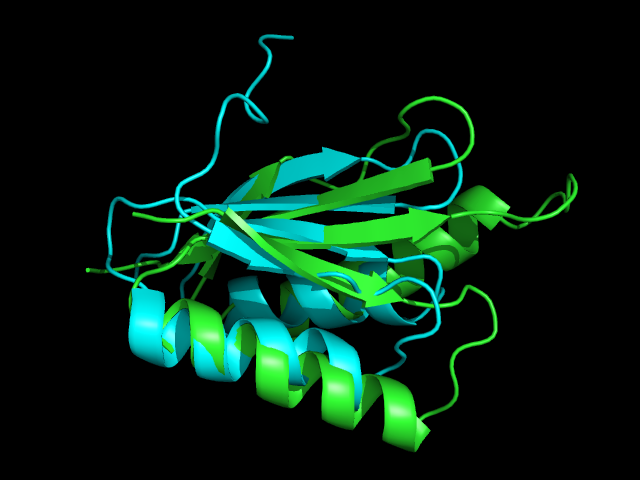
Зеленым выделен 1TIG, голубым - 1DCJ.
Полученное RMSD - 2.244.
Как видно, структуры хоть и неидеально, однако все-таки достаточно хорошо наложились друг на друга (альфа-спирали совпали
друг с другом, тоже можно сказать и о бета-листах).
|
|
|
