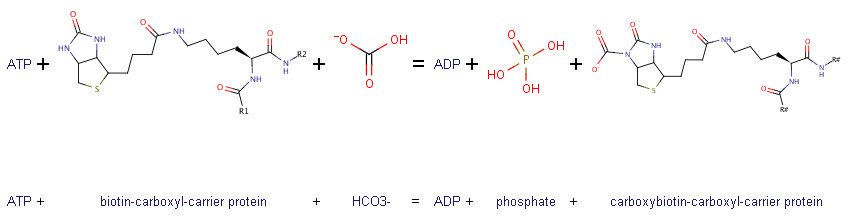

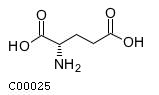
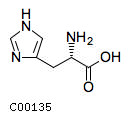
| Организм | Возможна ли цепочка реакций (да/нет/неизвестно) |
Обоснование |
| Escherichia coli K-12 MG1655 | да | присутствуют все ферменты, необходимые для осуществления цепочки реакций |
| Archaeoglobus fulgidus | да | присутствуют все ферменты, необходимые для осуществления цепочки реакций |
| Arabidopsis thaliana | да | присутствуют все ферменты, необходимые для осуществления цепочки реакций |
| Homo sapiens | да | присутствуют все ферменты, необходимые для осуществления цепочки реакций |