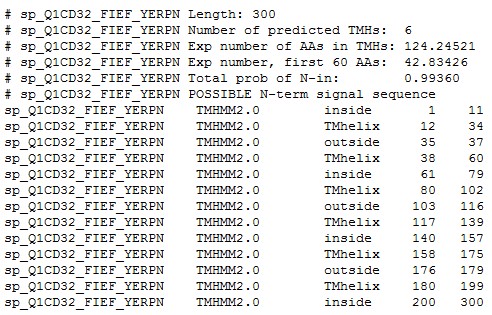
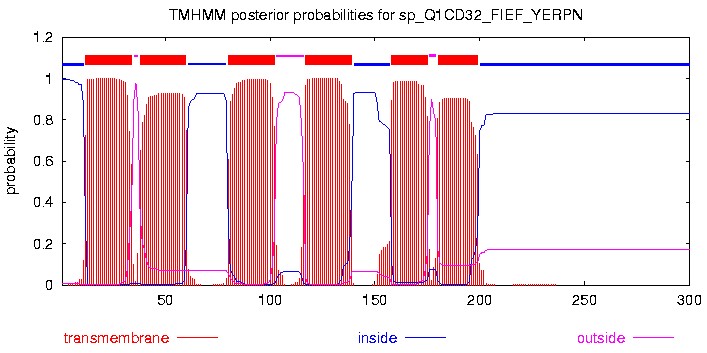
Сенсорный родопсин II
Организм - Natronomonas pharaonis
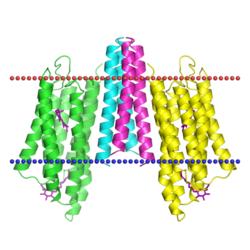
Сериновая протеаза EspP, пострасщепляющая стадия.
Организм - Escherichia coli
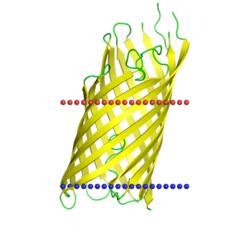
CIC хлоридный транспорт
Организм - Escherichia coli.

Фосфопорин (PhoE).
Организм - Escherichia coli.

Димер, бета2-адренергический рецептор.
Организм - Homo sapiens
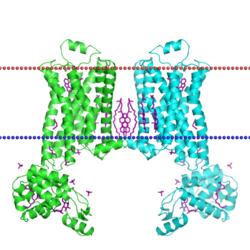
Цитолизин.
Организм - Vibrio cholerae
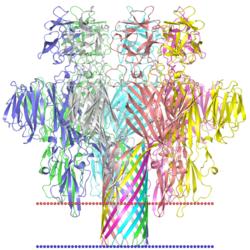
Ионный канал закрытый лигандом, активная конформация.
Организм - Gloeobacter violaceus.
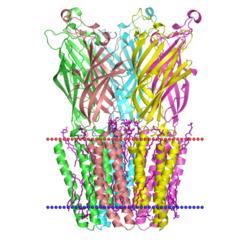
Порин MspA.
Организм - Mycobacterium smegmatis.
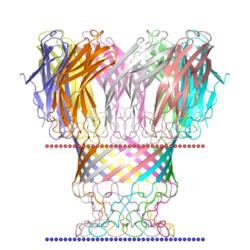
Компонент транспортера Saccharide, EIIC (ChbC).
Организм - Bacillus cereus.
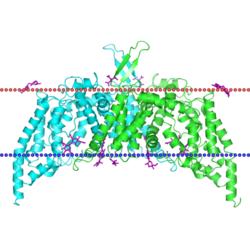
Ацетилаза PagP липида А.
Организм - Escherichia coli.