Реконструкция деревьев по нуклеотидным последовательностям
В базе данных EMBL были добыты последовательности гена 16S. Для Listeria monocytogenes не удалось найти данную последовательность, так как в файле entret не было записи для 16S.
Выравнивание

Для построения дерева я использовала программу fdnaml. Почему-то LACDAC довольно сильно отличается от остальных. Это видно в выравнивании и на дереве. Я предположила ошибку, но последовательность в выравнивании содержит довольно много консервативных участков и похожую длину и, сделав предположение, что она является аут-группой, я все-ранво не получила правильного укоренения. Хотя при данной топологии оно впринципе невозможно.
Анализ деревьев, содержащих паралоги.
Я построила дерево в программе JalView, так как в этой программе удобно сделать выравнивание и сразу воспользоваться нужным алгоритмом(Neighbour Joining Using % Identity):
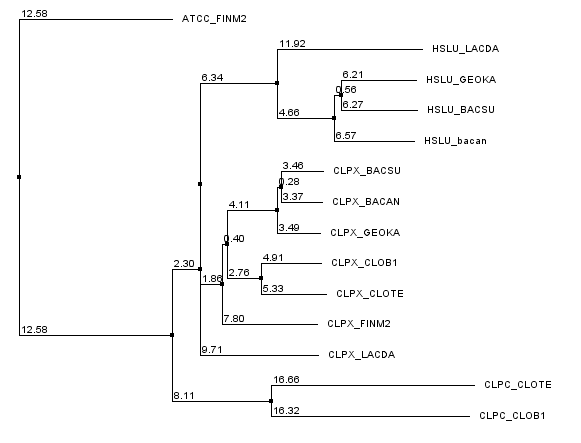
На дереве видно четкое разделение по типам.
Пример паролога:CLPX_BACSU и HLSU_BACSU
Пример ортолога:HSLU_GEOKA, HSLU_LACDA, HSLU_BACAN, HSLU_BACSU
©Умарова, 2009-2012

