| Характеристика паттерна | Паттерн | В скольких последовательностях банка Swiss-Prot найден мотив, удовлетворяющий паттерну? | Все ли последовательности из Вашего выравнивания найдены? |
| Фрагмент последовательности | HLSYFRR | 1 | Найден только мой белок YESU_BACSU |
| Сильный | [HR]-[LVA]-S-[YF]-F-[ERP]-[RP]-x(0,3) | 5 | Найден только мой белок, так как его гомологи были найдены в "nr". Все белки могут быть найдены, если искать по TREMBL. |
| Слабый | [HR]-[LIVMA]-S-[FYW]-F-[ERP]-[RP]-x(0,7) | 8 | Найден только мой белок. |
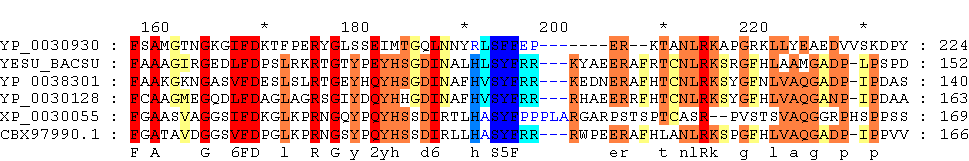
Первый паттерн - фрагмент последовательности YESU_BACSU. В сильный паттерн включены все позиции выбранного фрагмента выравнивания, а в каждой позиции разрешены все буквы, встретившиеся в этой позиции, и только они. Слабый паттерн создан на основе предыдущего, но с более мягкими требованиями к последовательности. На сайте Pro-Site проведен поиск последовательностей по Swiss-Prot, включающих мотивы, соответствующие полученным паттернам. При ненахождении последовательностей из выравнивания проведен аналогичный поиск, но охватывающий также TREMBL.
| Идентификатор документа PROSITE (AC) | Название мотива | Краткое описание мотива | Тип подписи (паттерн, профиль) | Паттерн (регулярное выражение) | Специфична ли подпись? | Сколько мотивов нашлось в белке? |
| PS00006 | CK2_PHOSPHO_SITE | Сайт фосфорилирования казеин киназы II | паттерн | [ST]-x(2)-[DE] | неспецифична | 6 |
| PS00008 | MYRISTYL | Сайт N-миристоилирования | паттерн | G-{EDRKHPFYW}-x(2)-[STAGCN]-{P} | неспецифична | 2 |
| PS00005 | PKC_PHOSPHO_SITE | Сайт фосфорилирования протеин киназы C | паттерн | [ST]-x-[RK] | неспецифична | 1 |
| PS00004 | CAMP_PHOSPHO_SITE | Сайт фосфорилирования cAMP- и cGMP-зависимой протеин киназы | паттерн | [RK](2)-x-[ST] | неспецифична | 1 |
| PS00007 | TYR_PHOSPHO_SITE | Сайт фосфорилирования тирозин киназы | паттерн | [RK]-x(2)-[DE]-x(3)-Yor[RK]-x(3)-[DE]-x(2)-Y | неспецифична | 1 |