| Название | Мнемоника | Таксономия |
| Bacillus anthracis | BACAN | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group |
| Bacillus subtilis | BACSU | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
| Clostridium botulinum | CLOB1 | cellular organisms; Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Clostridium tetani | CLOTE | cellular organisms; Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Enterococcus faecalis | ENTFA | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Finegoldia magna | FINM2 | cellular organisms; Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiales incertae sedis; Clostridiales Family XI. Incertae Sedis; Finegoldia |
| Geobacillus kaustophilus | GEOKA | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Lactobacillus acidophilus | LACAC | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
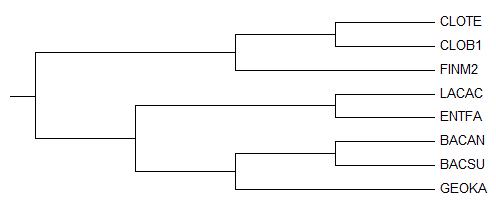
(((CLOTE,CLOB1),FINM2),((LACAC,ENTFA),((BACAN,BACSU),GEOKA)));
Выравнивание белков семейства IF2 из отобранных бактерий было создано программой muscle.
((IF2_ENTFA,((IF2_GEOKA,(IF2_BACSU,(IF2_BACAN,(IF2_CLOTE,IF2_CLOB1)))), IF2_FINM2)),IF2_LACAC);
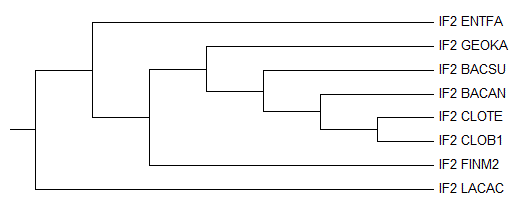
Оно значительно отличается от правильного. В нем отсутствуют ветви
{CLOTE,CLOB1,FINM2}vs{LACAC,ENTFA,BACAN,BACSU,GEOKA},
{CLOTE,CLOB1,FINM2,LACAC,ENTFA}vs{BACAN,BACSU,GEOKA},
{CLOTE,CLOB1,FINM2,LACAC,ENTFA,GEOKA}vs{BACAN,BACSU},
и присутствуют
{CLOTE,CLOB1,BACAN}vs{FINM2,LACAC,ENTFA,BACSU,GEOKA},
{CLOTE,CLOB1,BACAN,BACSU}vs{FINM2,LACAC,ENTFA,GEOKA},
{CLOTE,CLOB1,BACAN,BACSU,GEOKA}vs{FINM2,LACAC,ENTFA},
потому что клада {CLOTE,CLOB1} отходит от узла не с FINM2, а с BACAN.
8
IF2_LACAC 0.000000 0.931545 0.817082 0.877344 0.664057 0.666956 0.680116 0.681323
IF2_FINM2 0.931545 0.000000 0.686237 0.642369 0.928257 0.716631 0.814132 0.699183
IF2_CLOB1 0.817082 0.686237 0.000000 0.379515 0.738980 0.633717 0.686087 0.608255
IF2_CLOTE 0.877344 0.642369 0.379515 0.000000 0.765868 0.664837 0.663418 0.645844
IF2_ENTFA 0.664057 0.928257 0.738980 0.765868 0.000000 0.558361 0.590738 0.524516
IF2_BACSU 0.666956 0.716631 0.633717 0.664837 0.558361 0.000000 0.416354 0.320268
IF2_GEOKA 0.680116 0.814132 0.686087 0.663418 0.590738 0.416354 0.000000 0.315306
IF2_BACAN 0.681323 0.699183 0.608255 0.645844 0.524516 0.320268 0.315306 0.000000
Расстояния отклоняются от ультраметричности, например:(IF2_ENTFA:0.28155,((IF2_FINM2:0.38605,(IF2_CLOB1:0.18231, IF2_CLOTE:0.19720):0.08849):0.14020,(IF2_BACSU:0.17732,(IF2_GEOKA:0.19590, IF2_BACAN:0.11940):0.03334):0.03806):0.06048,IF2_LACAC:0.38251);
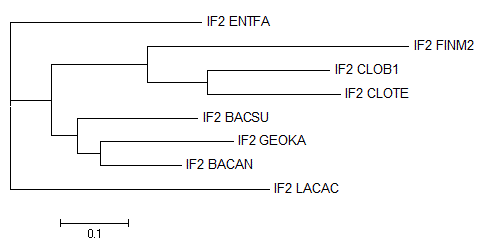
От правильного дерева отличается наличием ветви {CLOTE,CLOB1,FINM2,LACAC,ENTFA,BACSU}vs{BACAN,GEOKA} и отсутствием ветви {CLOTE,CLOB1,FINM2,LACAC,ENTFA,GEOKA}vs{BACAN,BACSU}.
((IF2_LACAC:0.33656,(IF2_ENTFA:0.27894,(IF2_BACSU:0.18416,(IF2_GEOKA:0.15765, IF2_BACAN:0.15765):0.02650):0.09478):0.05762):0.03648, (IF2_FINM2:0.33215,(IF2_CLOB1:0.18976,IF2_CLOTE:0.18976):0.14239):0.04089);
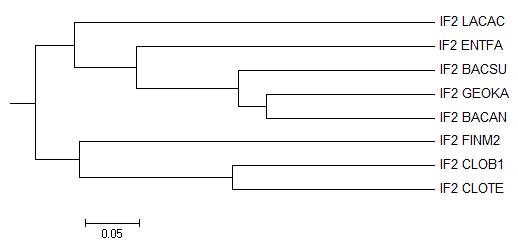
Отличается от предыдущих деревьев отсутствием ветви {CLOTE,CLOB1,FINM2,BACAN,BACSU,GEOKA}vs{LACAC,ENTFA} и наличием ветви {CLOTE,CLOB1,FINM2,LACAC}vs{BACAN,GEOKA,ENTFA,BACSU}. Так же, как и при алгоритме Neighbor-Joining имеется ветвь {CLOTE,CLOB1,FINM2,LACAC,ENTFA,BACSU}vs{BACAN,GEOKA} и отсутствует ветвь {CLOTE,CLOB1,FINM2,LACAC,ENTFA,GEOKA}vs{BACAN,BACSU}.