| Название | AC записей EMBL, описывающих полные геномы | Координаты рРНК |
| Bacillus anthracis | AE017225 | 9336-10845 |
| Bacillus subtilis | AL009126 | 9810-11364 |
| Clostridium botulinum | CP000726 | 9282-10783 |
| Clostridium tetani | AE015297 | 8715-10223 (complement) |
| Enterococcus faecalis | AE016830 | 248466-249987 |
| Finegoldia magna | AP008971 | 197837-199361 |
| Geobacillus kaustophilus | BA000043 | 10421-11973 |
| Lactobacillus acidophilus | CP000033 | 59255-60826 |
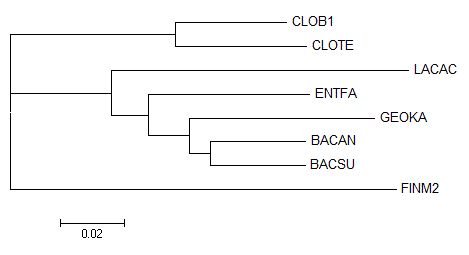
Выравнивание последовательностей 16S рибосомальной РНК данных бактерий, построенное с помощью muscle, было подано на вход программе fdnadist, а полученная матрица расстояний - программе fneighbor.

Дерево отличается от правильного только отсутствием ветви {CLOTE,CLOB1,FINM2,BACAN,BACSU,GEOKA}vs{LACAC,ENTFA} и наличием ветви {CLOTE,CLOB1,FINM2,LACAC}vs{BACAN,BACSU,GEOKA,ENTFA}. Оно лучше, чем некоторые деревья, построенные по белкам (например, методом максимальной экономии), но такое отличие от правильного дерева встречается впервые.
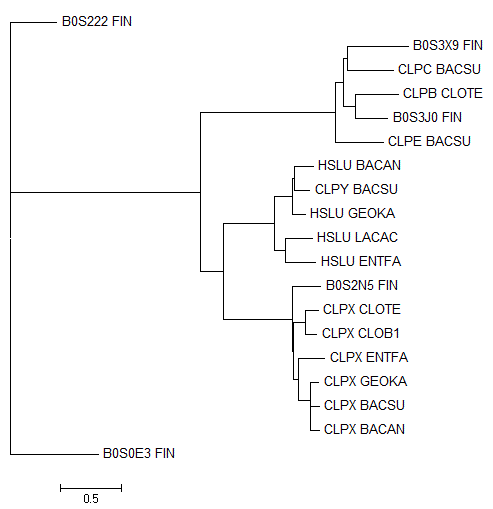
Ортологи: HSLU_LACAC и HSLU_ENTFA, CLPX_CLOTE и CLPX_CLOB1, CLPX_GEOKA, CLPX_BACSU и CLPX_BACAN.
Паралоги: B0S222_FINM2 и B0S0E3_FINM2.