Работа велась по хромосоме №6.
Анализ качества чтений. Триммирование.
После запуска программы FastQC до и после триммирования были получены html-файлы, содержащие отчёт о качестве чтений: до триммирования - отчёт №1, после триммирования - отчёт №2 Изначально было получено 10289 чтений. Качество чтений до очистки отображено на Рис. 1. С помощью Trimmomatic с конца каждого чтения были отрезаны нуклеотиды с качеством ниже 20, после чего оставлены только чтения длиной не меньше 50 нуклеотидов. После очистки осталось 10123 ридов. Качество чтений после очистки отображено на Рис. 2.
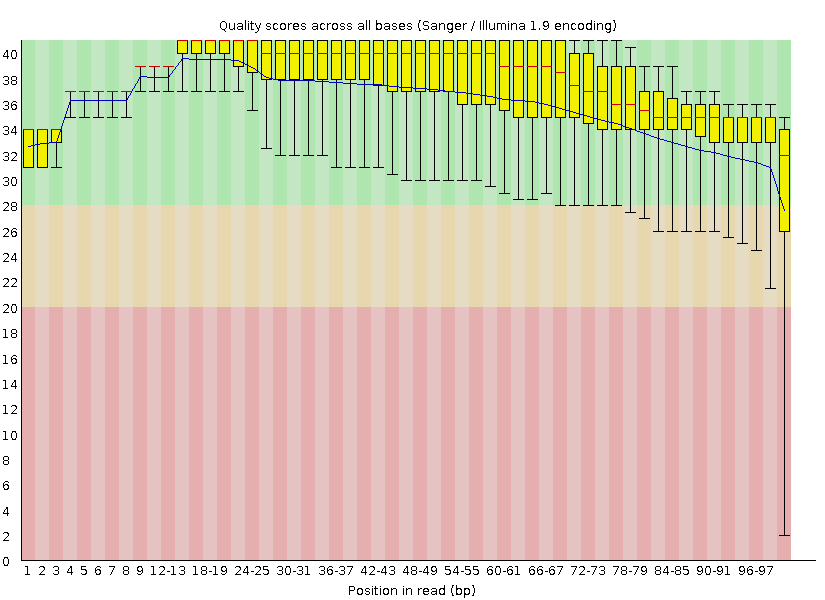
Из рисунка 1 мы видим, что не все основания имеют качество выше 20, после триммирования эти основания обрезаются.
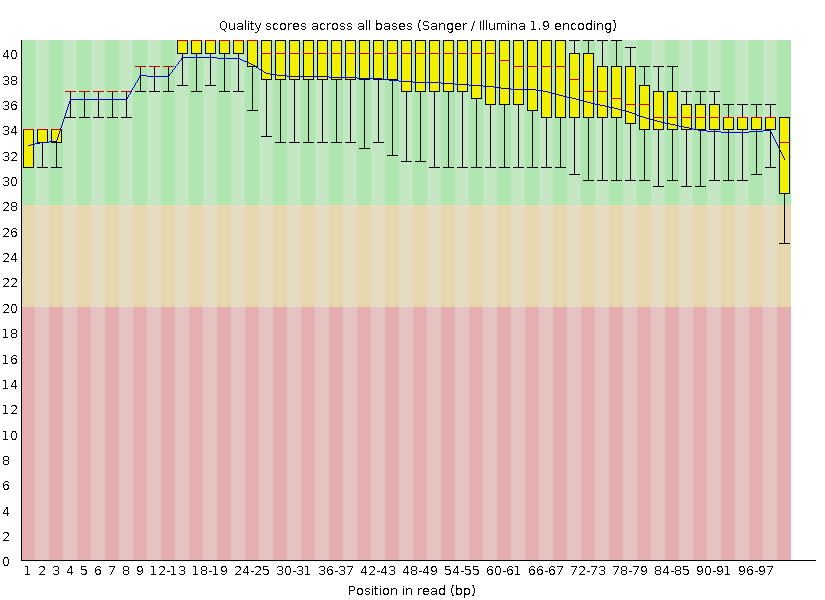
После триммирования из 10289 прочтений осталось 10123, было отброшено 166 - это менее 2% (1,61%). Сравнение остальных графиков не показало существенных изменений, разве что в графиках "Per sequence quality scores" есть небольшие различия - в гафике, составленном после триммирования количество последовательностей с качеством, меньше 20 равно 0, чего нельзя сказать о другом графике (Рис. 3, 4). Поэтому, я считаю, что можно было обойтись без триммирования.
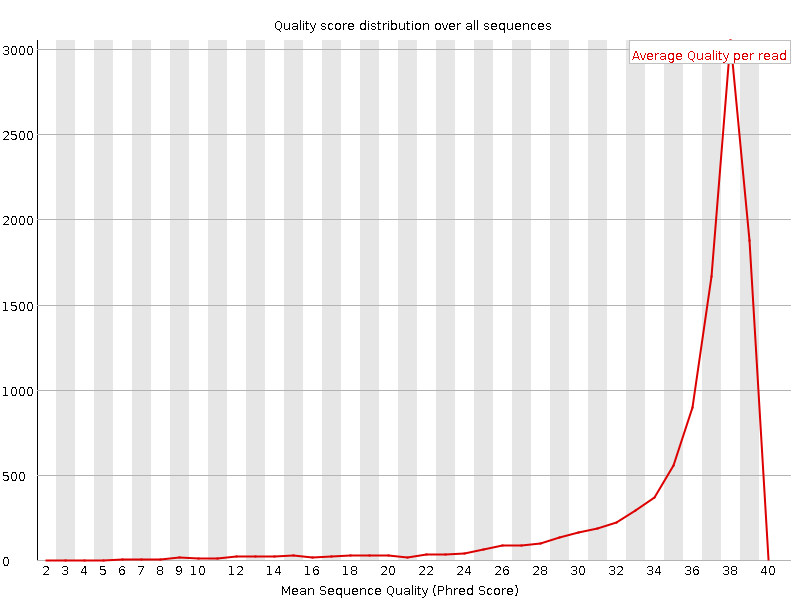
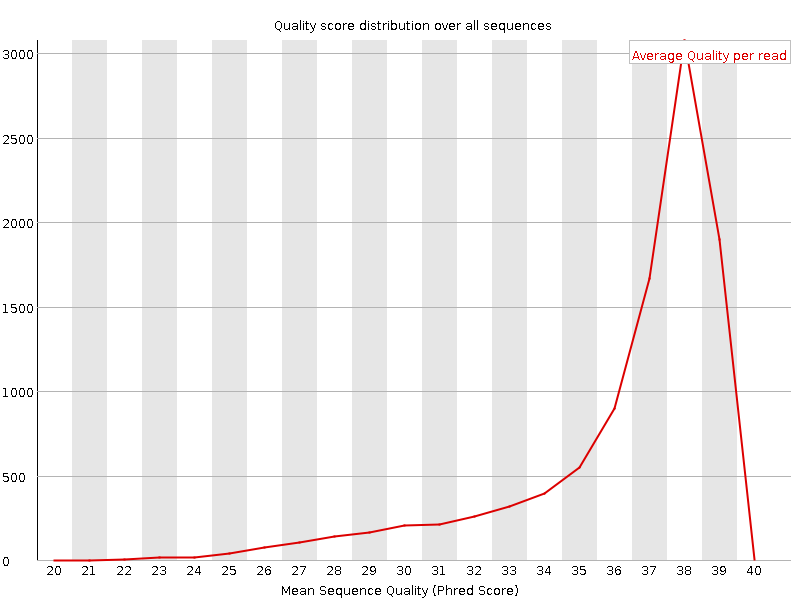
Картирование ридов
Команда "hisat2 -x index/chr6.idx -U reads/chr6_trimmed.fastq -S map/chr6_align.sam --no-softclip --no-spliced-alignment" вывела результат:
10123 reads; of these:
10123 (100.00%) were unpaired; of these:
77 (0.76%) aligned 0 times
10046 (99.24%) aligned exactly 1 time
0 (0.00%) aligned >1 times
99.24% overall alignment rate
из которого понятно, что всего на хромосому было картировано 10123 чтения (99.24%). Поэтому можно сделать вывод, что качество картирования высокое.
При рассмотрении файла chr6_snp.vcf с полиморфизмами понятно, что из 84 полиморфизмов 5 инделей и 79 snp. Описание трёх полиморфизмов приведено в таблице 2.
| № | Координата | Тип полиморфизма | Референс | Чтения | Глубина покрытия | Качество |
| 1. | 106961119 | замена | T | C | 6 | 36,0297 |
| 2. | 107016135 | замена | A | G | 33 | 159,009 |
| 3. | 154344146 | вставка | tc | tCc | 1 | 3,25208 |
База данных refseq в annovar обозначила 3 категории SNP (файл refgene.variant_function):
| № | Категория | Количество SNP в группе |
| 1. | exonic | 9 |
| 2. | intronic | 42 |
| 3. | UTR3 | 2 |
Гистограммы, отражающие распределение полиморфизмов по качеству прочтения и глубине покрытия приведены на рисунках 5, 6.
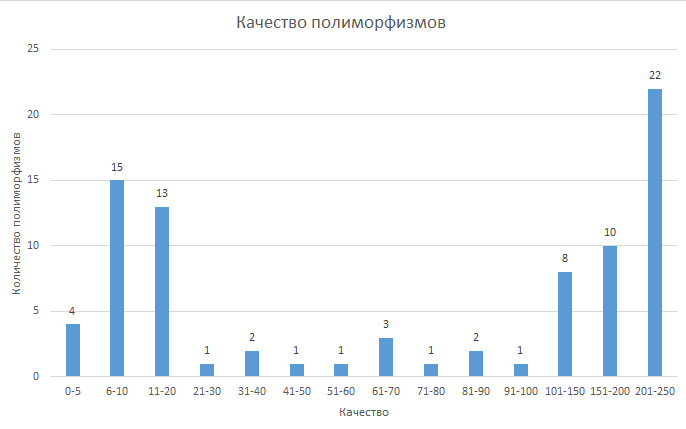
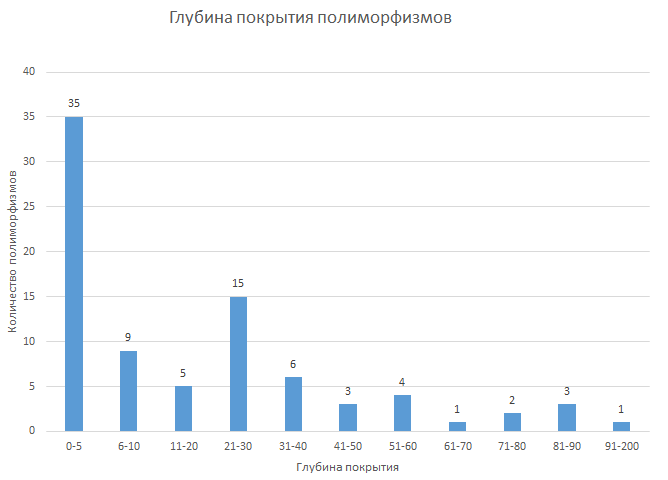
Гены, в которые попали SNP: AIM1, TNFAIP3, OPRM1.
Нуклеотидные замены приведены в том же файле refgene.variant_function:
intronic AIM1 chr6 106961119 106961119 T C het 36.0297 6 intronic AIM1 chr6 106966701 106966701 A G hom 65.5132 4 intronic AIM1 chr6 106966801 106966801 T C het 137.008 25 exonic AIM1 chr6 106967185 106967185 A C het 177.009 38 exonic AIM1 chr6 106967778 106967778 T C het 183.009 26 exonic AIM1 chr6 106967833 106967833 G A het 191.009 25 exonic AIM1 chr6 106968091 106968091 C T het 114.008 19 exonic AIM1 chr6 106968369 106968369 G A het 218.009 41 intronic AIM1 chr6 106974933 106974933 T G het 81.013 5 intronic AIM1 chr6 106975415 106975415 A T het 225.009 46 intronic AIM1 chr6 106976802 106976802 G T het 3.01618 1 intronic AIM1 chr6 106978314 106978314 G A het 183.009 170 intronic AIM1 chr6 106980118 106980118 G C hom 10.4247 1 intronic AIM1 chr6 106982532 106982532 G T hom 11.3429 1 intronic AIM1 chr6 106982560 106982560 G A hom 9.52546 1 intronic AIM1 chr6 106982642 106982642 G A hom 5.46383 1 intronic AIM1 chr6 106982672 106982672 T C hom 6.20226 1 intronic AIM1 chr6 106982931 106982931 G T hom 7.79993 1 intronic AIM1 chr6 106987019 106987019 T C hom 65.9724 4 intronic AIM1 chr6 106987161 106987161 A G hom 221.999 57 exonic AIM1 chr6 106987370 106987370 A C hom 221.999 88 intronic AIM1 chr6 106989212 106989212 G A hom 221.999 36 intronic AIM1 chr6 106991195 106991195 G A hom 180.999 25 intronic AIM1 chr6 106991747 106991747 G A hom 7.79993 1 exonic AIM1 chr6 106992464 106992464 A G hom 221.999 58 intronic AIM1 chr6 106992592 106992592 A T hom 221.999 77 intronic AIM1 chr6 106992844 106992844 C G hom 221.999 25 intronic AIM1 chr6 106998973 106998973 T C het 4.13164 1 intronic AIM1 chr6 106999658 106999658 A T hom 221.999 36 intronic AIM1 chr6 107001456 107001456 A T het 169.009 25 intronic AIM1 chr6 107001581 107001581 T C het 113.008 9 intronic AIM1 chr6 107003444 107003444 G C het 81.0075 10 intronic AIM1 chr6 107003810 107003810 T C het 225.009 65 intronic AIM1 chr6 107006120 107006120 C - hom 15.1077 5 intronic AIM1 chr6 107006336 107006336 G C het 225.009 51 intronic AIM1 chr6 107008422 107008422 C T hom 101.264 5 intronic AIM1 chr6 107008430 107008430 C T hom 116.133 6 intronic AIM1 chr6 107009090 107009090 G A het 94.0077 9 intronic AIM1 chr6 107009119 107009119 A G het 114.008 11 intronic AIM1 chr6 107011930 107011930 T C hom 176.001 12 intronic AIM1 chr6 107014734 107014734 A G hom 11.3429 1 intronic AIM1 chr6 107016127 107016127 G C het 145.008 28 intronic AIM1 chr6 107016135 107016135 A G het 159.009 33 intronic AIM1 chr6 107016136 107016136 T G het 157.009 33 UTR3 AIM1(NM_001624:c.*290T>C) chr6 107016731 107016731 T C het 225.009 70 UTR3 AIM1(NM_001624:c.*400_*401insTT) chr6 107016841 107016841 - TT het 217.468 36 exonic TNFAIP3 chr6 138192607 138192607 G T het 28.0137 29 intronic TNFAIP3 chr6 138192745 138192745 G A het 15.1417 4 intronic TNFAIP3 chr6 138192761 138192761 A G het 5.4626 2 intronic TNFAIP3 chr6 138195693 138195693 T C hom 41.7648 2 intronic TNFAIP3 chr6 138195723 138195723 C G het 65.0073 9 exonic TNFAIP3 chr6 138196066 138196066 T G het 225.009 46 intronic TNFAIP3 chr6 138197331 138197331 A C het 111.008 27 intronic TNFAIP3 chr6 138197824 138197824 C T het 5.46092 2 intronic OPRM1 chr6 154344147 154344147 - C het 3.25208 1 intronic OPRM1 chr6 154344284 154344284 C T hom 9.52546 1 intronic OPRM1 chr6 154357986 154357986 T G hom 11.3429 1 intronic OPRM1 chr6 154357987 154357987 A C hom 11.3429 1 exonic OPRM1 chr6 154360569 154360569 C T het 165.009 23 exonic OPRM1 chr6 154360696 154360696 C T hom 221.999 21 intronic OPRM1 chr6 154362920 154362920 - T het 4.4191 1 intronic OPRM1 chr6 154382542 154382542 A G hom 7.79993 1 intronic OPRM1 chr6 154382572 154382572 G T het 5.46137 2 intronic OPRM1 chr6 154387317 154387317 G T hom 39.765 2 intronic OPRM1 chr6 154392675 154392675 T C hom 11.3429 1 intronic OPRM1 chr6 154402589 154402589 C T hom 11.3429 1 intronic OPRM1 chr6 154406316 154406316 A G hom 10.4247 1 intronic OPRM1 chr6 154411847 154411847 C T het 50.0106 4 exonic OPRM1 chr6 154412385 154412385 G A het 225.009 59 exonic OPRM1 chr6 154414446 154414446 A T hom 221.999 85 exonic OPRM1 chr6 154414563 154414563 A G hom 221.999 84 intronic OPRM1 chr6 154417209 154417209 G T hom 6.20226 1 intronic OPRM1 chr6 154422744 154422744 G A hom 7.79993 1 intronic OPRM1 chr6 154423714 154423714 C G hom 6.20226 1 intronic OPRM1 chr6 154426153 154426153 T C hom 6.20226 1 intronic OPRM1 chr6 154428537 154428537 G A het 73.0074 15 exonic OPRM1 chr6 154428666 154428666 C T het 225.009 26 UTR3 OPRM1(NM_001145286:c.*4A>G) chr6 154428702 154428702 A G hom 221.999 24 UTR3 OPRM1(NM_001145284:c.*162delT) chr6 154429092 154429092 T - hom 18.7798 7 intronic OPRM1 chr6 154431393 154431393 C G hom 221.999 20 UTR3 OPRM1(NM_001145282:c.*19G>A) chr6 154431565 154431565 G A het 218.009 27 intronic OPRM1 chr6 154431742 154431742 G T hom 9.52546 1 intronic OPRM1 chr6 154436236 154436236 T C hom 10.4247 1 intronic OPRM1 chr6 154437252 154437252 A G hom 11.3429 1
Аминокислотные замены приведены файле refgene.exonic_variant_function:
line4 nonsynonymous SNV AIM1:NM_001624:exon2:c.A878C:p.Q293P, chr6 106967185 106967185 A C het 177.009 38 line5 nonsynonymous SNV AIM1:NM_001624:exon2:c.T1471C:p.C491R, chr6 106967778 106967778 T C het 183.009 26 line6 nonsynonymous SNV AIM1:NM_001624:exon2:c.G1526A:p.R509H, chr6 106967833 106967833 G A het 191.009 25 line7 nonsynonymous SNV AIM1:NM_001624:exon2:c.C1784T:p.A595V, chr6 106968091 106968091 C T het 114.008 19 line8 nonsynonymous SNV AIM1:NM_001624:exon2:c.G2062A:p.D688N, chr6 106968369 106968369 G A het 218.009 41 line21 nonsynonymous SNV AIM1:NM_001624:exon7:c.A3587C:p.E1196A, chr6 106987370 106987370 A C hom 221.999 88 line25 synonymous SNV AIM1:NM_001624:exon10:c.A3834G:p.K1278K, chr6 106992464 106992464 A G hom 221.999 58 line47 nonsynonymous SNV TNFAIP3:NM_001270508:exon2:c.G243T:p.K81N, chr6 138192607 138192607 G T het 28.0137 29 line47 nonsynonymous SNV TNFAIP3:NM_006290:exon2:c.G243T:p.K81N, chr6 138192607 138192607 G T het 28.0137 29 line47 nonsynonymous SNV TNFAIP3:NM_001270507:exon2:c.G243T:p.K81N, chr6 138192607 138192607 G T het 28.0137 29 line52 nonsynonymous SNV TNFAIP3:NM_001270508:exon3:c.T380G:p.F127C, chr6 138196066 138196066 T G het 225.009 46 line52 nonsynonymous SNV TNFAIP3:NM_006290:exon3:c.T380G:p.F127C, chr6 138196066 138196066 T G het 225.009 46 line52 nonsynonymous SNV TNFAIP3:NM_001270507:exon3:c.T380G:p.F127C, chr6 138196066 138196066 T G het 225.009 46 line59 stopgain OPRM1:NM_001145279:exon3:c.C169T:p.Q57X, chr6 154360569 154360569 C T het 165.009 23 line59 stopgain OPRM1:NM_001285524:exon2:c.C169T:p.Q57X, chr6 154360569 154360569 C T het 165.009 23 line60 nonsynonymous SNV OPRM1:NM_001145285:exon1:c.C17T:p.A6V, chr6 154360696 154360696 C T hom 221.999 21 line60 nonsynonymous SNV OPRM1:NM_000914:exon1:c.C17T:p.A6V, chr6 154360696 154360696 C T hom 221.999 21 line60 nonsynonymous SNV OPRM1:NM_001145284:exon1:c.C17T:p.A6V, chr6 154360696 154360696 C T hom 221.999 21 line60 nonsynonymous SNV OPRM1:NM_001285522:exon1:c.C17T:p.A6V, chr6 154360696 154360696 C T hom 221.999 21 line60 nonsynonymous SNV OPRM1:NM_001008503:exon1:c.C17T:p.A6V, chr6 154360696 154360696 C T hom 221.999 21 line60 nonsynonymous SNV OPRM1:NM_001285523:exon1:c.C17T:p.A6V, chr6 154360696 154360696 C T hom 221.999 21 line60 nonsynonymous SNV OPRM1:NM_001145279:exon3:c.C296T:p.A99V, chr6 154360696 154360696 C T hom 221.999 21 line60 nonsynonymous SNV OPRM1:NM_001145282:exon1:c.C17T:p.A6V, chr6 154360696 154360696 C T hom 221.999 21 line60 nonsynonymous SNV OPRM1:NM_001008504:exon1:c.C17T:p.A6V, chr6 154360696 154360696 C T hom 221.999 21 line60 nonsynonymous SNV OPRM1:NM_001145286:exon1:c.C17T:p.A6V, chr6 154360696 154360696 C T hom 221.999 21 line60 nonsynonymous SNV OPRM1:NM_001285524:exon2:c.C296T:p.A99V, chr6 154360696 154360696 C T hom 221.999 21 line60 nonsynonymous SNV OPRM1:NM_001008505:exon1:c.C17T:p.A6V, chr6 154360696 154360696 C T hom 221.999 21 line69 synonymous SNV OPRM1:NM_001145285:exon3:c.G942A:p.T314T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_000914:exon3:c.G942A:p.T314T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001145284:exon3:c.G942A:p.T314T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001145283:exon3:c.G942A:p.T314T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001008503:exon3:c.G942A:p.T314T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001285523:exon3:c.G942A:p.T314T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001145279:exon5:c.G1221A:p.T407T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001285528:exon2:c.G642A:p.T214T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001145282:exon3:c.G942A:p.T314T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001145280:exon3:c.G642A:p.T214T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001145287:exon3:c.G642A:p.T214T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001145281:exon3:c.G699A:p.T233T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001008504:exon3:c.G942A:p.T314T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001145286:exon3:c.G942A:p.T314T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001285527:exon2:c.G642A:p.T214T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001285524:exon4:c.G1221A:p.T407T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001008505:exon3:c.G942A:p.T314T, chr6 154412385 154412385 G A het 225.009 59 line69 synonymous SNV OPRM1:NM_001285526:exon3:c.G642A:p.T214T, chr6 154412385 154412385 G A het 225.009 59 line70 nonsynonymous SNV OPRM1:NM_001008505:exon4:c.A1206T:p.Q402H, chr6 154414446 154414446 A T hom 221.999 85 line71 synonymous SNV OPRM1:NM_001008505:exon4:c.A1323G:p.G441G, chr6 154414563 154414563 A G hom 221.999 84 line77 stopgain OPRM1:NM_001145286:exon4:c.C1231T:p.Q411X, chr6 154428666 154428666 C T het 225.009 26
84 SNP имеет RS, что известно из результатов работы с базой snp138.
Частоту найденых SNP можно узнать из результатов работы с базой 1000genomes. Она колеблется от 0,00658946 до 0,987819 (оба значения относятся к интронам), среднее составляет 0,454245787.
Клиническую аннотацию полиморфизмов можно узнать из результатов работы с базой Gwas. Было найдено всего 4 полиморфизма, имеющих клиническую аннотацию из них есть полиморфизмы, приводящие к инсульту, ревматоидному артриту, ишемической болезни сердца и системной красной волчанке.