Предсказание вторичной структуры тРНК по нуклеотидной последовательности проводилось двумя способами - командами einverted и RNAfold. Результаты сравнили с описанием, полученным в практикуме 2 с помощью find_pair. Анализируемая последовательность тРНК имеет PDB ID 1qu2. Она была скачена со страницы сайта RCSB PDB в формате fasta (1qu2.fasta).
Упр.1. Предсказание вторичной структуры тРНК путем поиска инвертированных повторов.
Была запущена программа einverted из пакета EMBOSS:
einverted 1qu2.fasta -gap 0 -thr 0 -match 20 -mis -4
На выходе были получены 2 файла: sequence.inv и sequence.inv.
Из файла sequence.inv была получена информация о найденных комплиментарных парах (рис 1). Указанные в команде параметры были подобранны, с ними удалось определить максимальное количество верных комплиментарных пар.
Из количества предскзанных пар, приведённых в таблице 1 можно сделать вывод, что даже с оптимально выбранными параметрами поиска метод инвертированных повторов плохо справляется с задачей предсказания вторичной структуры.

Упр.2. Предсказание вторичной структуры тРНК по алгоритму Зукера.
Была использована web-версия программы RNAfold. RNAfold полностью предсказала структуру тРНК. Результат:
(((((((..((((.........)))).(((((.......)))))......((((.......)))).)))))))..
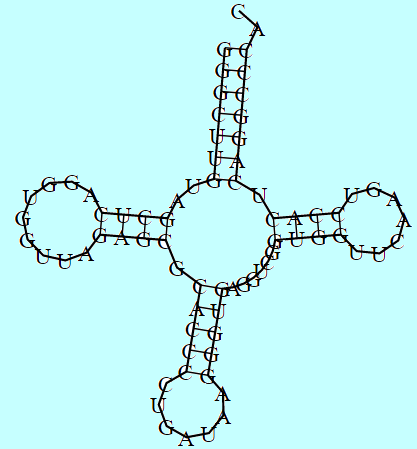
В таблице 1 приведено сравнение результатов работы трёх программ.
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-1-7-3' 5'-66-72-3' Всего 7 пар |
Предсказано 4 пары из 7 реальных и 3 лишних | Предсказано 7 пары из 7 реальных |
| D-стебель | 5'-10-13-3' 5'-22-25-3' Всего 4 пары |
- | Предсказано 4 пары из 4 реальных |
| T-стебель | 5'-50-53-3' 5'-61-64-3' Всего 4 пары |
- | Предсказано 4 пары из 4 реальных |
| Антикодоновый стебель | 5'-27-31-3' 5'-39-43-3' Всего 5 пар |
- | Предсказано 5 пар из 5 реальных |
| Общее число канонических пар нуклеотидов | 19 | 4 | 19 |
Задание 2. Поиск ДНК-белковых контактов в заданной структуре
Исследуемая структура имеет PDB ID 1efa.
Упр.1. Задание и выделение множеств разных атомов в структуре ДНК.
Был написан скрипт задающий в JMol set1 - множество атомов кислорода 2'-дезоксирибозы, set2 - множество остатков фосфорной кислоты и set3 - множество атомов азота остатков азотистых оснований, а также выдающий последовательно изображения: всей структуры, ДНК в проволочной модели, ДНК с выделенными set1, set2, set3.
Упр.1. Задание и выделение множеств разных атомов в структуре ДНК.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 1 | 42 | 43 |
| остатками фосфорной кислоты | 21 | 20 | 41 |
| остатками азотистых оснований со стороны большой бороздки | 5 | 26 | 31 |
| остатками азотистых оснований со стороны малой бороздки | 24 | 41 | 65 |
Поиск контактов производился с помощью скрипта: командой define задавалось нужное множество атомов,
а select выделялись атомы, образующие разыскиваемые контакты.
Из таблицы 2 нетрудно заметить, что количество неполярных ДНК-белковых контактов существенно превосходит количество полярных в остатках 2'-дезоксирибозы и в атотистых основаниях.
Упр.3. Получение популярной схемы ДНК-белковых контактов с помощью программы nucplot.
Схема белковых контактов была получена так же с помощью команды nucplot, в результате которой был получен файл nucplot.ps
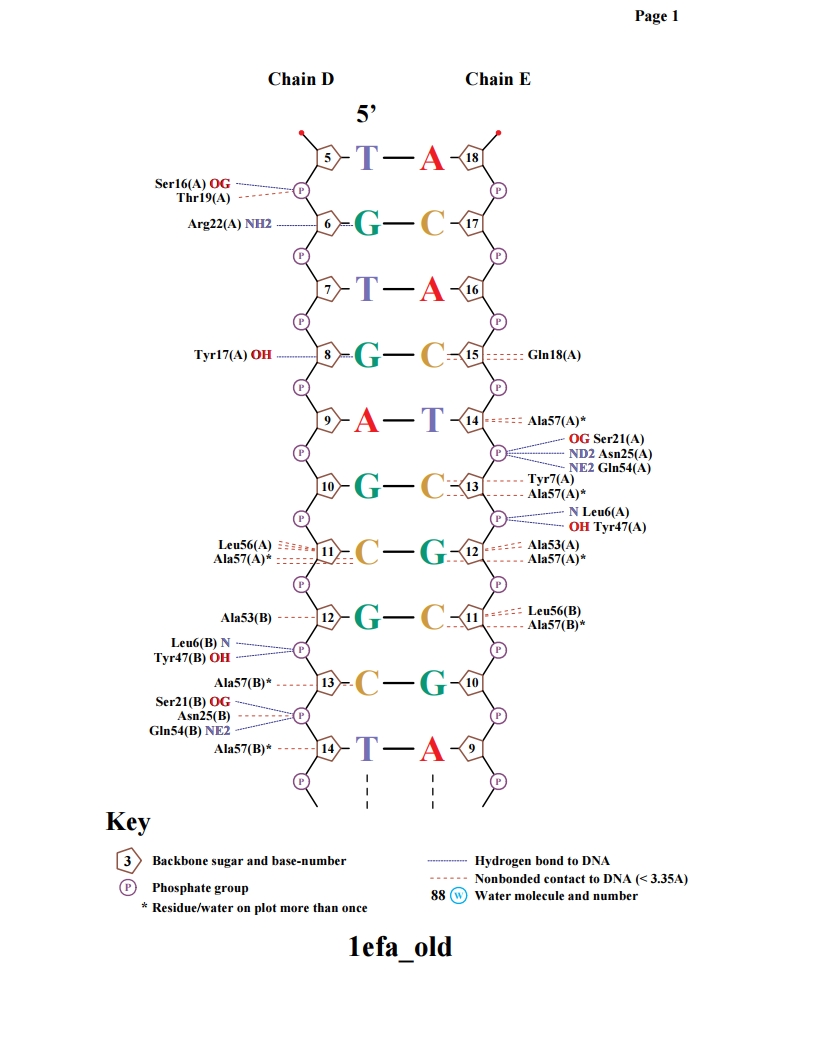
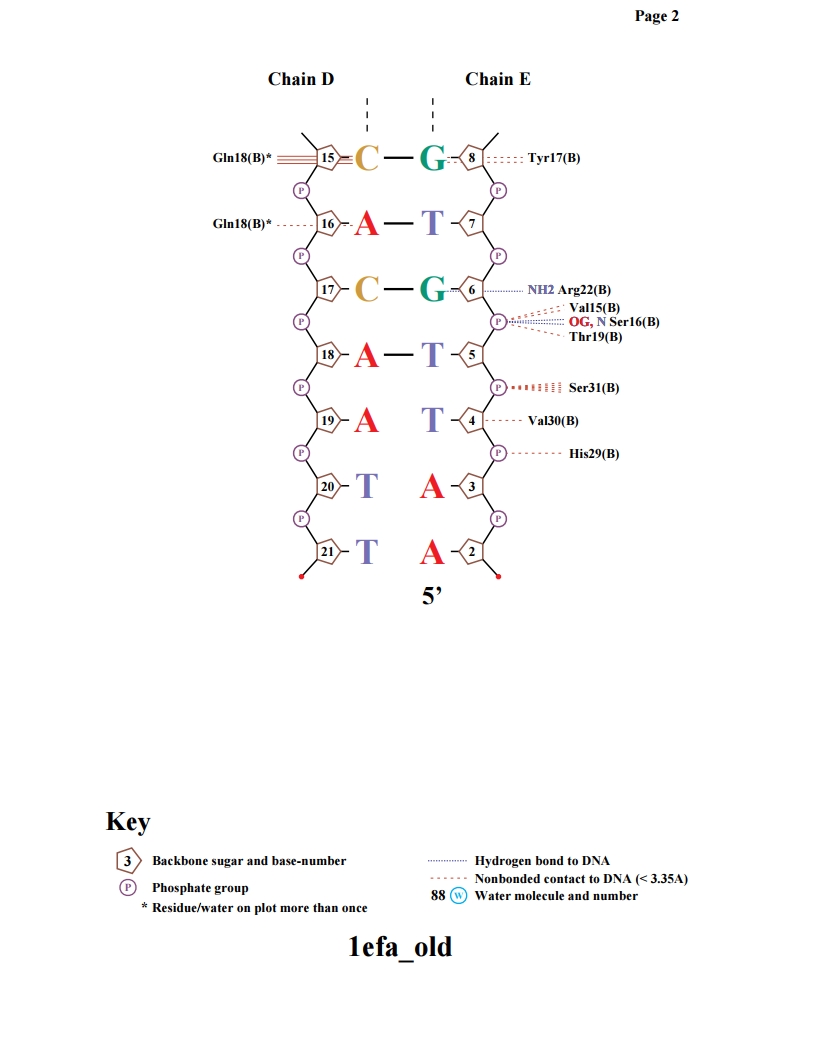
Упр.3. Визуализация наиболее важных ДНК-белковых контактов с помощью Jmol.
Самым большим числом контактов с ДНК обладают аминокислоты Ala57 цепи A и Ala57 цепи B. Ala57(A) вступает в неполярные взаимодействия взаиодействия с атомами азотистых оснований С(11) D-цепи ДНК, G(12), C(13) и c атомами дезоксирибозы при T(14) E-цепи, а также в неполярные взаимодействия с азотистыми основаниями С(11) и G(12). Ala57(B) образует только неполярные взаимодействия с азотистыми основаниями С(11) E-цепи, С(13) и c атомами дезоксирибозы при T(14) D-цепи.
Предположительно, эти аминокислотные остатки являются наиболее важными для распознавания последовательности ДНК, так как, кроме их самых многочисленных контактов с ДНК, эти контакты принадлежат не только остову, но и нескольким азотистым основаниям. Визуализация данных контактов с помощью Jmol приведена на рисунках 2-5.
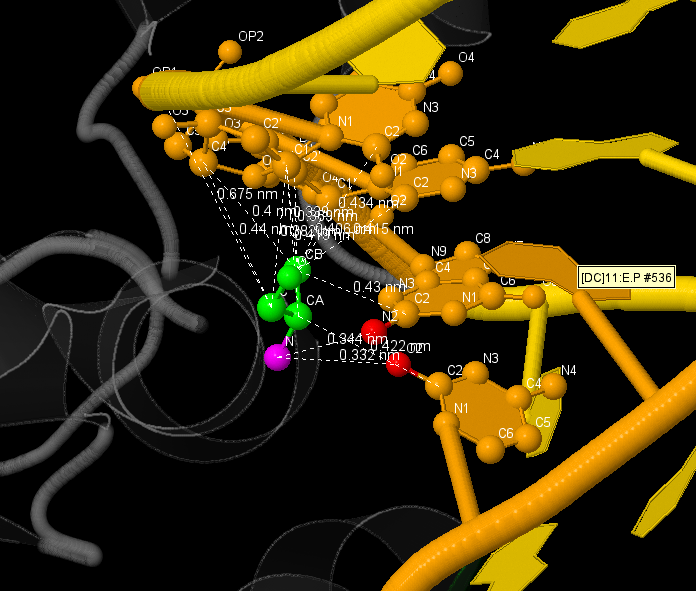
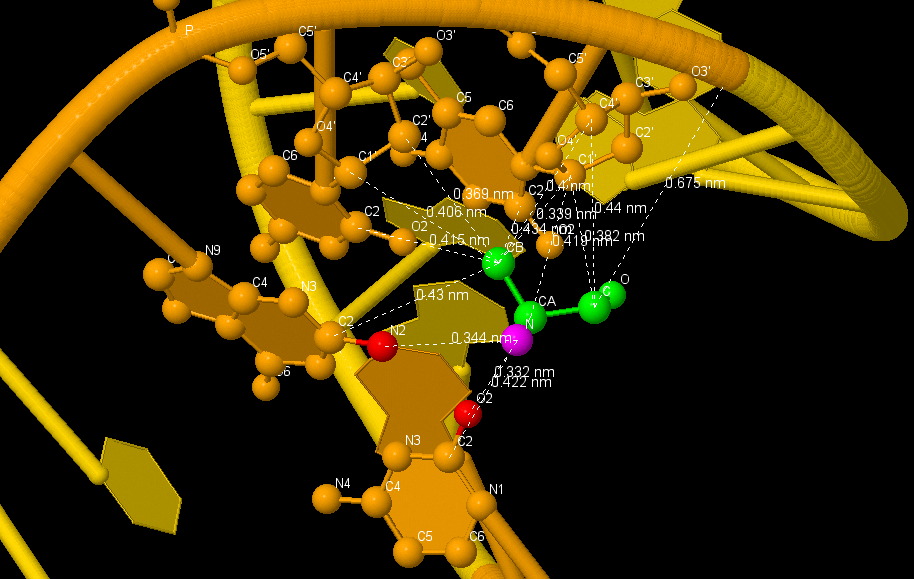
Структура ДНК изображена в жёлто-оранжевом цвете, шариками изображены атомы, основания которых включены во взаимодействие, структура белка - в сером. Ala57(A) изображена в проволочно-шариковой модели в зелёном цвете. На рисунках выделены все связи, имеющие длину менее 4,5Å. Розовым выделен полярный атом Ala57(A), красным - полярные атомы ДНК, вступающие с ним во взаимодействие.
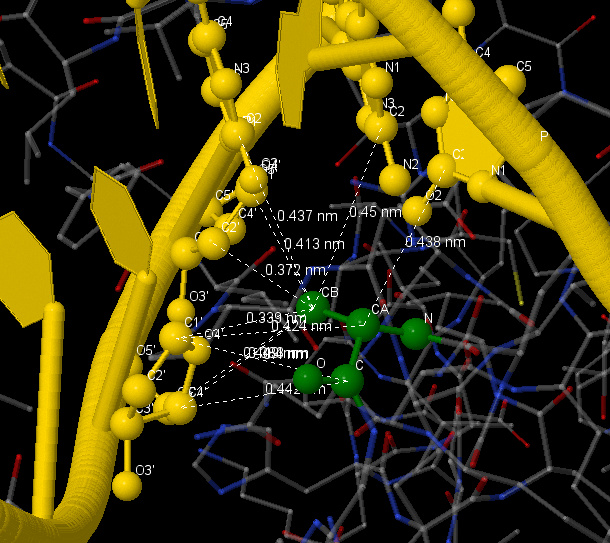
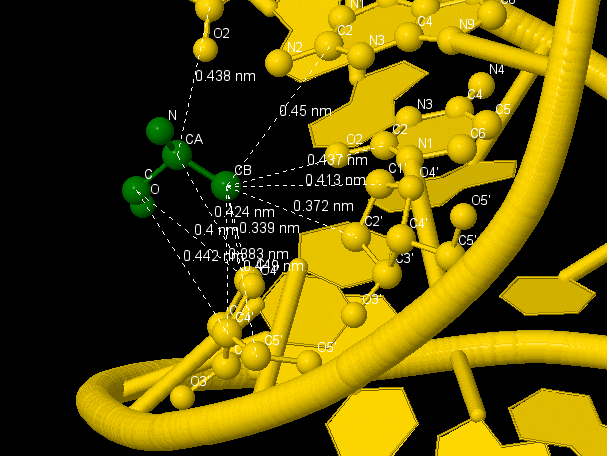
Структура ДНК изображена в жёлтом цвете, шариками изображены атомы, основания которых включены во взаимодействие, структура белка - в проволочной модели расскраске по атомам. Ala57(B) изображена в проволочно-шариковой модели в зелёном цвете. На рисунках выделены все связи, имеющие длину менее 4,5Å.
Ниже приведено изображение локализации Ala57(А) и Ala57(B) отосительно ДНК.
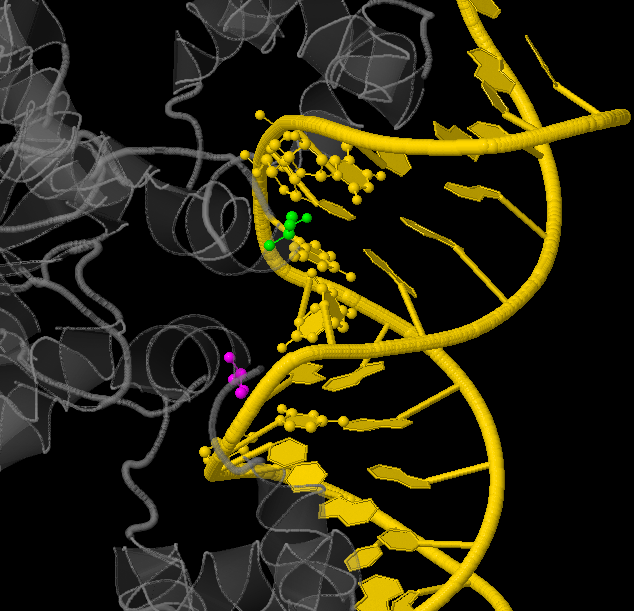
Структура ДНК изображена в жёлтом цвете, шариковой моделью изображены основания включённые во взаимодействие с выделенными аминокислотными статками, структура белка изображена в сером цвете. Зелёным выделен Ala57(А), розовым - Ala57(B).