1. Определение таксономии и функции прочтённой (в практикуме 6) нуклеотидной последовательности
В данном практикуме продолжается работа с последовательностью, полученной методом секвенирования по Сэнгеру и расшифрованная в 6 практикуме. Для её опеделения применим нуклеотидный BLAST. Для начала воспользуемся blastn , так как мы не знаем о существовании гомологичных ей последовательностей в природе и базе данных. Параметры blastn оставим по умолчанию. Результат поиска был следующим:
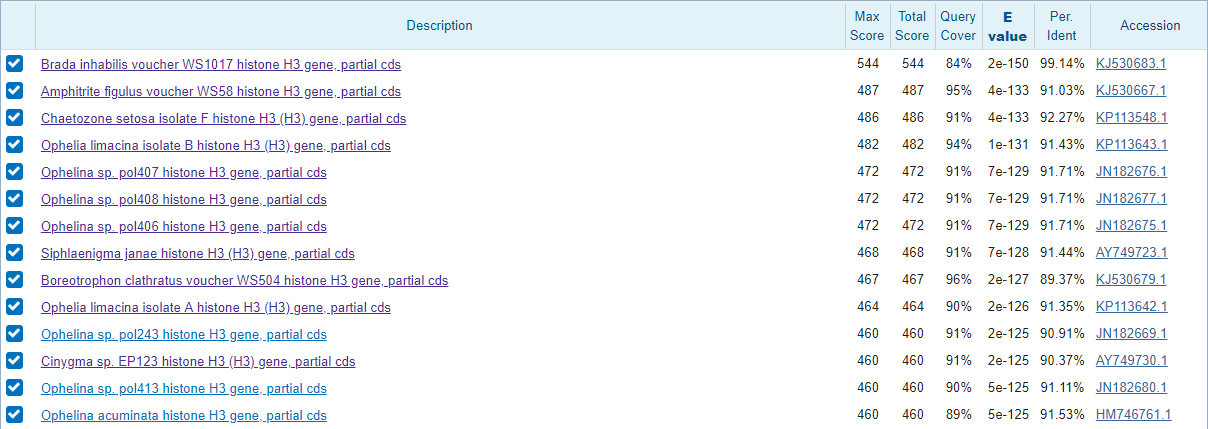
Можно заметить, что все приведённые выше находки кодируют гистоны H3, который участвует в образовании структуры нуклеосом, его переменные состояния модификации, как полагают, играют роль в динамической и долгосрочной регуляции генов. Поиск с помощью megablast выдал те же самые находки с несущественными различиями в порядке расположения. Самой лучшей находкой в обоих сеансах поиска оказалась последовательность, кодирующая гистон H3 у Brada inhabilis (Eukaryota; Opisthokonta; Metazoa; Eumetazoa; Bilateria; Protostomia; Spiralia; Lophotrochozoa; Annelida; Polychaeta; Palpata; Canalipalpata; Flabelligerida; Flabelligeridae; Brada). Я рассмотрела остальные первые 10 находок - все они, за исключением 8 и 9, также относятся к кольчатым червям, классу Polychaeta (8 - членистоногим, 9 - моллюскам), отсюда можно сделать вывод, что данная последовательность, скорее всего, взята из организма, принадлежащего к этому классу. Установить семейство и род оказалось проблематично, так как представителей, чьи последовательности находятся в первой десятке и имеют близкие значения показателей E-value, веса, процента идентичности и покрытия, относятся к разным семействам, а конкретно к семействам Scolecida и Palpata. Интересно ещё и то, что вид Brada inhabilis встречается ещё и на 26 месте в списке находок, после после последовательностей других семейств и даже других классов. Из этого всего можно сделать вывод, что последовательность, кодирующая гистон H3 - консервативна, и мы можем определить точно только класс (Polychaeta).
2. Сравнение списков находок нуклеотидных последовательностей тремя разными вариантами blast
В данном задании нужно было сравнить списки находок megablast, blastn (с параметрами по умолчанию) и blastn (с чувствительными параметрами). Для исследования была взята последовательность из задания №1 и одна последовательность из CDS вируса из практикума 7. Параметры, используемые при каждом запуске BLAST, приведены в Таблице 1.
| Параметры BLAST | megablast | blastn (по умолч.) | blastn (чувствит.) |
| Max target sequences | 1000 | 1000 | 1000 |
| Short queries | ✓ | ✓ | ✓ |
| Expect threshold | 1 | 1 | 1 |
| Word size | 28 | 11 | 7 |
| Max matches in a query range | 0 | 0 | 0 |
| Match/Mismatch Scores | 1, -2 | 2, -3 | 1, -1 |
| Gap Costs | Linear | Existence: 5 Extension: 2 | Existence: 0 Extension: 2 |
| Filter | Low complexity regions | Low complexity regions | Low complexity regions |
| Mask | Mask for lookup table only | Mask for lookup table only | Mask for lookup table only |
Для начала сравнивали по гену из задания 1. Без ограничений на организмы получилось слишком много результатов, поэтому было решено ограничить их по классу Polychaeta. Количество находок при каждом алгоритме приведено в Таблице 2.
Как видно из таблицы, результатов поиска первой последовательности всё равно оказалось много, и они не сильно различаются между собой.
| последовательность, кодирующая гистон Н3 | CDS Staphylococcus phage PMBT8 | |
| megablast | 594 | 3 |
| blastn (по умолч.) | 1027 | 39 |
| blastn (чувствит.) | 1028 | 523 |
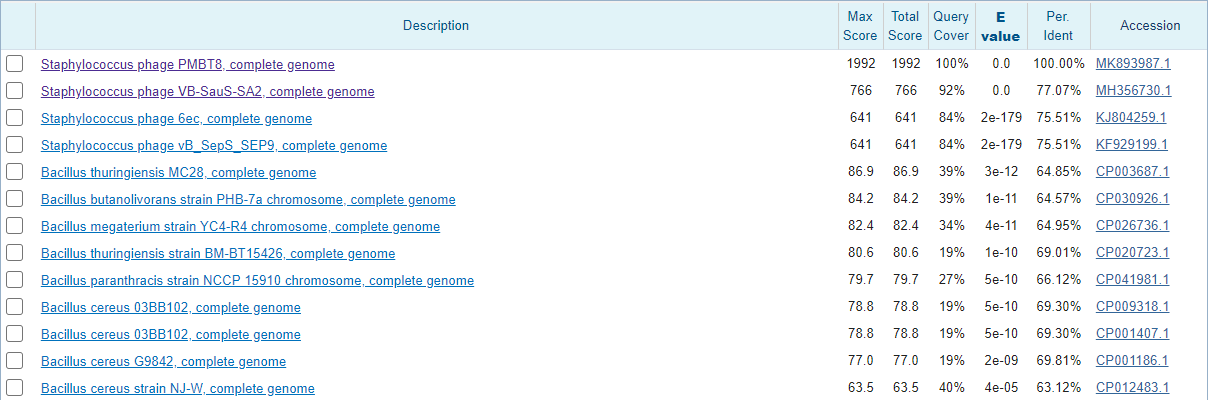
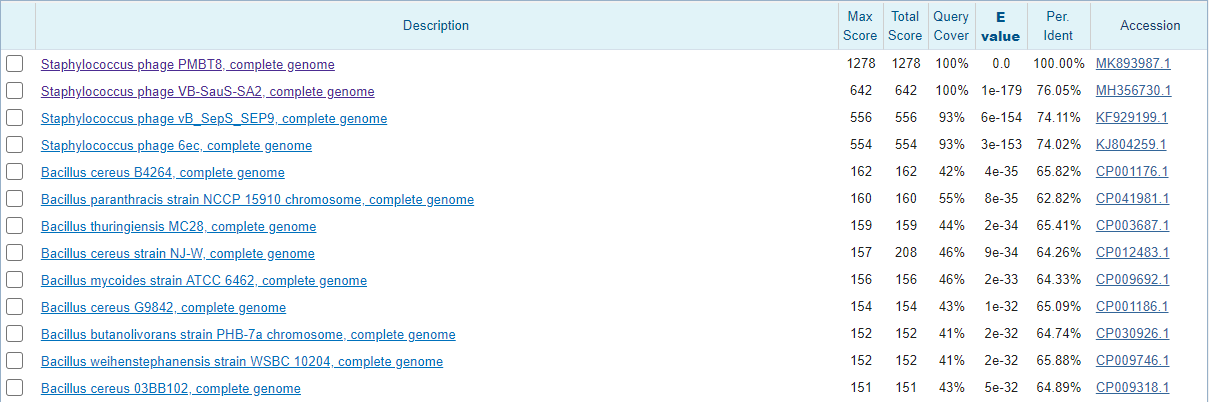
Гораздо интереснее дело обстоит с последовательностью CDS вируса. При поиске CDS Staphylococcus phage PMBT8 всеми тремя способами удалось найти данный вирус и родственные ему вирусы, принадлежащие роду Sextaecvirus, которых оказалось всего 3. С помощью megablast удалось найти 2 из них (Рис.2), третий (Staphylococcus phage VB-SauS-SA2) нашёлся с помощью blastn (Рис.3,4). Blastn добавил много послетовательностей, принадлежащих к разным типам, напимер, при запуске с параметрами по умолчанию добавились такие типы как Firmicutes, Streptophyt, Chordata. При изменении параметров добавилось ещё больше новых последовательностей из других классов. Сразу после рода Sextaecvirus лучшие показатели по выравниванию имеет род Bacillus (Рис.3,4).
3. Гомологи трех белков в неаннотированном геноме
В данном задании необходимо было проверить наличие гомологов трёх выбранных белков в неаннотированном геноме Amoeboaphelidium protococcarum. Для исследования были выбраны белки:
Цитохром С - митохондриальный белок, основной функцией которого является перенос электронов между комплексами III и IV (Цитохром c-оксидаза) дыхательной цепи митохондрий. Цитохром c является консервативным белком, найденным у растений, животных и многих простейших.
Гистон H3.1, как уже было сказано, является одним из пяти основных гистонов, участвующих в образовании структуры хроматина в эукариотических клетках. Он обладаут основным глобулярным доменом и длинным N-концевым хвостом, является наиболее сильно посттрансляционно модифицированным из пяти гистонов. Как было выяснено в 1 задании, является очень консервативным.
Фосфоглицераткиназа - фермент, катализирующий обратимую реакцию переноса фосфатной группы от 1,3-дифосфоглицериновой кислоты к АДФ, в результате которой образуются 3-фосфоглицерат и АТФ. Фосфоглицераткиназа присутствует во всех живых организмах и является одним из двух АТФ-производящих ферментов в процессе гликолиза. В рамках глюконеогенеза ФГК катализирует обратную реакцию, в результате чего образуются АДФ и 1,3-дифосфоглицерат. Он находится во всех живых организмах, и его последовательность была высоко консервативны в процессе эволюции.
Сначала в Swiss-Prot были взяты их ID, с помощью команд:
seqret sw:cyc_human cytochrome.fasta
seqret sw:h31_human histone.fasta
seqret sw:pgk1_human phosphoglycerate_kinase_1.fasta
были получены их последовательности для поиска гомологов: cytochrome.fasta, histone.fasta, phosphoglycerate_kinase_1.fasta.
Далее с помощью команды makeblastdb -in /P/y18/term3/block2/X5.fasta -out ./X5 -dbtype nucl была создана локальная база данных генома Amoeboaphelidium protococcarum в файле X5.fasta.
Затем при использовании локального BLAST+ на kodomo и с помощью команд:
tblastn -query cytochrome.fasta -db X5.fasta -db_gencode 6 > cytochrome_hits.fasta
tblastn -query histone.fasta -db X5.fasta -db_gencode 6 > histone_hits.fasta
tblastn -query phosphoglycerate_kinase_1.fasta -db X5.fasta -db_gencode 6 > phosphoglycerate_kinase_1_hits.fasta
был произведён поиск гомологов.
При поиске гомологов Цитохрома С было найдено 4 последовательности, две из которых можно назвать гомологами цитохрома (scaffold-282, scaffold-287). Покрытия 97% и 73% соответственно:
> scaffold-282
Length=195711
Score = 146 bits (368), Expect = 7e-41, Method: Compositional matrix adjust.
Identities = 76/143 (53%), Positives = 85/143 (59%), Gaps = 40/143 (28%)
Frame = -1
Query 2 GDVEKGKKIFIMKCSQCHTVEK-------------------------------------- 23
G+ +KG K+F +C+QCHT+E
Sbjct 48693 GNTKKGAKLFQTRCAQCHTLEAVRICIFIICMSDIQFIYYHVGTTAQAGYDPIHLTSLLQ 48514
Query 24 --GGKHKTGPNLHGLFGRKTGQAPGYSYTAANKNKGIIWGEDTLMEYLENPKKYIPGTKM 81
H GPNLHGLFGRKTGQA GYSYTAAN NKGI W EDTL EYLENPKKYIPGTKM
Sbjct 48513 LASSLHYVGPNLHGLFGRKTGQAEGYSYTAANVNKGITWSEDTLFEYLENPKKYIPGTKM 48334
Query 82 IFVGIKKKEERADLIAYLKKATN 104
F G+KK EERADLIAYLK+AT+
Sbjct 48333 AFAGLKKPEERADLIAYLKQATS 48265
Score = 25.4 bits (54), Expect = 7.8, Method: Composition-based stats.
Identities = 9/23 (39%), Positives = 17/23 (74%), Gaps = 0/23 (0%)
Frame = +3
Query 80 KMIFVGIKKKEERADLIAYLKKA 102
K+++VG+ +ERAD++ L K+
Sbjct 185676 KLLYVGLPSVDERADILRTLSKS 185744
> scaffold-287
Length=687983
Score = 137 bits (346), Expect = 7e-38, Method: Compositional matrix adjust.
Identities = 65/78 (83%), Positives = 68/78 (87%), Gaps = 0/78 (0%)
Frame = +1
Query 27 HKTGPNLHGLFGRKTGQAPGYSYTAANKNKGIIWGEDTLMEYLENPKKYIPGTKMIFVGI 86
H GPNLHGLFGRKTGQA GYSYTAAN NKGI W EDTL EYLENPKKYIPGTKM F G+
Sbjct 469879 HYVGPNLHGLFGRKTGQAEGYSYTAANVNKGITWSEDTLFEYLENPKKYIPGTKMAFAGL 470058
Query 87 KKKEERADLIAYLKKATN 104
KK EERADLIAYLK+AT+
Sbjct 470059 KKPEERADLIAYLKQATS 470112
Score = 33.5 bits (75), Expect = 0.010, Method: Composition-based stats.
Identities = 11/21 (52%), Positives = 17/21 (81%), Gaps = 0/21 (0%)
Frame = +3
Query 2 GDVEKGKKIFIMKCSQCHTVE 22
G+ +KG K+F +C+QCHT+E
Sbjct 469674 GNTKKGAKLFQTRCAQCHTLE 469736
Score = 25.4 bits (54), Expect = 7.8, Method: Composition-based stats.
Identities = 9/23 (39%), Positives = 17/23 (74%), Gaps = 0/23 (0%)
Frame = +2
Query 80 KMIFVGIKKKEERADLIAYLKKA 102
K+++VG+ +ERAD++ L K+
Sbjct 367820 KLLYVGLPSVDERADILRTLSKS 367888
> scaffold-358
Length=194751
Score = 25.8 bits (55), Expect = 4.7, Method: Composition-based stats.
Identities = 9/17 (53%), Positives = 11/17 (65%), Gaps = 0/17 (0%)
Frame = -3
Query 47 YSYTAANKNKGIIWGED 63
YS A +N GI+WG D
Sbjct 170155 YSDVAIQRNHGIVWGND 170105
> scaffold-170
Length=449377
Score = 25.4 bits (54), Expect = 7.6, Method: Composition-based stats.
Identities = 14/39 (36%), Positives = 21/39 (54%), Gaps = 6/39 (15%)
Frame = -2
Query 45 PGYSYTAANKNKGIIWGEDTLMEYLENPKKYIPGTKMIF 83
PGY Y A + N G ++GE + Y N + TK++F
Sbjct 364047 PGYFYAANSFNAGPVYGE---LLYFPNESR---DTKLVF 363949
В случае гистона H3.1 ответ также будет положительный. Из всех 14-ти последовательностей, найденных локальным BLAST, гомологами являются 4ипоследовательности с покрытиями: scaffold-104 (99%), 126 (99%), 153 (71%), unplaced-984 (64,7%).
> scaffold-104
Length=736462
Score = 254 bits (650), Expect = 4e-78, Method: Compositional matrix adjust.
Identities = 129/136 (95%), Positives = 130/136 (96%), Gaps = 1/136 (1%)
Frame = +3
Query 1 MARTKQTARKSTGGKAPRKQLATKAARKSAPATGGVKKPHRYRPGTVALREIRRYQKSTE 60
MARTKQTARKSTGGKAPRKQLATKA RKS PATGGVKKPHRYRPGTVALREIRRYQKSTE
Sbjct 185496 MARTKQTARKSTGGKAPRKQLATKA-RKSTPATGGVKKPHRYRPGTVALREIRRYQKSTE 185672
Query 61 LLIRKLPFQRLVREIAQDFKTDLRFQSSAVMALQEACEAYLVGLFEDTNLCAIHAKRVTI 120
LLIRKLPFQRLVREIAQDFKTDLRFQSSA+ ALQEA EAYLV LFEDTNLCAIHAKRVTI
Sbjct 185673 LLIRKLPFQRLVREIAQDFKTDLRFQSSAIGALQEAAEAYLVSLFEDTNLCAIHAKRVTI 185852
Query 121 MPKDIQLARRIRGERA 136
PKDIQLARRIRGERA
Sbjct 185853 FPKDIQLARRIRGERA 185900
Score = 254 bits (650), Expect = 4e-78, Method: Compositional matrix adjust.
Identities = 129/136 (95%), Positives = 130/136 (96%), Gaps = 1/136 (1%)
Frame = -2
Query 1 MARTKQTARKSTGGKAPRKQLATKAARKSAPATGGVKKPHRYRPGTVALREIRRYQKSTE 60
MARTKQTARKSTGGKAPRKQLATKA RKS PATGGVKKPHRYRPGTVALREIRRYQKSTE
Sbjct 178362 MARTKQTARKSTGGKAPRKQLATKA-RKSTPATGGVKKPHRYRPGTVALREIRRYQKSTE 178186
Query 61 LLIRKLPFQRLVREIAQDFKTDLRFQSSAVMALQEACEAYLVGLFEDTNLCAIHAKRVTI 120
LLIRKLPFQRLVREIAQDFKTDLRFQSSA+ ALQEA EAYLV LFEDTNLCAIHAKRVTI
Sbjct 178185 LLIRKLPFQRLVREIAQDFKTDLRFQSSAIGALQEAAEAYLVSLFEDTNLCAIHAKRVTI 178006
Query 121 MPKDIQLARRIRGERA 136
PKDIQLARRIRGERA
Sbjct 178005 FPKDIQLARRIRGERA 177958
> scaffold-126
Length=92370
Score = 254 bits (650), Expect = 5e-78, Method: Compositional matrix adjust.
Identities = 129/136 (95%), Positives = 130/136 (96%), Gaps = 1/136 (1%)
Frame = -3
Query 1 MARTKQTARKSTGGKAPRKQLATKAARKSAPATGGVKKPHRYRPGTVALREIRRYQKSTE 60
MARTKQTARKSTGGKAPRKQLATKA RKS PATGGVKKPHRYRPGTVALREIRRYQKSTE
Sbjct 85096 MARTKQTARKSTGGKAPRKQLATKA-RKSTPATGGVKKPHRYRPGTVALREIRRYQKSTE 84920
Query 61 LLIRKLPFQRLVREIAQDFKTDLRFQSSAVMALQEACEAYLVGLFEDTNLCAIHAKRVTI 120
LLIRKLPFQRLVREIAQDFKTDLRFQSSA+ ALQEA EAYLV LFEDTNLCAIHAKRVTI
Sbjct 84919 LLIRKLPFQRLVREIAQDFKTDLRFQSSAIGALQEAAEAYLVSLFEDTNLCAIHAKRVTI 84740
Query 121 MPKDIQLARRIRGERA 136
PKDIQLARRIRGERA
Sbjct 84739 FPKDIQLARRIRGERA 84692
Score = 64.7 bits (156), Expect = 2e-12, Method: Compositional matrix adjust.
Identities = 35/37 (95%), Positives = 35/37 (95%), Gaps = 1/37 (3%)
Frame = +2
Query 1 MARTKQTARKSTGGKAPRKQLATKAARKSAPATGGVK 37
MARTKQTARKSTGGKAPRKQLATK ARKS PATGGVK
Sbjct 92261 MARTKQTARKSTGGKAPRKQLATK-ARKSTPATGGVK 92368
> unplaced-984
Length=389
Score = 191 bits (485), Expect = 1e-62, Method: Compositional matrix adjust.
Identities = 93/98 (95%), Positives = 94/98 (96%), Gaps = 0/98 (0%)
Frame = +2
Query 39 PHRYRPGTVALREIRRYQKSTELLIRKLPFQRLVREIAQDFKTDLRFQSSAVMALQEACE 98
PHRYRPGTVALREIRRYQKSTELLIRKLPFQRLVREIAQDFKTDLRFQSSA+ ALQEA E
Sbjct 2 PHRYRPGTVALREIRRYQKSTELLIRKLPFQRLVREIAQDFKTDLRFQSSAIGALQEAAE 181
Query 99 AYLVGLFEDTNLCAIHAKRVTIMPKDIQLARRIRGERA 136
AYLV LFEDTNLCAIHAKRVTI PKDIQLARRIRGERA
Sbjct 182 AYLVSLFEDTNLCAIHAKRVTIFPKDIQLARRIRGERA 295
> scaffold-153
Length=574
Score = 170 bits (430), Expect = 1e-53, Method: Compositional matrix adjust.
Identities = 84/89 (94%), Positives = 85/89 (96%), Gaps = 0/89 (0%)
Frame = -2
Query 48 ALREIRRYQKSTELLIRKLPFQRLVREIAQDFKTDLRFQSSAVMALQEACEAYLVGLFED 107
ALREIRRYQKSTELLIRKLPFQRLVREIAQDFKTDLRFQSSA+ ALQEA EAYLV LFED
Sbjct 573 ALREIRRYQKSTELLIRKLPFQRLVREIAQDFKTDLRFQSSAIGALQEAAEAYLVSLFED 394
Query 108 TNLCAIHAKRVTIMPKDIQLARRIRGERA 136
TNLCAIHAKRVTI PKDIQLARRIRGERA
Sbjct 393 TNLCAIHAKRVTIFPKDIQLARRIRGERA 307
> scaffold-70
Length=635
Score = 66.6 bits (161), Expect = 3e-14, Method: Compositional matrix adjust.
Identities = 36/38 (95%), Positives = 36/38 (95%), Gaps = 1/38 (3%)
Frame = +3
Query 1 MARTKQTARKSTGGKAPRKQLATKAARKSAPATGGVKK 38
MARTKQTARKSTGGKAPRKQLATK ARKS PATGGVKK
Sbjct 525 MARTKQTARKSTGGKAPRKQLATK-ARKSTPATGGVKK 635
> unplaced-368
Length=899
Score = 63.9 bits (154), Expect = 5e-13, Method: Compositional matrix adjust.
Identities = 35/37 (95%), Positives = 35/37 (95%), Gaps = 1/37 (3%)
Frame = +1
Query 1 MARTKQTARKSTGGKAPRKQLATKAARKSAPATGGVK 37
MARTKQTARKSTGGKAPRKQLATK ARKS PATGGVK
Sbjct 790 MARTKQTARKSTGGKAPRKQLATK-ARKSTPATGGVK 897
> scaffold-157
Length=706690
Score = 31.6 bits (70), Expect = 0.11, Method: Composition-based stats.
Identities = 19/61 (31%), Positives = 29/61 (48%), Gaps = 7/61 (11%)
Frame = +1
Query 21 LATKAARKSAPATGGVKKPHRYRPGTVALREIRRYQKSTELLIRKLPFQRLVREIAQDFK 80
LATK A ++ ++ HRY P V +QKS + + L +QR + +FK
Sbjct 253978 LATKTQFTQAASSSDLQTRHRYPPNFV-------HQKSNSVYVSSLTYQRRNSTLYANFK 254136
Query 81 T 81
T
Sbjct 254137 T 254139
> unplaced-13
Length=505
Score = 27.7 bits (60), Expect = 1.5, Method: Compositional matrix adjust.
Identities = 15/42 (36%), Positives = 20/42 (48%), Gaps = 2/42 (5%)
Frame = +3
Query 78 DFKTDLR--FQSSAVMALQEACEAYLVGLFEDTNLCAIHAKR 117
D+ DLR F V + E + + FEDTN+ A KR
Sbjct 261 DYMVDLRNSFNVRFVFGVLTTYEKWRILWFEDTNIAATETKR 386
> scaffold-693
Length=1268102
Score = 28.1 bits (61), Expect = 1.7, Method: Composition-based stats.
Identities = 18/61 (30%), Positives = 28/61 (46%), Gaps = 7/61 (11%)
Frame = +2
Query 21 LATKAARKSAPATGGVKKPHRYRPGTVALREIRRYQKSTELLIRKLPFQRLVREIAQDFK 80
LATK A ++ ++ HRY P V +QKS + + +QR + +FK
Sbjct 1197674 LATKTQFTQAASSSDLQTRHRYPPNFV-------HQKSNSVYVSSPTYQRRNSTLYANFK 1197832
Query 81 T 81
T
Sbjct 1197833 T 1197835
> scaffold-17
Length=2125590
Score = 28.1 bits (61), Expect = 1.8, Method: Composition-based stats.
Identities = 29/85 (34%), Positives = 37/85 (44%), Gaps = 15/85 (18%)
Frame = -1
Query 10 KSTGGKAPRKQLA----TKAARKSAP----ATGGVKKPHRYRPGTV--ALRE----IRRY 55
KS G K QLA T AR S G K + GTV L E I +
Sbjct 784575 KSIGRKEKISQLAMSESTTKARSSVDNIDDQVNGKVKAQKVYSGTVYNVLGETGDLINHF 784396
Query 56 QKSTELL-IRKLPFQRLVREIAQDF 79
+T L+ IRK+PF L+ + QD+
Sbjct 784395 APNTSLISIRKVPFDNLMASLVQDY 784321
> unplaced-752
Length=253
Score = 26.2 bits (56), Expect = 2.4, Method: Compositional matrix adjust.
Identities = 10/28 (36%), Positives = 17/28 (61%), Gaps = 0/28 (0%)
Frame = +1
Query 63 IRKLPFQRLVREIAQDFKTDLRFQSSAV 90
+ K+P RLV++ + T+L+ SAV
Sbjct 115 LNKVPISRLVKQASSILSTNLQISQSAV 198
> scaffold-168
Length=4885
Score = 26.9 bits (58), Expect = 3.7, Method: Composition-based stats.
Identities = 15/42 (36%), Positives = 20/42 (48%), Gaps = 2/42 (5%)
Frame = -1
Query 78 DFKTDLR--FQSSAVMALQEACEAYLVGLFEDTNLCAIHAKR 117
D+ DLR F V + E + + FEDTN+ A KR
Sbjct 3130 DYMVDLRNSFNVRFVFGVLTTYEKWRILWFEDTNIAATETKR 3005
> scaffold-242
Length=71096
Score = 26.6 bits (57), Expect = 5.4, Method: Composition-based stats.
Identities = 18/58 (31%), Positives = 28/58 (48%), Gaps = 0/58 (0%)
Frame = +2
Query 13 GGKAPRKQLATKAARKSAPATGGVKKPHRYRPGTVALREIRRYQKSTELLIRKLPFQR 70
G P+ LA K A T +K P G + +R I++YQ + L+ + LPF +
Sbjct 69701 GRAVPQ*LLAGKDACG*PSTTLVLKVPPMKSYGWIVIRRIQKYQPRSTLICQCLPFSQ 69874
> scaffold-100
Length=762135
Score = 26.2 bits (56), Expect = 7.7, Method: Composition-based stats.
Identities = 17/61 (28%), Positives = 29/61 (48%), Gaps = 1/61 (2%)
Frame = -1
Query 58 STELLIRKLPFQRLVREIAQDFKTDLRFQSSAVMALQEACEAYLVGLFEDTNLCAIHAKR 117
S +L I + RL + + + L Q + L ++ + +F+D LC +HAKR
Sbjct 513690 SNKLYIADRRYSRL-KTVHKPLGQSLFVQDFIIFTLYLRRSSFYMFIFQDEVLCHLHAKR 513514
При поиске фосфоглицераткиназы нашлось 3 последовательности, из которых 2 являются гомологами (scaffold-693, scaffold-157), так как, несмотря на небольшие значения Identities и Positives и наличие гэпов, процент покрытия у обоих составляет 98.
> scaffold-693
Length=1268102
Score = 533 bits (1373), Expect = 5e-170, Method: Compositional matrix adjust.
Identities = 266/435 (61%), Positives = 329/435 (76%), Gaps = 25/435 (6%)
Frame = -1
Query 1 MSLSNKLTLDKLDVKGKRVVMR-----------------------VDFNVPMKNNQITNN 37
M + NKL L +DVKGKRV+MR VDFNVP + +I+NN
Sbjct 1173191 MKIGNKLGLKDIDVKGKRVLMRQVDSHLN*C*LINIIHVLY*TCSVDFNVPFADGKISNN 1173012
Query 38 QRIKAAVPSIKFCLDNGAKSVVLMSHLGRPDGVPMPDKYSLEPVAVELKSLLGKDVLFLK 97
QRI AA+PSI+ CL NGAK+VVLMSHLGRPDG + KYSL+PVA E++ LL + V FL+
Sbjct 1173011 QRIVAAIPSIEHCLKNGAKAVVLMSHLGRPDG-KVVSKYSLKPVAAEVERLLKRKVTFLE 1172835
Query 98 DCVGPEVEKACANPAAGSVILLENLRFHVEEEGKGKDASGNKVKAEPAKIEAFRASLSKL 157
DCVG +VE C N A G VILLENLRFH+EEEG KD +GNKVKA +E FR SLS+L
Sbjct 1172834 DCVGQKVEDQCKNAANGEVILLENLRFHIEEEGSVKDEAGNKVKANKEDVEKFRQSLSQL 1172655
Query 158 GDVYVNDAFGTAHRAHSSMVGVNLPQKAGGFLMKKELNYFAKALESPERPFLAILGGAKV 217
GDVYVNDAFGTAHRAHSS+VG+ L +A GFLMKKEL++F ALE+P+RPFLAI+GGAKV
Sbjct 1172654 GDVYVNDAFGTAHRAHSSVVGIKLETRAAGFLMKKELDFFGMALENPQRPFLAIIGGAKV 1172475
Query 218 ADKIQLINNMLDKVNEMIIGGGMAFTFLKVLNNMEIGTSLFDEEGAKIVKDLMSKAEKNG 277
+DKIQLI N+LDKVN MIIGGGMAFTF KV++ + IG SLFDEEG KIVK L+ KA+
Sbjct 1172474 SDKIQLIENLLDKVNAMIIGGGMAFTFKKVVDGVSIGKSLFDEEGGKIVKKLVEKAKSKN 1172295
Query 278 VKITLPVDFVTADKFDENAKTGQATVASGIPAGWMGLDCGPESSKKYAEAVTRAKQIVWN 337
V++ PVD+VTA KF ++A+ G A SGI WMGLD G ES KK+ E V ++K I+WN
Sbjct 1172294 VELVFPVDYVTASKFAKDAEVGYADDKSGIKDEWMGLDVGSESVKKFKEVVLKSKTILWN 1172115
Query 338 GPVGVFEWEAFARGTKALMDEVVKATSR-GCITIIGGGDTATCCAKWNTEDKVSHVSTGG 396
GP+GVFE+++F+ GT+ ++D V++AT++ G I+I+GGGDTAT +KWN +DKVSHVSTGG
Sbjct 1172114 GPMGVFEFDSFSAGTRGVLDAVIEATAQNGAISIVGGGDTATAVSKWNADDKVSHVSTGG 1171935
Query 397 GASLELLEGKVLPGV 411
G+S+EL EGK LPGV
Sbjct 1171934 GSSVELAEGKELPGV 1171890
> scaffold-157
Length=706690
Score = 530 bits (1364), Expect = 8e-169, Method: Compositional matrix adjust.
Identities = 265/435 (61%), Positives = 327/435 (75%), Gaps = 25/435 (6%)
Frame = -3
Query 1 MSLSNKLTLDKLDVKGKRVVMR-----------------------VDFNVPMKNNQITNN 37
M + NKL L +DVKGKRV+MR VDFNVP + +I+NN
Sbjct 224402 MKIGNKLGLKDIDVKGKRVLMRQVDNHLN*C*LMNIIHVLY*TCSVDFNVPFADGKISNN 224223
Query 38 QRIKAAVPSIKFCLDNGAKSVVLMSHLGRPDGVPMPDKYSLEPVAVELKSLLGKDVLFLK 97
QRI AA+PSI+ CL NGAK+VVLMSHLGRPDG + KYSL+PVA E++ LL + V FL+
Sbjct 224222 QRIVAAIPSIEHCLKNGAKAVVLMSHLGRPDG-KVVSKYSLKPVAAEVERLLKRKVTFLE 224046
Query 98 DCVGPEVEKACANPAAGSVILLENLRFHVEEEGKGKDASGNKVKAEPAKIEAFRASLSKL 157
DCVG +VE C N A G VILLENLRFH+EEEG KD SGNK KA +E FR SLS+L
Sbjct 224045 DCVGQKVEDQCKNAANGEVILLENLRFHIEEEGSVKDESGNKAKANKEDVEKFRQSLSQL 223866
Query 158 GDVYVNDAFGTAHRAHSSMVGVNLPQKAGGFLMKKELNYFAKALESPERPFLAILGGAKV 217
GDVYVNDAFGTAHRAHSS+VG+ L +A GFLMKKEL++F ALE+P+RPFLAI+GGAKV
Sbjct 223865 GDVYVNDAFGTAHRAHSSVVGIKLETRAAGFLMKKELDFFGMALENPQRPFLAIIGGAKV 223686
Query 218 ADKIQLINNMLDKVNEMIIGGGMAFTFLKVLNNMEIGTSLFDEEGAKIVKDLMSKAEKNG 277
+DKIQLI N+LDKVN MIIGGGMAFTF KV++ + IG SLFDEEG KIVK L+ KA+
Sbjct 223685 SDKIQLIENLLDKVNAMIIGGGMAFTFKKVVDGVSIGKSLFDEEGGKIVKKLVEKAKSKN 223506
Query 278 VKITLPVDFVTADKFDENAKTGQATVASGIPAGWMGLDCGPESSKKYAEAVTRAKQIVWN 337
V++ PVD+VTA KF ++A+ G A SGI MGLD G ES KK+ E V ++K I+WN
Sbjct 223505 VELVFPVDYVTASKFAKDAEVGYADDKSGIKDELMGLDVGSESVKKFKEVVLKSKTILWN 223326
Query 338 GPVGVFEWEAFARGTKALMDEVVKATSR-GCITIIGGGDTATCCAKWNTEDKVSHVSTGG 396
GP+GVFE+++F+ GT+ ++D V++AT++ G I+I+GGGDTAT +KWN +DKVSHVSTGG
Sbjct 223325 GPMGVFEFDSFSAGTRGVLDAVIEATAQNGAISIVGGGDTATAVSKWNADDKVSHVSTGG 223146
Query 397 GASLELLEGKVLPGV 411
G+S+EL EGK LPGV
Sbjct 223145 GSSVELAEGKELPGV 223101
> scaffold-17
Length=2125590
Score = 31.6 bits (70), Expect = 0.88, Method: Compositional matrix adjust.
Identities = 19/71 (27%), Positives = 31/71 (44%), Gaps = 5/71 (7%)
Frame = +1
Query 254 GTSLFDEEGAKIVKDLMSKAEKNGVKITLPVDFVTADKFDENAKTGQATVASGIPAGWMG 313
LF E IV++ + NG K ++ ++ ENA Q + S +GW+
Sbjct 2095696 SVDLFQEFMVVIVRECLLPKVGNGFKTVCA--YIDKAQYSENADDTQLSSES---SGWVK 2095860
Query 314 LDCGPESSKKY 324
DCG + +Y
Sbjct 2095861 FDCGSQQCIRY 2095893
4. Поиск гена белка в одном из контигов
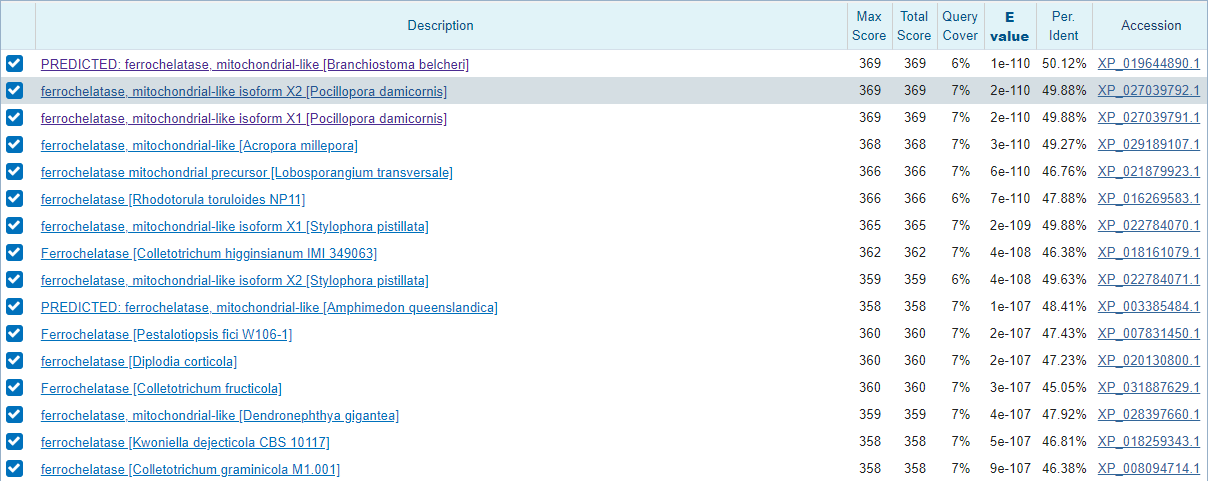
Для поиска был выбран скэффолд из геномной сборки Amoeboaphelidium protococcarum, состоящий из 17200 пар нуклеотидов, в котором ещё не аннотированы гены белков (scaffold-59). Поиск гомологов был выполнен на сайте NCBI BLAST (blastx: по refseq_protein, таксон - Opisthokonta (taxid:33154))
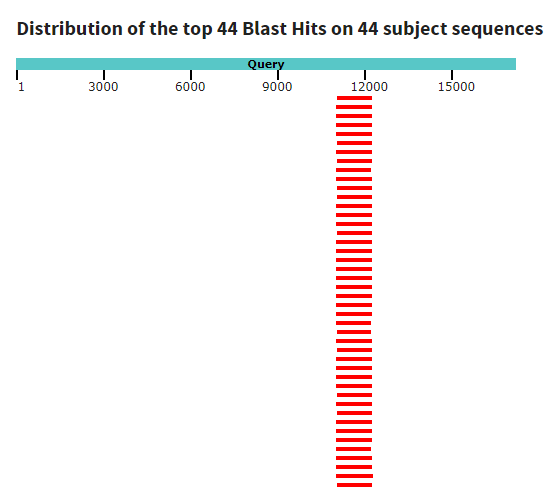
Результаты, предоставленные BLAST, показали наличие части гена белка ferrochelatase в данном контиге. Из рисунка 5 видно, что многие находки имеют близкие значения всех показателей, это говорит о том, что найденный участок очень консервативен. В этом также можно убедиться, посмотрев на график покрытия (Рис. 6) - все последовательности стоят под одним участком и имеют почти одинаковую длину.