Докинг низкомолекулярных лигандов в структуру белка
Докинг лиганда NAG в модельную структуру белка
В качестве структуры белка была использована структура №2 из предыдущего практикума. В качестве лиганда - NAG.
NAG: (файл .smi) → obgen → (файл .mol) → babel → (файл .pdb)
Далее с помощью скриптов prepare_ligand4.py и prepare_receptor4.py из пакета Autodock создал pdbqt файлы для лиганда и белка соответсвенно. Создал файл конфигурации vina.cfg, содержащий размеры и координаты цнтра параллелепипеда, внутри которого будет производиться докинг. Запустил первый докинг с использованием полученных файлов.
Выходные файлы: (файл .pdbqt) (файл .log)
Из лог-файла определил энергии трех лучших расположений лиганда: -5.6, -5.5 и -5.5 kCal/mol. Геометрическое расстояние второго и третьего расположений от первого: rmsd l.b. 2.408 и 2.247; rmsd u.b. 3.960 и 4.011 соответственно.
Расположения лиганда NAG в результате докинга
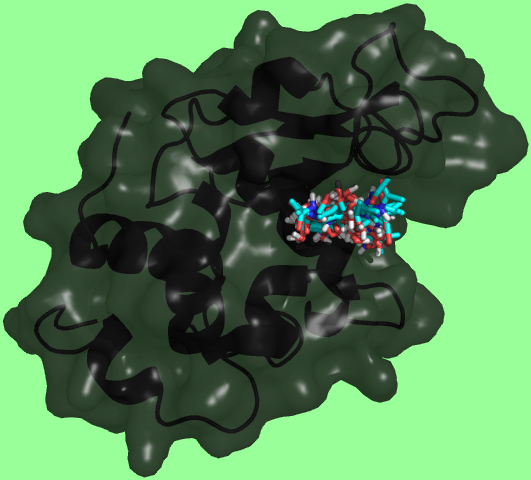
Ни одно положение лиганда после докинга не было достаточно близким к расположению соответствующей его части после моделирования.
Докинг лиганда NAG, рассматривая подвижность некоторых боковых радикалов белка
Провел докинг, рассматривая подвижность некоторых боковых радикалов белка. Разбил белок на две части, подвижную и неподвижную, с помощью скрипта prepare_flexreceptor4.py из пакета Autodock. Для подвижной части выбрал ASN46, ALA108 и VAL110 - 3 аминокислоты, которые были использованы в предыдущем практикуме для позиционирования лиганда. (файл .pdbqt для белка)
Выходные файлы: (файл .pdbqt) (файл .log)
Из лог-файла определил энергии трех лучших расположений лиганда: -5.4, -5.3 и -5.3 kCal/mol. Геометрическое расстояние второго и третьего расположений от первого: rmsd l.b. 2.084 и 2.084; rmsd u.b. 3.431 и 3.565 соответственно. Докинг с подвижными радикалами длился дольше, а результаты хуже, судя по аффинности.
Расположения лиганда NAG в результате докинга

Ни одно положение лиганда после докинга, рассматривая подвижность некоторых боковых радикалов белка, не было достаточно близким к расположению соответствующей части лиганда после моделирования.
Докинг лигандов с замененной группой СH3-
Провел обычный докинг для лиганда NAG, в котором в СH3С(=O)NH- группе СH3- был заменен на H-, OH- и NH2-.
NAG-H:&tab;(файл .smi) → obgen → (файл .mol) → babel → (файл .pdb) → prepare_ligand4.py → (файл .pdbqt) →→ vina → (файл .pdbqt) (файл .log)
NAG-OH: (файл .smi) → obgen → (файл .mol) → babel → (файл .pdb) → prepare_ligand4.py → (файл .pdbqt) →→ vina → (файл .pdbqt) (файл .log)
NAG-NH2: (файл .smi) → obgen → (файл .mol) → babel → (файл .pdb) → prepare_ligand4.py → (файл .pdbqt) →→ vina → (файл .pdbqt) (файл .log)
Аффинность трех лучших расположений
|
Лиганд
|
Первое расположение
|
Второе расположение
|
Третье расположение
|
|
NAG-H
|
-5.3 kCal/mol
|
-5.2 kCal/mol
|
-4.9 kCal/mol
|
|
NAG-OH
|
-5.8 kCal/mol
|
-5.5 kCal/mol
|
-5.5 kCal/mol
|
|
NAG-NH2
|
-5.9 kCal/mol
|
-5.8 kCal/mol
|
-5.7 kCal/mol
|
Расположения лиганда NAG-H в результате докинга
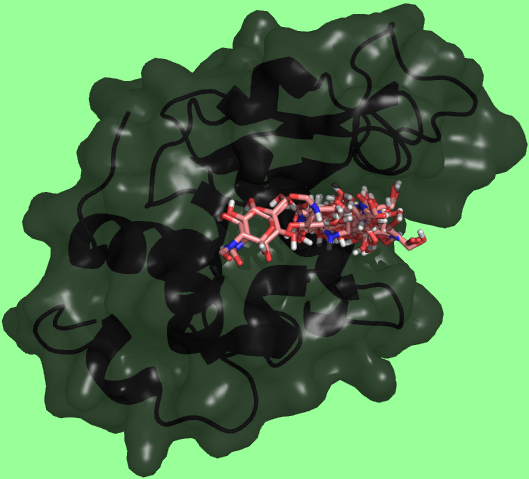
Расположения лиганда NAG-OH в результате докинга
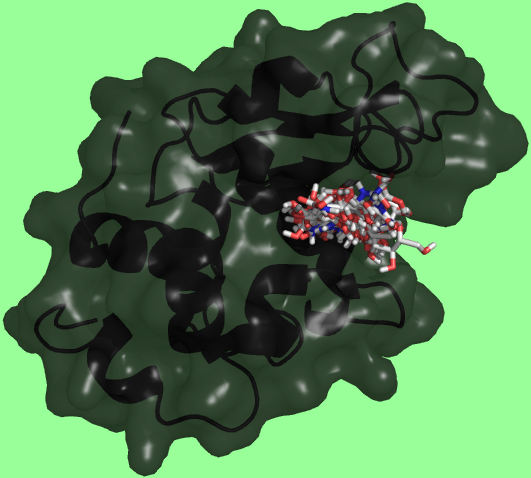
Расположения лиганда NAG-NH2 в результате докинга

Наибольшая аффинность наблюдается при замене CH3- группы на NH2- группу, также для этого лиганда наблюдается наименьший разброс в расположениях после докинга. Вероятно, такая замена может увеличить энергию связывания лиганда в реальности, а может быть и нет.

2011
©






