SCOP и CATH
SCOP
С помощью SCOP была получена информация о доменах структур 2PUB и 1J2R.
1J2R содержит четыре одинаковых домена, занимающих четыре цепи A, B, C, D. Название домена: "Hypothetical protein YecD from Escherichia coli"
- Класс: Alpha and beta proteins (a/b)
- Укладка: Isochorismatase-like hydrolases
- Суперсемейство: Isochorismatase-like hydrolases
- Семейство: Isochorismatase-like hydrolases
Есть 1 суперсемейство с укладкой "Isochorismatase-like hydrolases" и суперсемейство "Isochorismatase-like hydrolases" содержит только одно семейство.
2PUB включает два домена.
Домен "Purine repressor (PurR), N-terminal domain from Escherichia coli":
- Положение: цепь А, остатки 3-58
- Класс: All alpha proteins
- Укладка: lambda repressor-like DNA-binding domains (1)
- Суперсемейство: lambda repressor-like DNA-binding domains (13)
- Семейство: GalR/LacI-like bacterial regulator
Есть 1 суперсемейство с укладкой "lambda repressor-like DNA-binding domains" и суперсемейство "lambda repressor-like DNA-binding domains" содержит 13 семейств.
Домен "Purine repressor (PurR), C-terminal domain from Escherichia coli":
- Положение: цепь А, остатки 59-340
- Класс: Alpha and beta proteins (a/b)
- Укладка: Periplasmic binding protein-like I
- Суперсемейство: Periplasmic binding protein-like I
- Семейство: L-arabinose binding protein-like
Есть 1 суперсемейство с укладкой "Periplasmic binding protein-like I" и суперсемейство "Periplasmic binding protein-like I" содержит 1 семейство.
CATH
С помощью CATH была получена информация о доменах структур 2PUB и 1J2R.
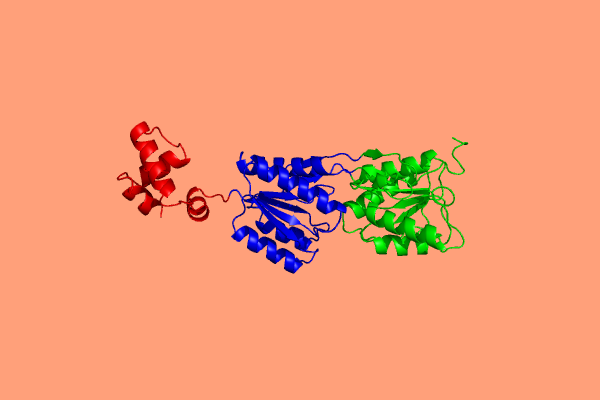
1J2R содержит четыре одинаковых домена, занимающих четыре цепи A, B, C, D. Название доменов: 1j2rA00 (3.40.50.850.2.1.1.1.1), 1j2rB00 (3.40.50.850.2.1.1.1.4), 1j2rC00 (3.40.50.850.2.1.1.1.3), 1j2rD00 (3.40.50.850.2.1.1.1.2).
- Положение: остатки 12-199 на соответствующей цепи
- Класс: Alpha Beta
- Архитектура: 3-Layer(aba) Sandwich
- Топология: Rossmann fold (содержит 120 суперсемейств)
- Суперсемейство: нет названия (включает 6 семейств)
- Семейство: нет названия
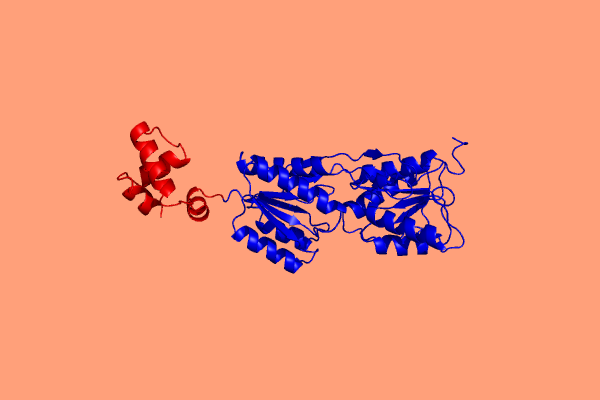
Структура 2PUB включает три домена. Домен 2pubA01:
- Положение: цепь A, остатки 3-59
- Классификация по CATH: 1.10.260.40.18.1.1.1.9
- Класс: Mainly Alpha
- Архитектура: Orthogonal Bundle
- Топология: Repressor (Amino-terminal Domain) (содержит 2 суперсемейства)
- Суперсемейство: lambda repressor-like DNA-binding domains (включает 23 семейства)
- Семейство: нет названия
Домен 2pubA02:
- Положение: цепь A, остатки 60-161, 292-323
- Классификация по CATH: 3.40.50.2300.49.1.1.1.12
- Класс: Alpha Beta
- Архитектура: 3-Layer(aba) Sandwich
- Топология: Rossmann fold (содержит 120 суперсемейства)
- Суперсемейство: нет названия (включает 103 семейства)
- Семейство: нет названия
Домен 2pubA03:
- Положение: цепь A, остатки 162-291, 324-340
- Классификация по CATH: 3.40.50.2300.39.2.1.2.3
- Класс: Alpha Beta
- Архитектура: 3-Layer(aba) Sandwich
- Топология: Rossmann fold (содержит 120 суперсемейства)
- Суперсемейство: нет названия (включает 103 семейства)
- Семейство: нет названия
Сравнение SCOP и CATH
Домены в структуре 1J2R по SCOP и CATH полностью совпадают, потому что он один на каждой цепи и занимает её полностью. Они относятся к одинаковым классам в SCOP и CATH, укладка по SCOP не соответствует топологии по CATH, так как в SCOP есть только одно суперсемейство "Isochorismatase-like hydrolases" с одним семейством "Isochorismatase-like hydrolases", тогда как топология по CATH включает 120 суперсемейств и 1283 семейств в общей сложности. В структуре 2PUB первый домен по положению совпадает с точностью до одной аминокислоты, его классификация по SCOP и CATH очень похожа. Второй домен 2PUB по SCOP разбит на два очень похожих в CATH.
Домены 2PUB по SCOP
Домены 2PUB по CATH
Выравнивание двух двух доменов с одинаковой топологией по CATH, но из разных суперсемейств
Были выбраны две структуры с укладкой "Ribonuclease HI; Chain A", из суперсмейств "Ribonuclease Hi; Chain A" (3bsuA00, PDB:3BSU) и "Carbon monoxide dehydrogenase alpha subunit. Chain D, domain 4" (1oaoC04, PDB: 1OAO). Далее было получено пространственное выравнивание с помощью PDBeFOLD (RMSD=2.51, файл в fasta формате), а потом найдено геометрическое ядро по Geometrical core. Две структуры были выровнены командой pair_fit в PYMOL по CA атомам геометрического ядра (RMSD=1.489). (сессия PyMOL)
Пространственное совмещение по геометрическому ядру доменов
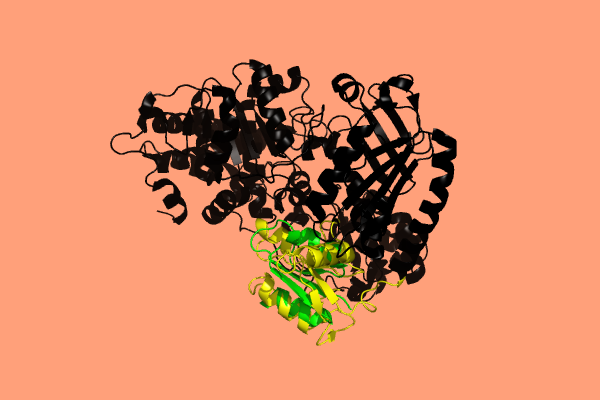
(На изображении 3BSU (цепь А, выбранный домен)- зеленая, 1OAO - черная, выбранный домен желтый)
Если учитывать, что домены достаточно сильно отличаются, то выравнивание можно назвать хорошим.

2010
©





