На главную На страницу третьего семестра
— — — — — — — —
Задание №1
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted** | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 5'-501-507-3' 5'-567-573-3' Всего 7 пар | 5'-01-07-3' 5'-67-73-3' Всего 7 пар | 5'-01-07-3' 5'-67-73-3' Всего 7 пар |
| D-стебель | 5'-510-513-3' 5'-523-526-3' Всего 4 пары | Всего 0 пар | 5'-10-13-3' 5'-23-26-3' Всего 4 пары |
| T-стебель | 5'-550-554-3' 5'-562-566-3' Всего 5 пар | Всего 0 пар | 5'-50-54-3' 5'-62-66-3' Всего 5 пар |
| Антикодоновый стебель | 5'-528-532-3' 5'-540-544-3' Всего 5 пар | 5'-31-33-3' 5'-47-49-3' Всего 3 пары | 5'-28-32-3' 5'-40-44-3' Всего 5 пар |
| Общее число канонических пар нуклеотидов | 20 (552-564 неканоничная) | 10 | 21 |
**Для программы einverted были использованы две конфигурации: 1 – einverted 1J1U_tRNA.fasta -gap 12 -threshold 0 -match 3 -mismatch -4 -outfile 1J1U_tRNA.inv -outseq 1J1U_tRNA_invregions.fasta – использовалась для лучшего поиска пар в акцепторном стебле, так как в этом случае найденные пары были ближе к реальности, однако в такой конфигурации он не находил никакие другие пары в других стеблях; для их поиска использовалась конфигурация 2 – einverted 1J1U_tRNA.fasta -gap 0 -threshold 0 -match 3 -mismatch -4 -outfile 1J1U_tRNA.inv -outseq 1J1U_tRNA_invregions.fasta – результат которой был менее схож с реальными данными, однако тут были хоть какие-то результаты для антикодонового стебля.
Скачать файл с данными по find_pair
Скачать файл с данными по einverted с наиболее близкими к реальности результатами
Скачать файл с данными по einverted с результатами в антикодоновом стебле
Скачать файл с данными по алгоритму Зукера
Задание №2
Упражнение №1Скрипт, задающий множества set1, set2 и set3
Скрипт, представляющий структуру
Упражнение №2
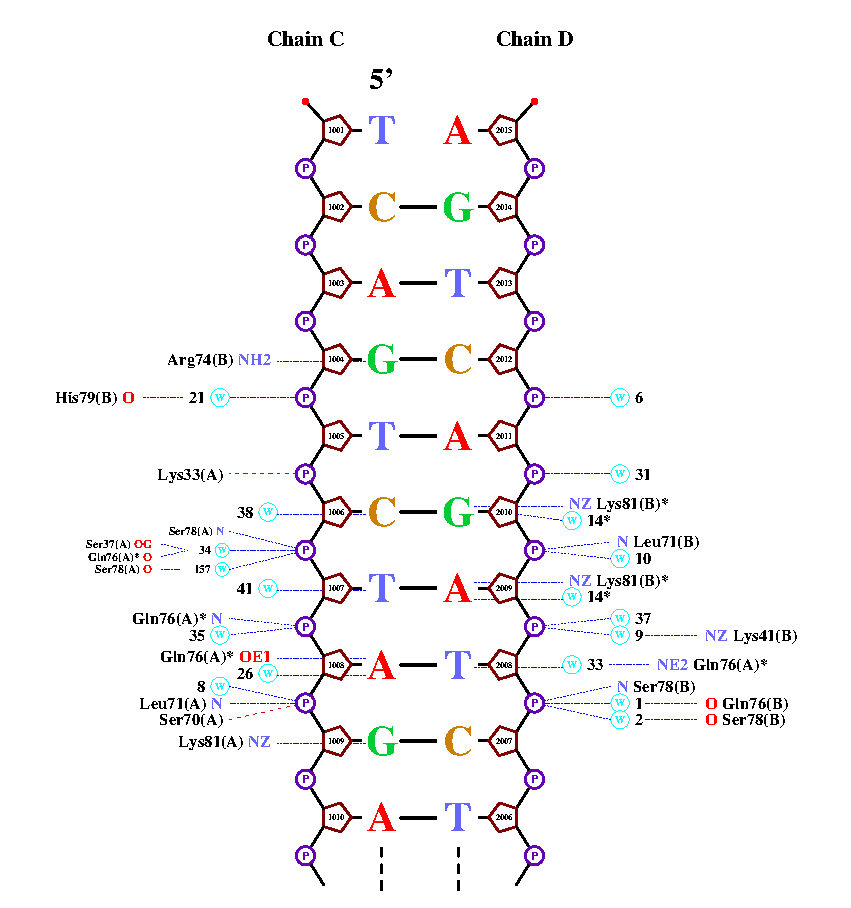
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 0 | 9 | 9 |
| остатками фосфорной кислоты | 9 | 10 | 19 |
| остатками азотистых оснований со стороны большой бороздки | 9 | 10 | 19 |
| остатками азотистых оснований со стороны малой бороздки | 8 | 17 | 25 |
Как мы можем видеть из таблицы, для любой выбранной части молекулы количество неполярных контактов больше, чем количество полярных. Я думаю, причина в первую очередь в том, что для полярных и неполярных контактов было взято разное расстояние в качестве критерия взаимодействия (3,5 Å и 4,5 Å соответственно).
Для поиска контактов использовались команды:
- Полярные, остатки 2'-дезоксирибозы: select (protein and (_N or _O)) and within(3.5, (*.O4' or *.O3' or *.O5') and DNA)
- Неполярные, остатки 2'-дезоксирибозы: select (protein and (_C or _P or _S)) and within(4.5, (*.C4' or *.C3' or *.C5' or *.C1' or *.C2') and DNA)
- Полярные, остатки фосфорной кислоты: select (protein and (_N or _O)) and within(3.5, (*.OP1 or *.OP2) and DNA)
- Неполярные, остатки фосфорной кислоты: select (protein and (_C or _P or _S)) and within(4.5, *.P and DNA)
- Полярные, сторона большой бороздки: select (protein and (_N or _O)) and within(3.5, (*.OP1 or *.OP2 or *.O5' or *.O3') and DNA)
- Неполярные, сторона большой бороздки: select (protein and (_C or _P or _S)) and within(4.5, *.P and DNA)
- Полярные, сторона малой бороздки: select (protein and (_N or _O)) and within(3.5, (not (*.OP1 or *.OP2 or *.O3' or *.O5')) and (_O or _N) and DNA)
- Неполярные, сторона малой бороздки: select (protein and (_C or _P or _S)) and within(4.5, _C and DNA)
Упражнение №3
Упражнение №4
Аминокислотный остаток с наибольшим числом контактов - 76 (GLN). Наиболее важный для распознавания структуры - 81 (LYS), так как он близко к центру ДНК, а центр может играть очень существенную роль в функционировании молекулы (в центре может располагаться активный каталитический центр, например), а также он взаимодействует с несколькими нуклеотидами, что означает, что он играет важную роль в молекуле.
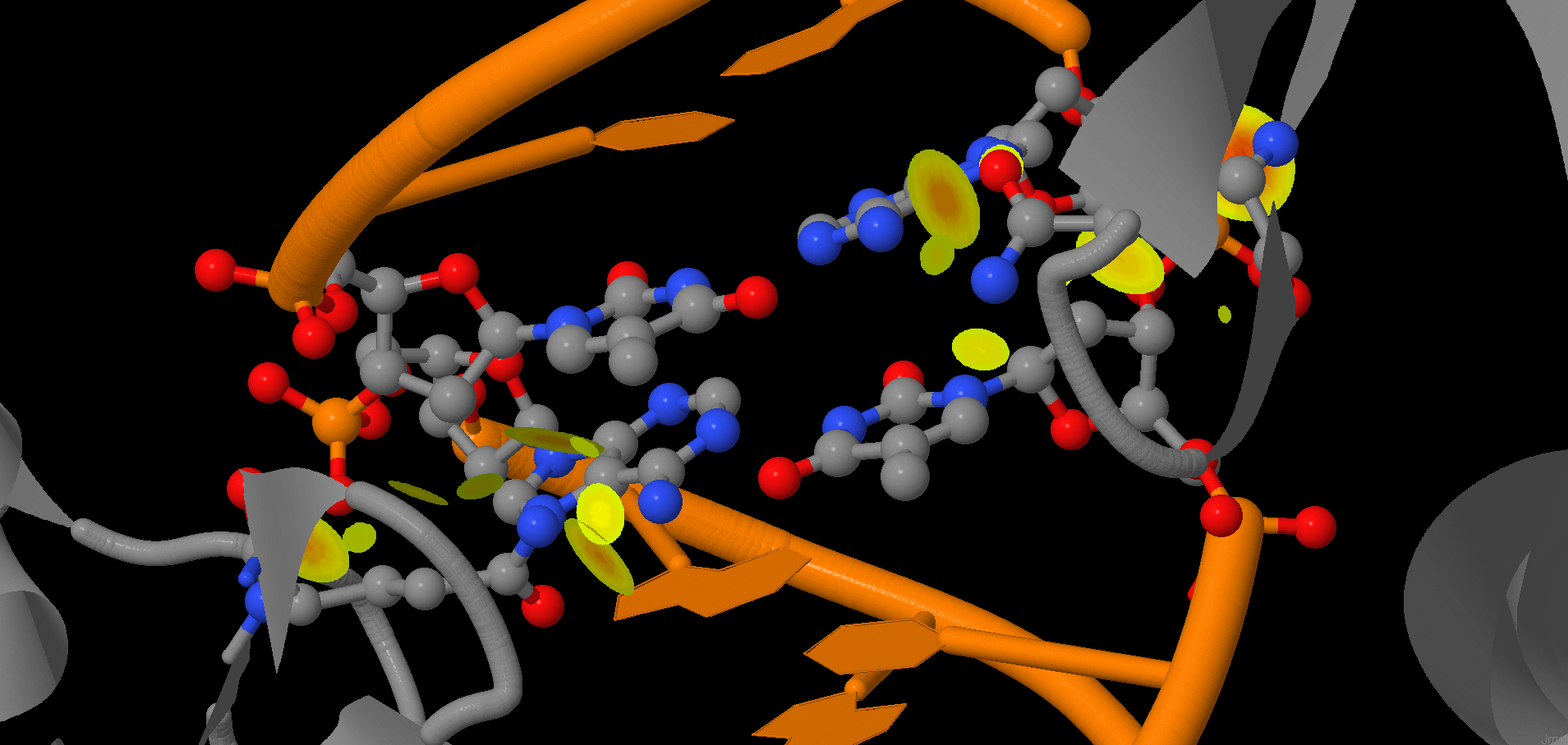
Картинка 3. Контакты между 76 (GLN) остатками (по одному в каждой цепи белка) и ДНК.
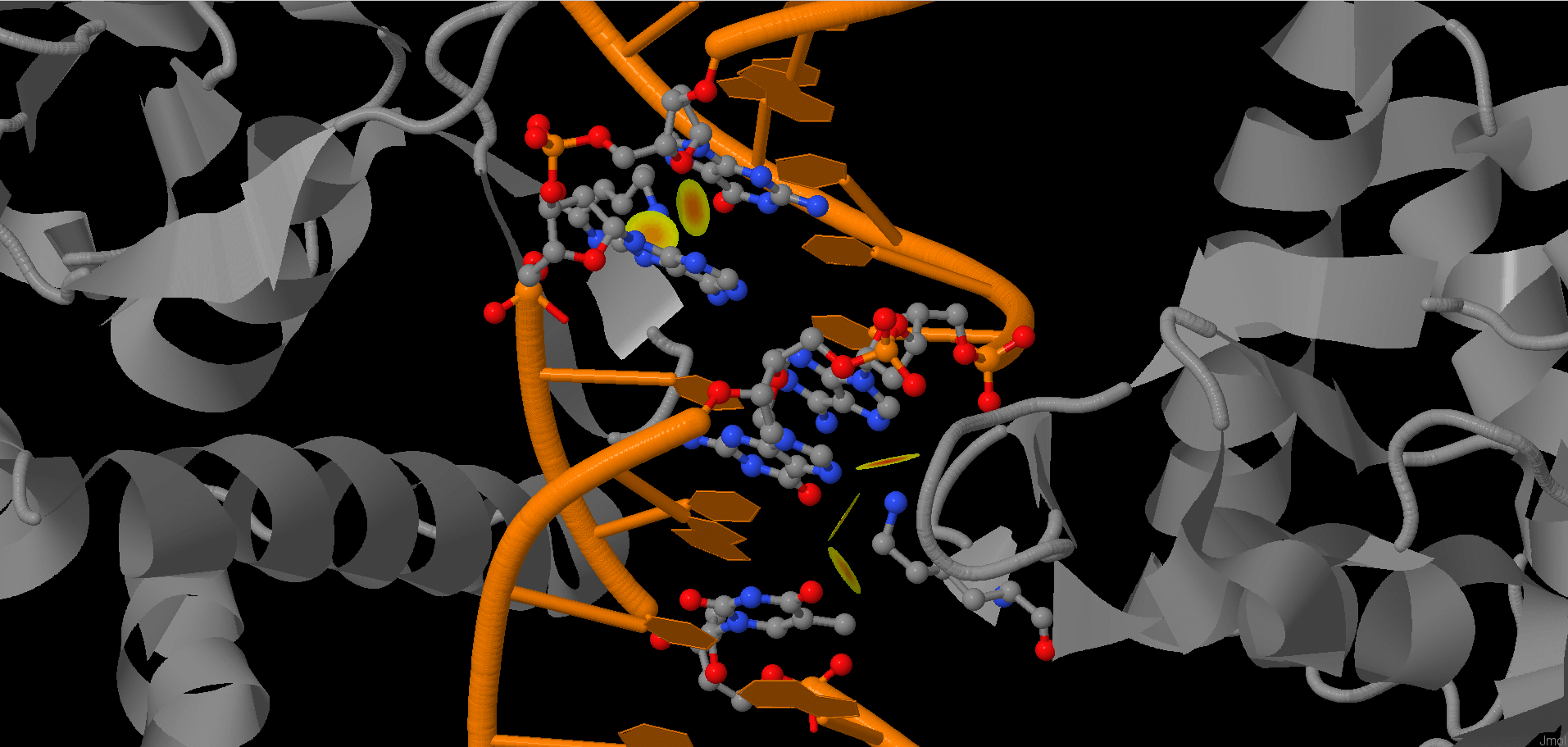
Картинка 4. Контакты между 81 (LYS) остатками (по одному в каждой цепи белка) и ДНК.