На главную На страницу третьего семестра
— — — — — — — —
Задание №1
Скачать итоговую последовательность из 6-ого практикума
Для поиска я использовал алгоритм blastn с параметрами по умолчанию, так как про данную нам последовательность мы вообще ничего не знаем. Полученный
результат:
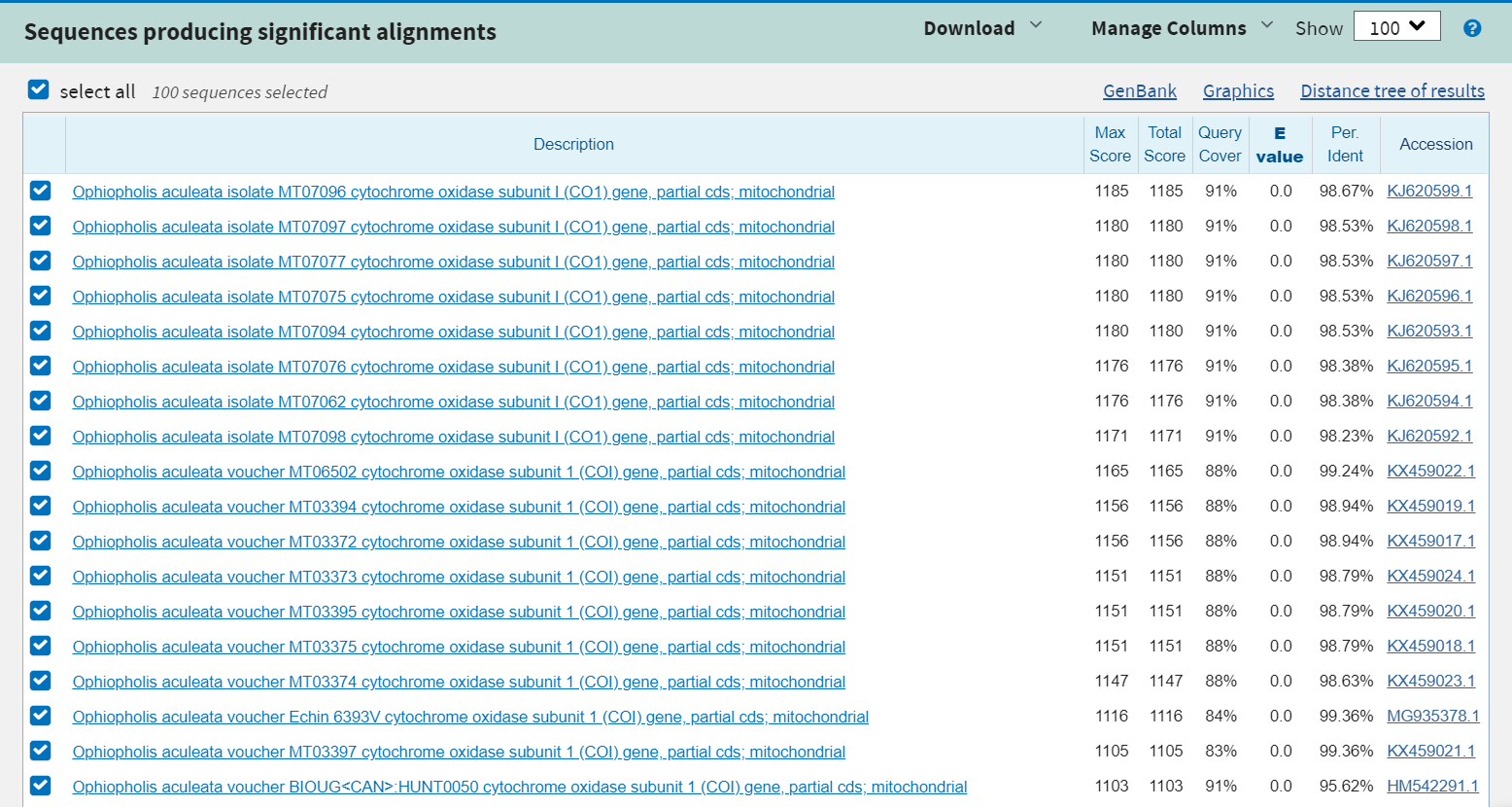
Для определения таксономии можно воспользоваться вкладкой "taxonomy", где находки распределены по разным таксономическим уровням. Бесспорным лидером по
весу выравниваний и количеству совпадений является вид Ophiopholis aculeata (cellular organisms; Eukaryota; Opisthokonta; Metazoa; Eumetazoa;
Bilateria; Deuterostomia; Echinodermata; Eleutherozoa; Asterozoa; Ophiuroidea; Ophiuridea; Ophiurida; Ophiurina; Gnathophiurina; Ophiactidae; Ophiopholis).
Ниже приведены результаты. Примечательно, что в выдачу попало одно хорошее совпадение с некой бабочкой, что резко выбивается из остальных результатов. Я
проверил этот результат, запустив поиск ещё несколько раз с более подробными критериями поиска, однако этот результат так и остался единственным, так что я
думаю, что это просто ошибка в базе данных.
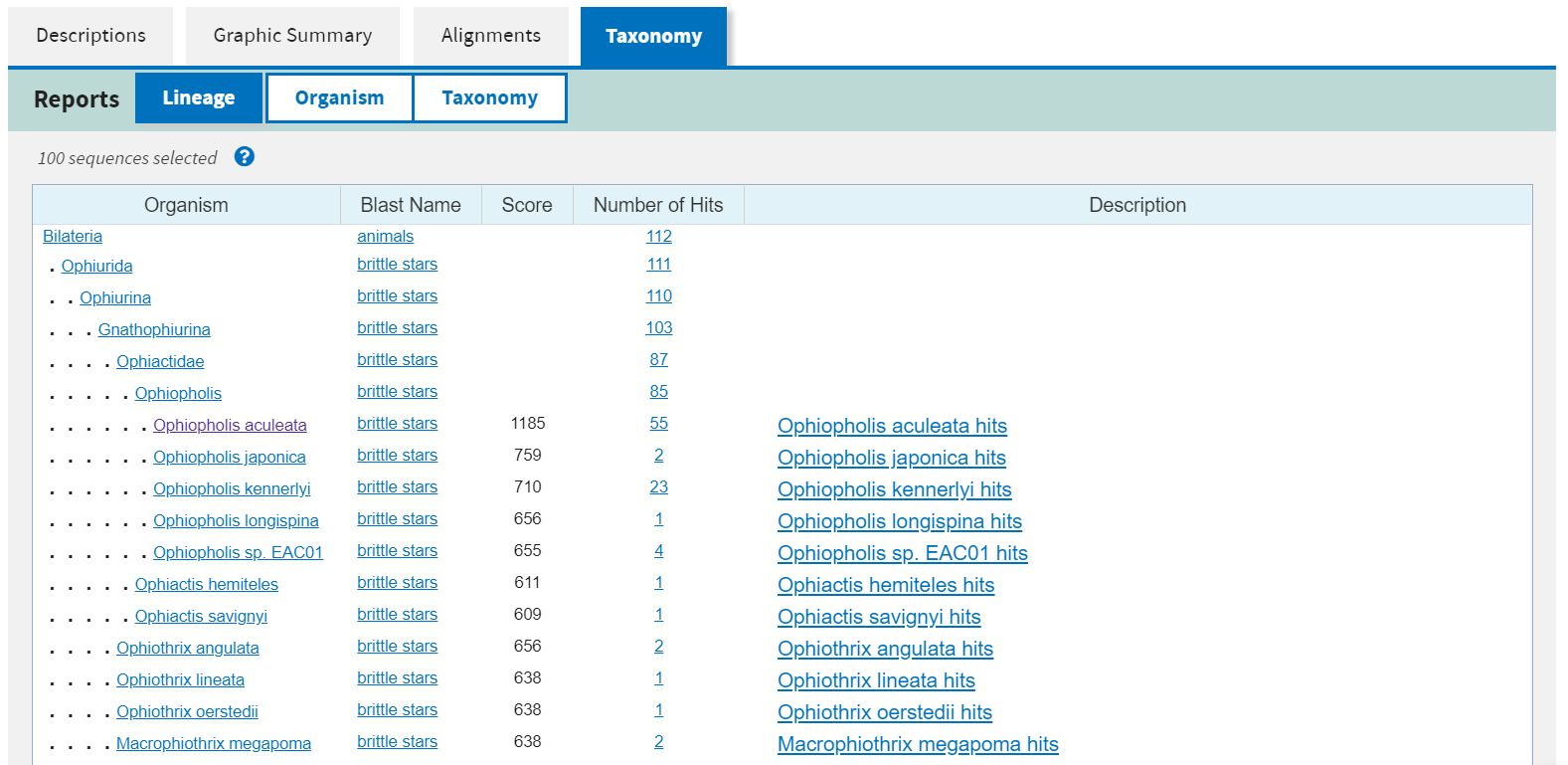
С Ophiopholis aculeata моя последовательность практически идеально сходится (просто визуально, но кроме того выравнивания с данным видом имеют
крайне низкие значения E-value и большие веса и перекрытия), за исключением неизвестных нуклеотидов, что говорит в пользу того, что можно определять
таксономию с точностью до вида.

Данный ген кодирует белок cytochrome oxidase subunit I, partial (mitochondrion) [Ophiopholis aculeata]. Ссылки на описание гена, описание белка.
Задание №2
Поиск по последовательности из 6-ого практикума
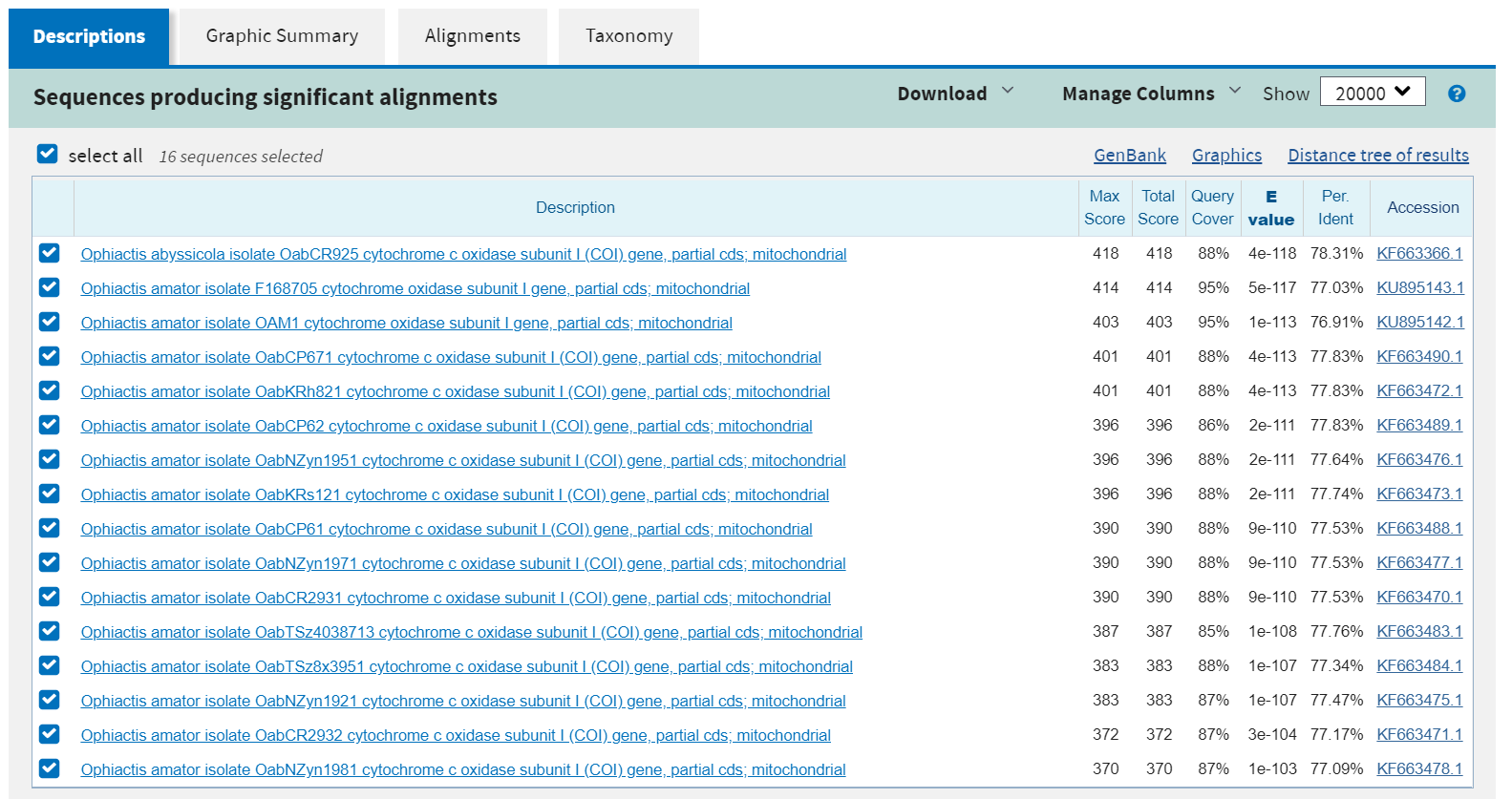
Поиск с использованием алгоритма megablast. На таксоны было наложено ограничение – исключение рода и ограничение по семейству (Ophiactidae and exclude
Ophiopholis). Остальные параметры по умолчанию, кроме максимального количества находок – 20000.
Поиск по алгоритму blastn. Аналогичное ограничение на таксоны. И так же, параметры по умолчанию, но максимальное количество находок – 20000.
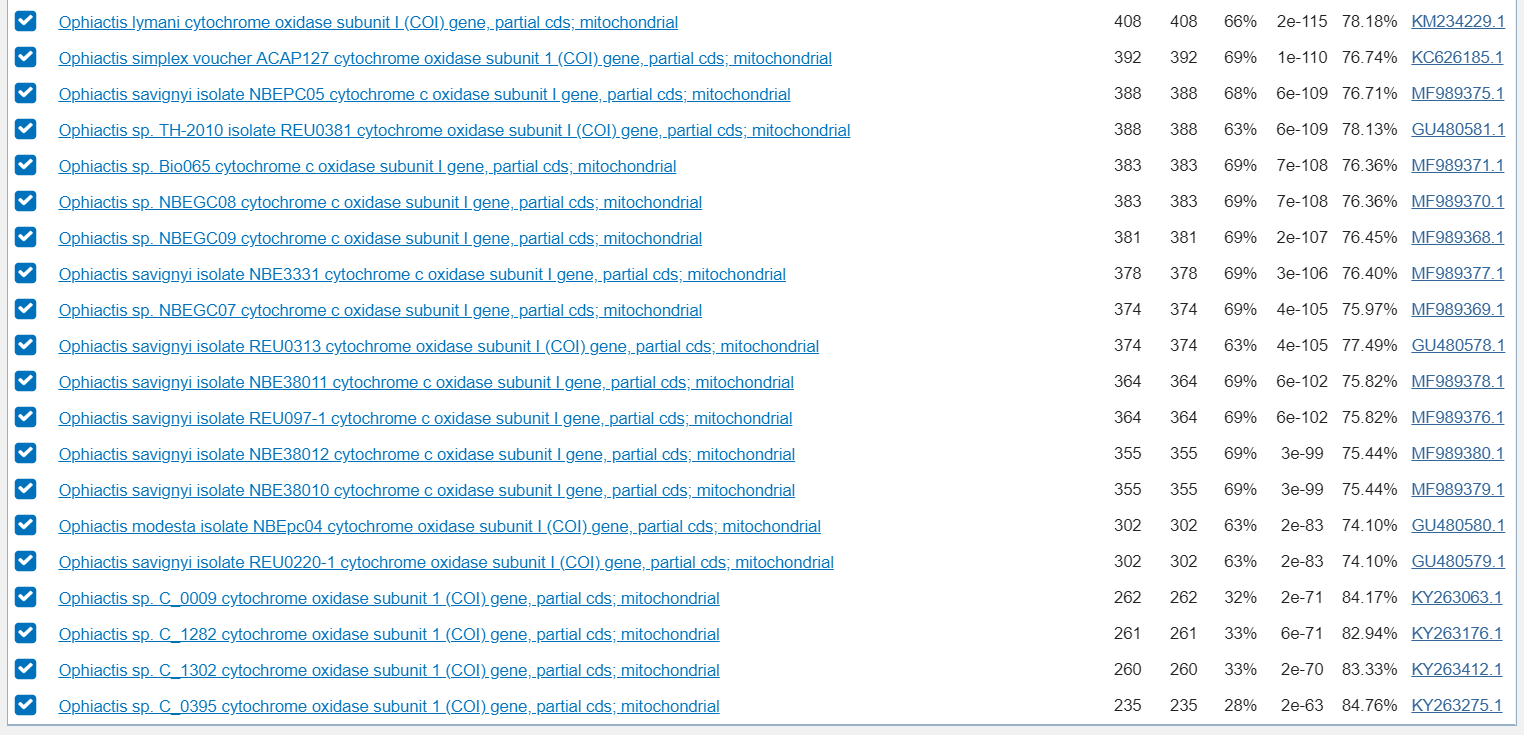
Поиск по алгоритму blastn с наиболее чувствительными параметрами. Аналогичное ограничение на таксоны. Все параметры по умолчанию, кроме максимального
количества находок (20000), word size (7) и Match/Mismatch Scores (1,-1), что является самой чувствительной комбинацией.

| Алгоритм | Число находок |
|---|---|
| Megablast | 16 |
| Blastn с параметрами по умолчанию | 288 |
| Blastn с наиболее чувствительными параметрами | 288 |
Исходя из результатов сравнения, можно сказать, что в основном алгоритмы отличаются тем, насколько близкие последовательности они ищут. Megablast ищет только очень схожие последовательности (поэтому результатов всего 16), в то время как blastn работает дольше и находит менее схожие варианты. Однако в данном случае для нахождения всех вариантов из заданного ограничения по таксономии оказалось достаточно настроек чувствительности по умолчанию, поэтому более чувствительный вариант blastn просто повторил результат предыдущего алгоритма с базовыми настройками.
Поиск по последовательности вируса
Везде было задано ограничение по таксономии – unclassified Siphoviridae; и максимальное количество результатов – 20000. Результаты поиска
megablast:
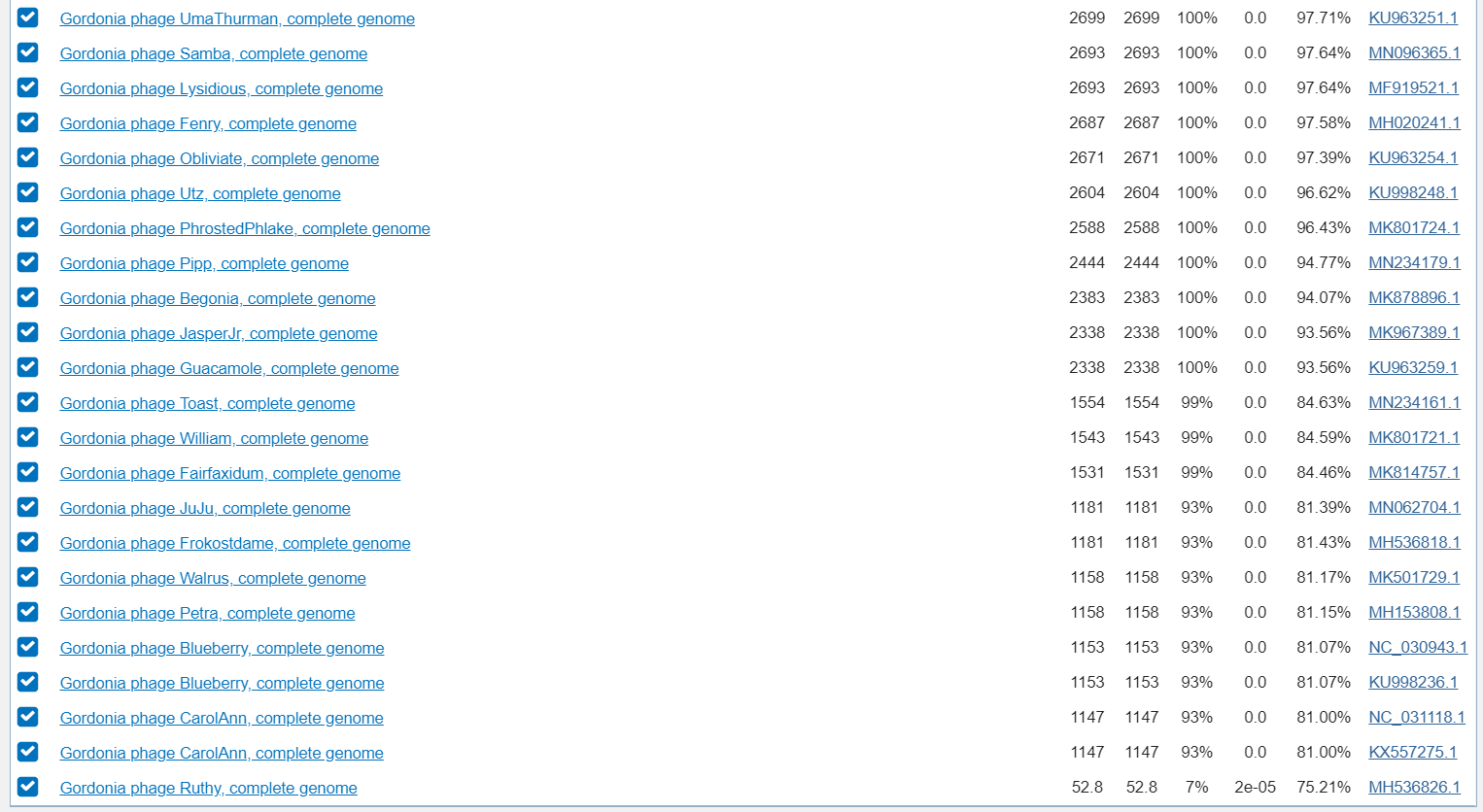
Результаты поиска blastn:
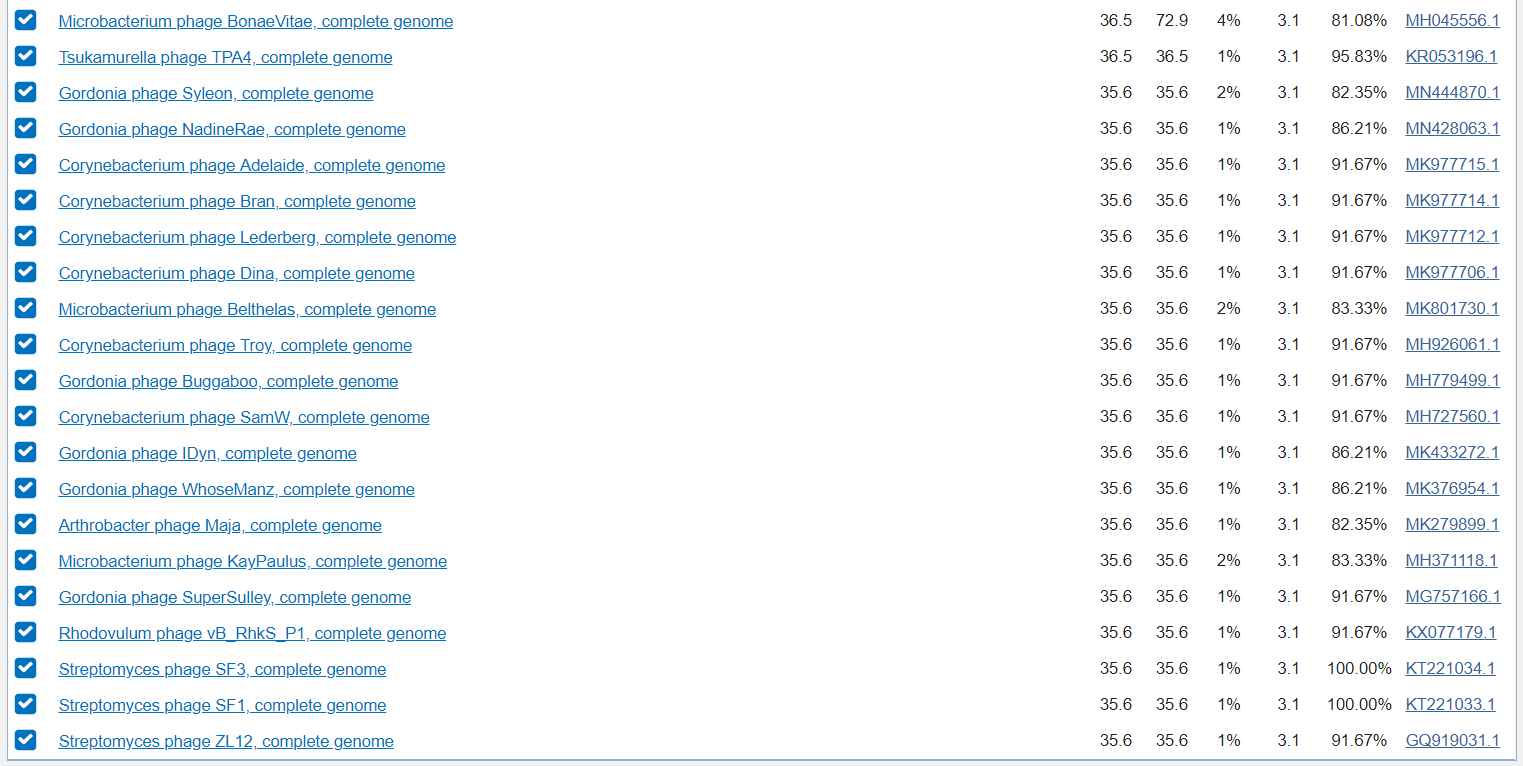
Параметры были заданы такие же, как и поиске по наиболее чувствительному blastn в предыдущей части задания. Результаты поиска blastn с наиболее
чувствительными параметрами:

| Алгоритм | Число находок |
|---|---|
| Megablast | 30 |
| Blastn с параметрами по умолчанию | 87 |
| Blastn с наиболее чувствительными параметрами | 194 |
Ожидаемо, чем чувствительнее алгоритм, тем больше результатов от более дальних в эволюционном плане организмов получает программа. Так что применение того или иного алгоритма зависит от того, что требуется найти.
Задание №3
Для данного задания были выбраны белки: P08898.4, P35579.4, NP_001287541.1. Последовательности белков были получены с их страниц в NCBI.
Для поиска гомологов использовались команды: "makeblastdb -in X5.fasta -dbtype nucl" и "tblastn -query histone.fasta -db X5.fasta -out histone.out".
| Белок | Результат | Вывод |
|---|---|---|
| Histone H3 | Скачать | Лучшие результаты – Score = 239 bits (610), Expect = e-72, Identities = 123/136 (90%), Positives = 126/136 (93%) – такой высокий уровень сходства (последовательности совпадают почти полностью) говорит о гомологии этих белков. |
| Aldolase | Скачать | Лучшие результаты – Score = 332 bits (850), Expect = 4e-101, Identities = 205/384 (53%), Positives = 248/384 (65%) – такой высокий уровень сходства (вкупе с некоторыми участками, которые вообще не претерпели изменений) говорит о том, что это гомологичные белки. |
| Myosin-9 | Скачать | Лучшие результаты – Score = 912 bits (2358), Expect = 0.0, Identities = 471/835 (56%), Positives = 581/835 (70%) – такой высокий уровень сходства отдельных участков последовательности, а также тот факт, что в выравнивания попала далеко не вся последовательность, говорит о частичной гомологии белков. Скорее всего, в данном случае гомологичны отдельные домены. |
Задание №4
В качестве последовательности был взят scaffold-700 из генома
Amoeboaphelidium protococcarum длинной 38453 bp. Далее, используя алгоритм blastx был получен результат:
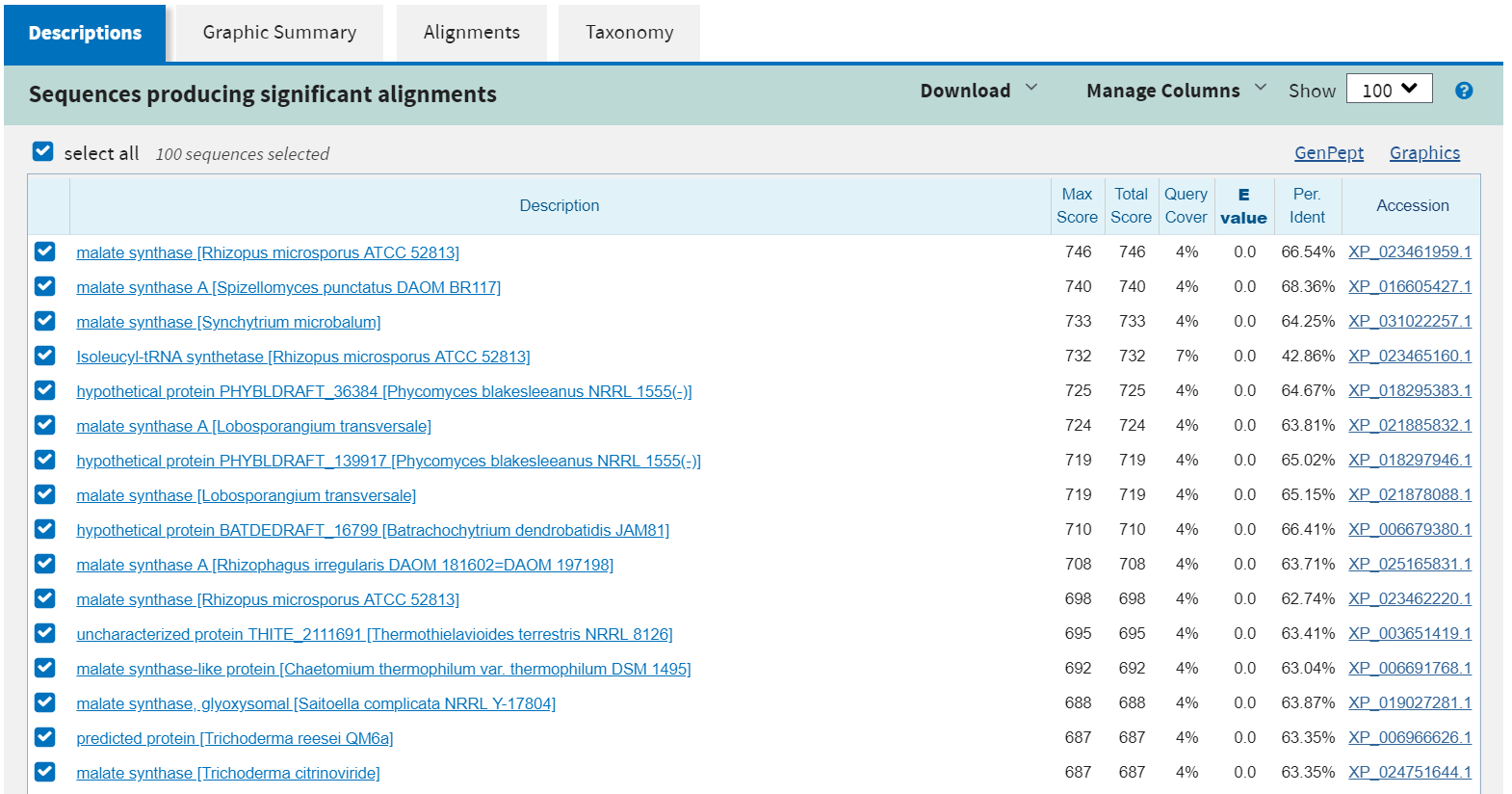
Данный скэффолд содержит множество генов, например, ген малат синтазы.