Для поиска гомологов белка из E.coli CLPX была создана база из протеомов отобранных бактерий и проведен blastp белка по этой базе. Результаты blastp:
Score E Sequences producing significant alignments: (Bits) Value sp|Q2RL30|CLPX_MOOTA ATP-dependent Clp protease ATP-binding sub... 533 0.0 sp|A5I6W0|CLPX_CLOBH ATP-dependent Clp protease ATP-binding sub... 530 0.0 sp|Q81LB9|CLPX_BACAN ATP-dependent Clp protease ATP-binding sub... 513 2e-180 sp|Q5HNM9|CLPX_STAEQ ATP-dependent Clp protease ATP-binding sub... 499 4e-175 sp|Q833M7|CLPX_ENTFA ATP-dependent Clp protease ATP-binding sub... 498 2e-174 sp|P63791|CLPX_STRPN ATP-dependent Clp protease ATP-binding sub... 486 4e-170 tr|Q5FKR6|Q5FKR6_LACAC ATP-dependent Clp protease ATP-binding s... 484 3e-169 sp|Q834K4|HSLU_ENTFA ATP-dependent protease ATPase subunit HslU... 103 3e-24 sp|Q5FKD8|HSLU_LACAC ATP-dependent protease ATPase subunit HslU... 101 1e-23 tr|Q2RJP5|Q2RJP5_MOOTA ATP-dependent protease ATPase subunit Hs... 100 4e-23 sp|Q81WK6|HSLU_BACAN ATP-dependent protease ATPase subunit HslU... 98.2 2e-22 sp|Q5HPT8|HSLU_STAEQ ATP-dependent protease ATPase subunit HslU... 93.6 8e-21 tr|A5I766|A5I766_CLOBH ATP-dependent zinc metalloprotease FtsH ... 52.0 2e-07 tr|Q2RLR4|Q2RLR4_MOOTA AAA domain-containing protein OS=Moorell... 50.1 6e-07 tr|A0A0H2USJ7|A0A0H2USJ7_STRPN ATP-dependent Clp protease, ATP-... 50.4 6e-07 tr|Q5FHW6|Q5FHW6_LACAC UVR domain-containing protein OS=Lactoba... 48.9 2e-06 tr|Q2RLP6|Q2RLP6_MOOTA AAA domain-containing protein OS=Moorell... 48.5 2e-06 tr|A5I7Q0|A5I7Q0_CLOBH ATP-dependent zinc metalloprotease FtsH ... 46.2 1e-05 sp|O69076|FTSH_STRPN ATP-dependent zinc metalloprotease FtsH OS... 46.2 1e-05 tr|A0A347ZXP1|A0A347ZXP1_BACAN ATP-dependent zinc metalloprotea... 43.1 1e-04 tr|Q2RM95|Q2RM95_MOOTA ATP-dependent zinc metalloprotease FtsH ... 43.1 1e-04 tr|A0A1Q4LW06|A0A1Q4LW06_BACAN AAA domain-containing protein OS... 42.4 2e-04 tr|Q5FMA3|Q5FMA3_LACAC ATP-dependent zinc metalloprotease FtsH ... 42.4 2e-04 sp|Q5HPD3|Y979_STAEQ Uncharacterized protein SERP0979 OS=Staphy... 41.6 3e-04 tr|A5I501|A5I501_CLOBH AAA domain-containing protein OS=Clostri... 41.6 3e-04 tr|Q839B1|Q839B1_ENTFA ATP-dependent zinc metalloprotease FtsH ... 41.6 4e-04 tr|A5HYU4|A5HYU4_CLOBH ATP-dependent zinc metalloprotease FtsH ... 41.2 4e-04 tr|Q5HRP3|Q5HRP3_STAEQ ATP-dependent zinc metalloprotease FtsH ... 41.2 6e-04 tr|Q2RJJ8|Q2RJJ8_MOOTA AAA domain-containing protein OS=Moorell... 40.4 9e-04
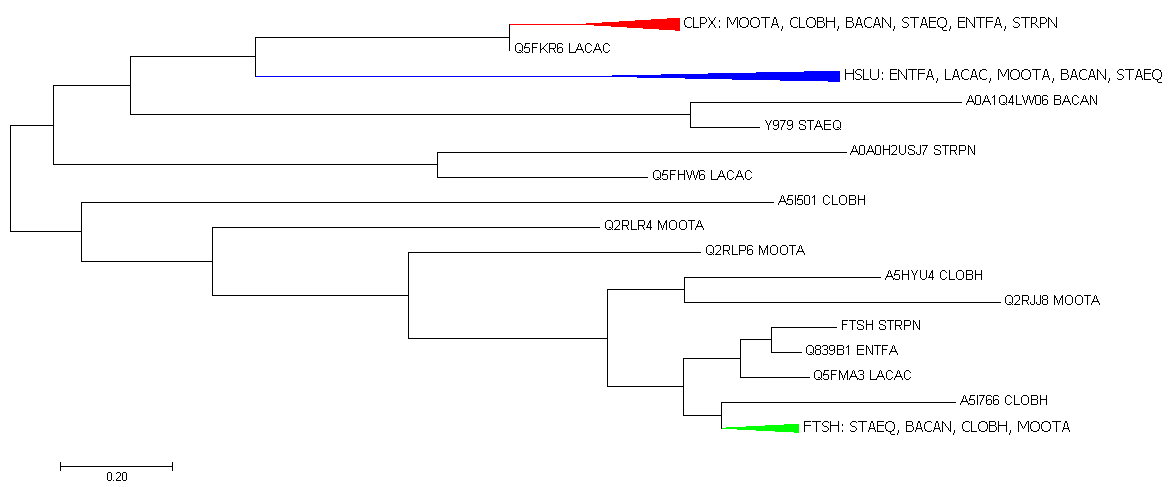
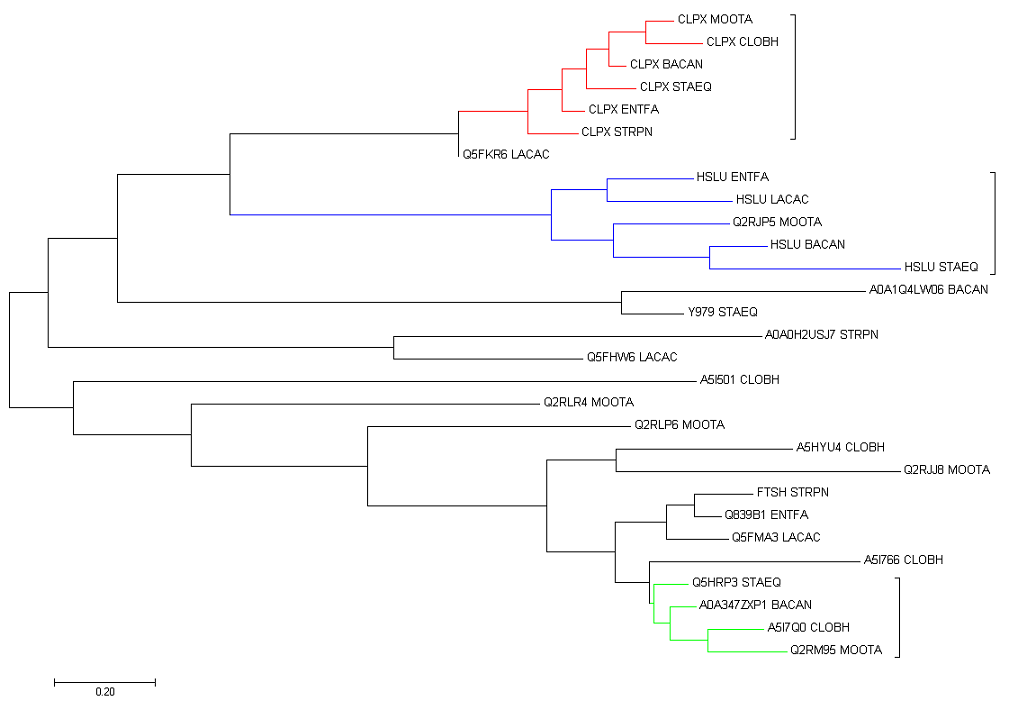
В результате работы с выравниванием было построено дерево методом максимального правдоподобия. Скобочную формулу в формате .nwk можно посмотреть здесь. Сначала были выделены цветом разные ортологичные группы. К ортологам относятся, например: CLPX_MOOTA и CLPX_CLOBH, HSLU_ENTFA и HSLU_LACAC, A5I7Q0_CLOBH и Q2RM95_MOOTA. К паралогам относятся, например: A5I501_CLOBH и A5HYU4_CLOBH, Q2RLP6_MOOTA и Q2RJJ8_MOOTA, Q5FHW6_LACAC и Q5FMA3_LACAC.

Затем ортологичные группы были "схлопнуты" и подписаны. Реконструкция филогении по этим белкам отличается от филогенетического дерева бактерий.