Практикум 11. BLAST.
Для выполнения заданий был взят белок, с которым я работала ранее, - аргининосукцинат-синтетаза археи Picrophilus torridus DSM 9790. Идентификаторы данного белка в базах данных:
| База данных | Идентификатор |
| RefSeq | WP_011177331.1 |
| UniProt | Q6L1N7 |
Задание 1. Поиск сходных последовательностей в базах данных Refseq_protein и Swiss-prot.
Ссылка на search strategy в БД RefSeq
Ссылка на search strategy в БД Swiss-prot
Поиск по БД RefSeq дал 8946 находок, по БД Swiss-prot - 569 находок. С результатами поиска по RefSeq было сложно работать из-за большого объема находок, поэтому я выполняла последующие задания с находками по Swiss-prot.
-
(Поиск по Swiss-prot) Количество находок последовательностей из геномов различных таксономических групп: эубактерий - 514, эукариот - 20, архей - 35.
-
Характеристики трех находок - первой, последней и из середины списка:
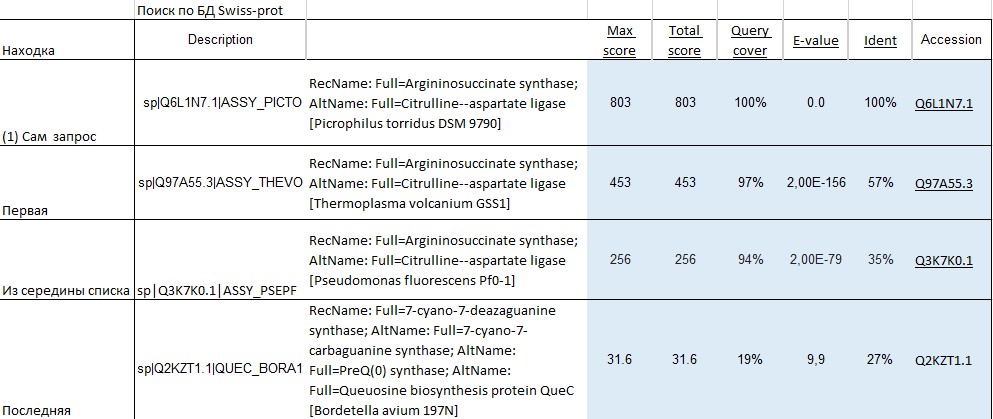
-
То же для RefSeq:
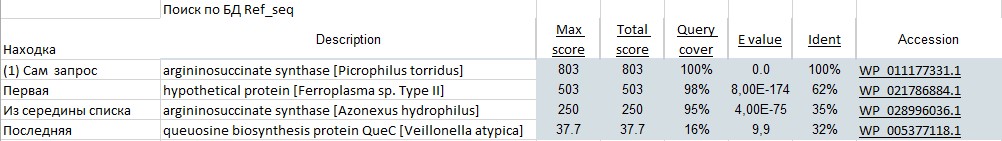
-
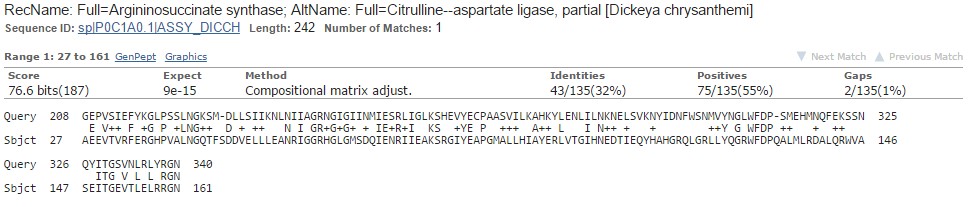
В качестве условного критерия гомологичности находки данной последовательности использовались следующие условия: E-value < 1e-3 и не менее 70% запроса вошло в полученное выравнивание (Query cover). Исходя из этого, в поиске по Swiss-prot гомологами можно считать 504 находки. (Я отсортировала находки по убыванию Query cover и отобрала удовлетворяющие критерию; при этом E-value даже у последних оставлось весьма маленьким (порядка е-20). При изменении в Formatting options максимально возможного E-value на 0.001 в находках остается только одна, чей Query cover меньше 70%. Это аргининосукцинат-синтаза бактерии Erwinia chrysanthemi. Совпадения с искомым белком частичные, но в сходном фрагменте почти нет гэпов (рис.1) и E-value достаточно мал (9e-15). Поэтому, думаю, ее можно считать гомологом искомого белка.
Рис.1 Выравнивание последовательности искомого белка - Q6L1N7 (аргининосукцинат-синтетаза Picrophilus torridus DSM 9790) и P0C1A0.1 (аргининосукцинат-синтазы Dickeya chrysanthemi)
Задание 2. Поиск сходных последовательностей среди белков из определенной таксономической группы.
Был произведен поиск среди архей в БД Swiss-prot. Было получено 56 находок. Это больше, чем число находок архейных белков из поиска по всем таксонам - 35 шт. (см. п.1). Но примерно у половины E-value > 0.001, то есть такие находки не могут считаться гомологами.
Ссылка на search strategy в БД Swiss-prot
Для одной из находок, которая также находилась в п.1, нужно было сравнить, одинаковое ли получилось выравнивание, совпадает ли score, не изменилось ли E-value.
Я искала две из найденных последовательностей по их идентификаторам в находках из пункта 1 (поиск по всем таксонам).
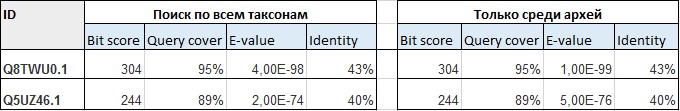
Score совпадает, E-value изменилось, стало меньше. Score совпадает, потому что не изменилась матрица и последовательности. E-value изменилось (уменьшилось), потому что изменилось количество результатов (уменьшилось), и, соответственно, размер банка. (Чем больше банк, тем больше E-value).
Задание 3. Карта локального сходства для одной из находок.
Было выполнено выравнивание двух последовательностей: функция Align two sequences на странице, где запускается поиск по базе данных.
Была выбрана последовательность аргининосукцинат-синтазы из Klebsiella pneumoniae subsp. pneumoniae MGH 78578 (ID A6TEJ0.1).
Полученное выравнивание:
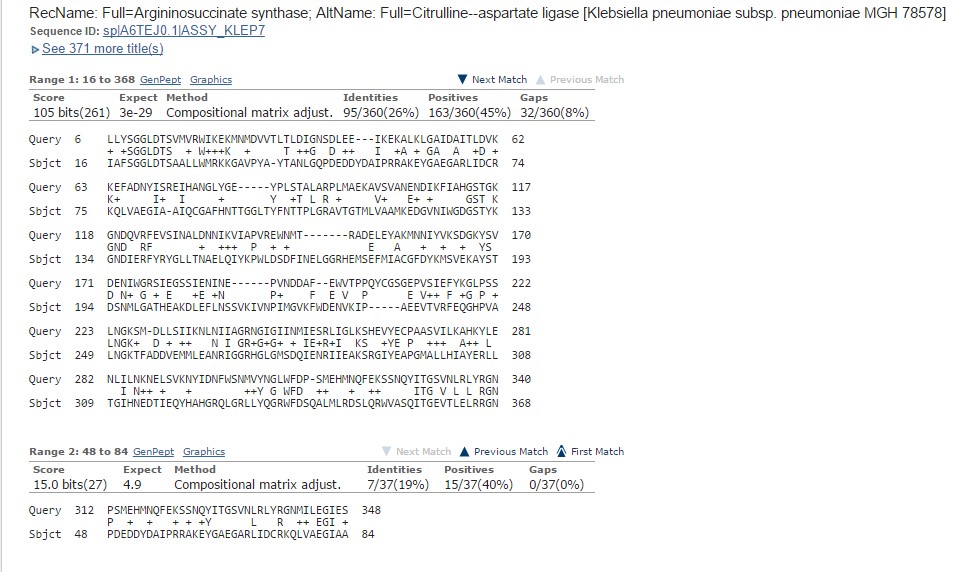
Карта локального сходства:
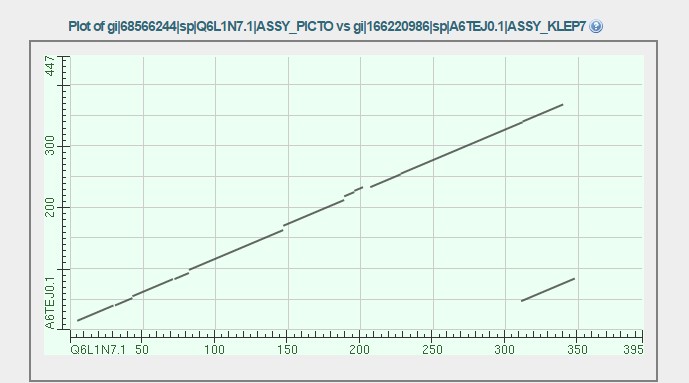
Сплошные участки графика соответствуют сходным фрагментам выравнивания без гэпов. Из наиболее длинных консервативных участков присутствует SGGLDTS (9-15 в query), который встречается в большей части находок. Вероятно, это функционально важный участок, возможно, принадлежащий активному центру.
Задание 4. Использование BLAST для поиска в своей базе данных.
Требовалось создать базу данных из случайных последовательностей и испрользовать BLAST для поиска в ней своего белка. В качестве базы данных было использовано множественное выравнивание 10 из практикума 8.
Выдача программа для лучшей находки:

Процент идентичных остатков (для найденного похожего фрагмента п-ти) = 27%
Процент сходных остатков = 55%
Bit score = 20.0
E-value = 0.75
Score лучшей находки на порядок меньше, чем при поиске в больших базах данных в предыдущих заданиях. E-value велик, несмотря на малый размер БД. Несомненно, эти данные свидетельствуют об отсутствии гомологии между последовательностями, что неудивительно. Query cover = 33/396 = 8.3%