Для выполнения заданий был взят белок, с которым я работала ранее, - аргининосукцинат-синтетаза археи Picrophilus torridus DSM 9790. Идентификаторы данного белка в базах данных:
| База данных | Идентификатор |
| RefSeq | WP_011177331.1 |
| UniProt | Q6L1N7 |
Файл с результатами работы в виде проекта программы Jalview доступен по ссылке
Задание 1. Составление семейства гомологов данного белка с использованием PSI-BLAST.
Ссылка на search strategy с порогом E-value по умолчанию (E-value = 10)
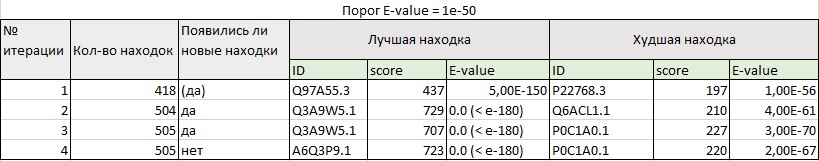
Ссылка на search strategy с порогом E-value 1e-50
Поиск осуществлялся по БД Swiss-prot.
Рис.1 Результаты поиска со значением порога E-value по умолчанию (E-value = 10)
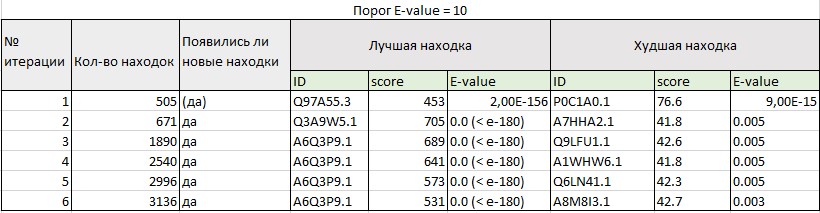
При поиске со значением порога E-value по умолчанию (E-value = 10, рис.1) не стабилизировались даже после 7 итераций, потому что в находки попали белки из соседних семейств. Это произошло уже после второй итерации: в списке появились tRNA-specific 2-thiouridylase и 7-cyano-7-deazaguanine synthase. У этих белков есть общий домен с аргининосукцинат синтазами (это было видно на множественном выравнивании, которое я построила с помощью muscle), но они, вероятно, не гомологичны по всей длине. Лучшую находку во второй итерации (рис. 2) можно, вероятно, считать случайной или, по крайней мере, гомологичной, но принадлежащей к семейству белков, относительно далеко отстоящему от аргининосукцинат-синтаз. В пользу этого говорит большая разница в E-value ее и худшей находки первой итерации, а также маленький query cover.
Рис.1 Результаты поиска со значением порога E-value по умолчанию (E-value = 10)

Чтобы в результаты поиска попадали только белки из нужного семейства – аргининосукцинат-синтазы, я изменила в настройках поиска порог E-value на 1e-50, что больше E-value худшей находки аргининосукцинат-синтазы при пороге E-value = 10. Результаты стабилизировались после 4-й итерации. При этом итоговый список был полностью идентичен находкам в первой итерации поиска с параметрами по умолчанию (порог E-value = 10). Получилось 505 находок. В дальнейшем я работала с ними. Результаты поиска представлены на рис.3.
Рис.3 Результаты поиска со значением порога E-value по умолчанию (E-value = 10)
Задание 3. Построение множественное выравнивание отобранных последовательностей при помощи программы muscle.
Использованная команда: muscle -in assy.fasta -out align_all_muscle.fasta
Результаты представлены в приложенном проекте JalView, окно "assy_all_muscle".
Задания 4 и 5. Построение множественного выравнивания типичных представителей данного семейства (seed).
Для отбора 10-20 последовательностей, являющихся достоверными гомологами и отражающих разнообразие внутри семейства, была использована программа JalView (JalView=> Edit => Remove redundancy). Был выбран такой процент идентичности, после применения функции по которому осталось 15 последовательностей, = 70% (все находки в списке достоверны, E-value < 1e-60).
Оставшиеся после применения функции последовательности были скопированы в text-box и далее в файл в формате fasta с последующим удалением гэпов.
После формирования seed для него было постоено множественное выравнивание с пом-ю mucsle и mafft.
Выравнивание seed с пом-ю mucsle.
Использованная команда: muscle -in seed.fasta -out align_seed__muscle.fasta
Результаты представлены в приложенном проекте в окне "seed_muscle"
Выравнивание seed с пом-ю mucsle.
Использованная команда: mafft seed.fasta > align_seed_mafft.fasta
Результаты представлены в приложенном проекте в окне "seed_mafft"
Задание 6. Сравнение выравниваний seed, полученных c помощью mucsle и mafft.
Для выполнения этого задания я использовала программу muscle. (Команда: muscle -profile -in1 muscle.fasta -in2 mafft.fasta -out both.fasta
Результат представлен в приложенном файле в окне "muscle+mafft_align". Участки совпадения, т.е. те, на которых остатки в колонках первого выравнивания точно те же, что в колонках второго, отмечены + в строке разметки match_sites.
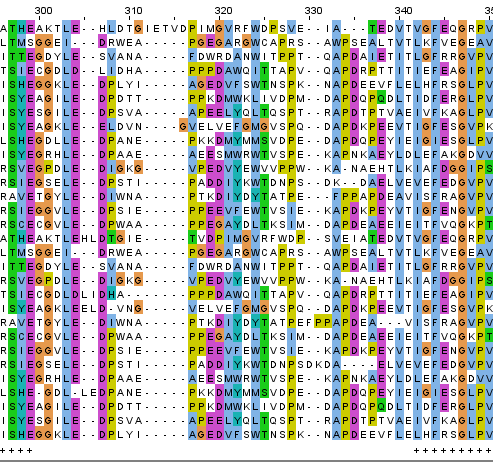
Можно сказать, что выравнивания, построенные двумя программами, почти не отличаются в относительно консервативных местах со множеством консервативных колонок, где "правильное" выравнивание может быть выбрано практически однозначно (рис.4). В более спорных местах встречаются расхождения, обычно касающиеся выравнивания одной-двух последовательностей (рис.5).
Рис.4 Фрагмент множественного выравнивания двух выравниваний, построенное с пом-ю muscle.

Рис 5. Наиболее длинный фрагмент без совпадения выравниваний из задания 6.